ncbi突变命名规则
蛋白名称转gene name

蛋白名称转基因名称的相关问题,在生物信息学研究中扮演着重要角色。
蛋白名称是指给定的一个生物化学蛋白质的命名,而基因名称是指编码该蛋白质的基因的标识符。
在研究中,我们常常需要将蛋白名称转换为基因名称,以便更好地分析、注释和解释基因和蛋白质的功能。
由于蛋白质命名的复杂性和多样性,蛋白名称转换为基因名称并不总是一个简单的任务。
此过程通常需要结合多种资源和方法来完成。
一种常见的方法是使用数据库和工具来进行蛋白名称到基因名称的转换。
一些常用的数据库如UniProt、NCBI和Ensembl等,它们存储了大量的蛋白质序列、基因信息和标识符。
这些数据库通常提供搜索和映射工具,可以根据蛋白名称查询对应的基因名称。
以UniProt为例,它是一个广泛使用的蛋白质数据库,提供了蛋白质序列、标识符、功能注释等信息。
它有一个基于关键词的搜索功能,用户可以输入蛋白名称或关键词进行查询。
查询结果通常包含与蛋白质名称匹配的蛋白质条目及其相关信息,例如基因名称、序列、功能注释等。
通过查看蛋白质条目中的基因名称,就可以将蛋白名称转换为基因名称。
另外,生物信息学研究中常用的一种方法是使用基因和蛋白质关联的规则和模式。
通过分析蛋白质名称的构成规律和常见模式,可以根据一定的规则将蛋白名称转换为基因名称。
这种方法通常需要根据具体问题和数据集进行调整和优化,因为蛋白质命名的规则和模式在不同物种、数据库和文献中可能有所不同。
此外,还有一些基于机器学习和自然语言处理的方法,可以根据大数据集和语义关联来进行蛋白名称到基因名称的转换。
这些方法利用训练好的模型和算法,可以自动识别蛋白质名称中的关键词、规则和语义,从而进行准确的转换。
这些方法通常需要大量的训练数据和计算资源,但具有较高的准确性和效率。
总之,蛋白名称转换为基因名称是生物信息学研究中的重要问题之一。
通过使用数据库、规则和模式、机器学习等方法,我们可以将蛋白名称转换为基因名称,并进一步分析和解释基因和蛋白质的功能。
定点突变小鼠命名方法

定点突变小鼠命名方法展开全文随着基因编辑技术的发展,基因工程小鼠的构建变得日益简单,它成为了研究人类基因功能的最好的模式动物之一。
虽然基因工程小鼠类别不多,但是其命名冗长复杂,对于这方面研究的科研工作者,还是需要学习一下。
我们继续参考International Committee on Standardized Genetic Nomenclature for mice中对大小鼠基因的描述部分。
首先,我们需要明确基因的写法要求1. 一般在文章书写小鼠基因符号是斜体(Rbp1),首字母大写;当是人源的基因时,字母全部大写,斜体;如果是蛋白质则是全部大写,不用斜体。
2. 一般基因用2-4个字母缩写表示,并尽可能地表达其功能和特点;3. 字体为新罗马字体和阿拉伯数字组合。
上一期小编带着大家学习了常见的小鼠分类及命名,基因工程小鼠即:野生型小鼠品系和基因符号的组合。
基因工程的小鼠主要分为定点突变(T argetedMutations)和转基因(Transgenes)小鼠。
C57BL/6,129,FVB是最常使用的基因工程小鼠。
本次小编先带大家了解定点突变(TargetedMutations)小鼠的规则要求。
定点突变(Targeted Mutation)主要分为ES细胞打靶、核酸内切酶介导的突变、基因捕获突变及增强子捕获等。
1ES细胞打靶利用同源重组技术对ES细胞特定位点的基因进行改造,通过显微注射或者胚胎融合的方法将遗传修饰的ES细胞引入受体胚胎内,是一种建立在ES细胞与DNA同源重组等技术基础上的分子生物学技术。
包括Knock-out,Knock-in及条件敲除等。
基因符号由四部分组成:①突变的目的基因;②tm标志代表targeted mutation;③起源于实验室的序列号;④实验室注册代号。
前面可以直接加上小鼠品系,具体参考上期常见小鼠品系命名。
1.1 常见举例:129X1-Cftrtm1Unc :Unc实验室在129X1品系小鼠上靶向Cftr 基因的进行的首次突变。
NCBI注释说明

GeneBank序列注释说明GeneBank格式的序列中,有一些注释很难弄明白它的意思,通过搜索,终于找到了其原始的解释说明。
现以NM_005715为一个简单的例子(##后面为说明)。
LOCUS NM_005715 4396 bp mRNA linear PRI 27-JUN-2012 ##说明accession num、序列长度和最后更新日期DEFINITION Homo sapiens uronyl-2-sulfotransferase (UST), mRNA. ##序列所在基因功能描述ACCESSION NM_005715 ##accession numVERSION NM_005715.2 GI:194578911##accession版本号和所在基因GI号KEYWORDS .SOURCE Homo sapiens (human) ##序列来源,即序列所在物种ORGANISM Homo sapiens ##序列物种生物学分类Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi;Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini;Catarrhini; Hominidae; Homo.REFERENCE 1 (bases 1 to 4396) ##参考文献序号AUTHORS Uher,R., Perroud,N., Ng,M.Y., Hauser,J., Henigsberg,N., Maier,W., ##参考文献作者Mors,O., Placentino,A., Rietschel,M., Souery,D., Zagar,T.,Czerski,P.M., Jerman,B., Larsen,E.R., Schulze,T.G., Zobel,A.,Cohen-Woods,S., Pirlo,K., Butler,A.W., Muglia,P., Barnes,M.R.,Lathrop,M., Farmer,A., Breen,G., Aitchison,K.J., Craig,I.,Lewis,C.M. and McGuffin,P.TITLE Genome-wide pharmacogenetics of antidepressant response in the ##参考文献标题GENDEP projectJOURNAL Am J Psychiatry 167 (5), 555-564 (2010) ##参考文献发表的杂志PUBMED 20360315 ##在pubmed数据库中序号REMARK GeneRIF: Clinical trial and genome-wide association study ofgene-disease association, gene-environment interaction, andpharmacogenomic / toxicogenomic. (HuGE Navigator)REFERENCE 2 (bases 1 to 4396)AUTHORS Trynka,G., Zhernakova,A., Romanos,J., Franke,L., Hunt,K.A.,Turner,G., Bruinenberg,M., Heap,G.A., Platteel,M., Ryan,A.W., deKovel,C., Holmes,G.K., Howdle,P.D., Walters,J.R., Sanders,D.S.,Mulder,C.J., Mearin,M.L., Verbeek,W.H., Trimble,V., Stevens,F.M.,Kelleher,D., Barisani,D., Bardella,M.T., McManus,R., van Heel,D.A.and Wijmenga,C.TITLE Coeliac disease-associated risk variants in TNFAIP3 and RELimplicate altered NF-kappaB signallingJOURNAL Gut 58 (8), 1078-1083 (2009)PUBMED 19240061REMARK GeneRIF: Observational study of gene-disease association. (HuGENavigator)REFERENCE 3 (bases 1 to 4396)AUTHORS Xu,D., Song,D., Pedersen,L.C. and Liu,J.chondroitin sulfate 2-O-sulfotransferaseJOURNAL J. Biol. Chem. 282 (11), 8356-8367 (2007)PUBMED 17227754REMARK GeneRIF: analysis of differences and similarities various residuesplay in the biological roles of the HS-2OST and CS-2OST enzymesREFERENCE 4 (bases 1 to 4396)AUTHORS Ohtake,S., Kimata,K. and Habuchi,O.TITLE Recognition of sulfation pattern of chondroitin sulfate by uronosyl2-O-sulfotransferaseJOURNAL J. Biol. Chem. 280 (47), 39115-39123 (2005)PUBMED 16192264REMARK GeneRIF: 2OST transfers sulfate preferentially to the GlcA residuelocated in a unique sequence, -GalNAc(4SO(4))-GlcA-GalNAc(6SO(4))-.REFERENCE 5 (bases 1 to 4396)AUTHORS Mungall,A.J., Palmer,S.A., Sims,S.K., Edwards,C.A., Ashurst,J.L.,Wilming,L., Jones,M.C., Horton,R., Hunt,S.E., Scott,C.E.,…………TITLE The DNA sequence and analysis of human chromosome 6JOURNAL Nature 425 (6960), 805-811 (2003)PUBMED 14574404REFERENCE 6 (bases 1 to 4396)AUTHORS Kobayashi,M., Sugumaran,G., Liu,J., Shworak,N.W., Silbert,J.E. andRosenberg,R.D.TITLE Molecular cloning and characterization of a human uronyl2-sulfotransferase that sulfates iduronyl and glucuronyl residuesin dermatan/chondroitin sulfateJOURNAL J. Biol. Chem. 274 (15), 10474-10480 (1999)PUBMED 10187838COMMENT V ALIDA TED REFSEQ: This record has undergone validation orpreliminary review. The reference sequence was derived fromAI570697.1, DB496757.1, BC093668.1, AB020316.1 and CA842761.1.On Jul 29, 2008 this sequence version replaced gi:5032218.Summary: Uronyl 2-sulfotransferase transfers sulfate to the2-position of uronyl residues, such as iduronyl residues indermatan sulfate and glucuronyl residues in chondroitin sulfate(Kobayashi et al., 1999 [PubMed 10187838]).[supplied by OMIM, Mar2008].##RefSeq-Attributes-START##Transcript_exon_combination_evidence :: AB020316.1, AK292922.1[ECO:0000332]##RefSeq-Attributes-END##PRIMARY REFSEQ_SPAN PRIMARY_IDENTIFIER PRIMARY_SPAN COMP41-248 DB496757.1 18-225249-1537 BC093668.1 1-12891538-4015 AB020316.1 1345-38224016-4396 CA842761.1 1-381 cFEA TURES Location/Qualifierssource 1..4396 ##序列范围/organism="Homo sapiens" ##物种/mol_type="mRNA" ##序列类型/db_xref="taxon:9606" ##物种编号/chromosome="6" ##所在染色体/map="6q25.1" ##所在染色体区域gene 1..4396 ##包括在基因中的序列范围/gene="UST" ##基因名称/gene_synonym="2OST" ##基因名称别名/note="uronyl-2-sulfotransferase" ##基因名称说明/db_xref="GeneID:10090" ##在GeneBank中的ID号/db_xref="HGNC:17223" ##在HGNC数据库中的ID号/db_xref="HPRD:10298" ##在HPRD数据库中的ID号/db_xref="MIM:610752" ##在MIM数据库中的ID号##有关种物种的数据库代号说明我会另外说明exon 1..543 ##一个外显子在序列中的区域/gene="UST"/gene_synonym="2OST"/inference="alignment:Splign" ##外显子得到的比对所用工具/number=1STS 249..1540 ##STS(sequence target site),在基因组中唯一存在的序列,用来作序列标记/gene="UST"/gene_synonym="2OST"/db_xref="UniSTS:485655" ##在UniSTS数据库中ID号misc_feature 249..251 ##生物学上有特殊意义,但区别于其它标记的区域/gene="UST"/gene_synonym="2OST"/note="upstream in-frame stop codon"##序列说明CDS 297..1517 ##编码区/gene="UST"/gene_synonym="2OST"/note="dermatan/chondroitin sulfate 2-sulfotransferase" ##编码产物说明/codon_start=1/product="uronyl 2-sulfotransferase" ##编码产物/protein_id="NP_005706.1" ##蛋白ID/db_xref="GI:5032219"/db_xref="CCDS:CCDS5213.1" ##CCDS数据库ID/db_xref="GeneID:10090"/db_xref="HGNC:17223"/db_xref="MIM:610752"/translation="MKKKQQHPGGGADPWPHGAPMGGAPPGLGSWKRRVPLLPFLRFSLRDYGFCMA TLLVFCLGSLLYQLSGGPPRFLLDLRQYLGNSTYLDDHGPPPSKVLPFPSQVVYNRVGKCGSRTVVLLLRILSEKHGFNLVTSDIHNKTRLTKNEQMELIKNISTAEQPYLFTRHVHFLNFSRFGGDQPVYINIIRDPVNRFLSNYFFRRFGDWRGEQNHMIRTPSMRQEERYLDINECILENYPECSNPRLFYIIPYFCGQHPRCREPGEW ALERAKLNVNENFLLVGILEELEDVLLLLERFLPHYFKGVLSIYKDPEHRKLGNMTVTVKKTVPSPEA VQILYQRMRYEYEFYHYVKEQFHLLKRKFGLKSHVSKPPLRPHFFIPTPLETEEPIDDEEQDDEKWLEDIYKR" ##翻译的氨基酸序列misc_feature 444..506/gene="UST"/gene_synonym="2OST"/inference="non-experimental evidence, no additionaldetails recorded"/note="propagated from UniProtKB/Swiss-Prot (Q9Y2C2.1);transmembrane region"exon 544..587/gene="UST"/gene_synonym="2OST"/inference="alignment:Splign"/number=2exon 588..743/gene="UST"/gene_synonym="2OST"/inference="alignment:Splign"/number=3exon 744..823/gene="UST"/gene_synonym="2OST"/inference="alignment:Splign"/number=4exon 824..977/gene="UST"/gene_synonym="2OST"/inference="alignment:Splign"/number=5exon 978..1075/gene="UST"/gene_synonym="2OST"/inference="alignment:Splign"/number=6exon 1076..1233/gene="UST"/gene_synonym="2OST"/number=7exon 1234..4391/gene="UST"/gene_synonym="2OST"/inference="alignment:Splign"/number=8STS 4236..4336/gene="UST"/gene_synonym="2OST"/standard_name="D6S1148E"/db_xref="UniSTS:83075"ORIGIN1 ggcgcggcgg ggcgcggggc gtggggacgc tagcgggcgc cggacgggcg cggcgccccg61 tcacgggcag cgccccgaac cggggccgga cacctcggcc gctcgggccg cggcggcggg………….还有很多在此例子中没有出现,现按字母顺序做一个汇总,方便大家查阅。
NCBI上下载基因序列
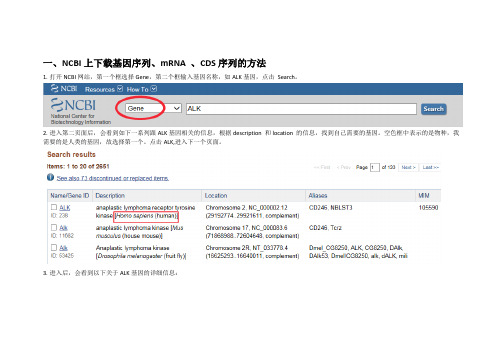
一、NCBI上下载基因序列、mRNA 、CDS序列的方法1.打开NCBI网站,第一个框选择Gene,第二个框输入基因名称,如ALK基因,点击Search。
2.进入第二页面后,会看到如下一系列跟ALK基因相关的信息,根据description 和location 的信息,找到自己需要的基因。
空色框中表示的是物种,我需要的是人类的基因,故选择第一个。
点击ALK,进入下一个页面。
3.进入后,会看到以下关于ALK基因的详细信息:这些信息可以略过,往下查看,点击下图红色框中的See ALK in Genome Data Viewer,进入下个页面。
4.进入后会看到如下页面,:将鼠标箭头放在图中红色框中的绿色线上2--5秒,不用点击。
会看到下面的界面:(有些基因会出现好几个,根据自己的要求选择)第一个红色框中:NP 004295.2 表示蛋白质序列,NP代表蛋白质,004295是编号,后面的.2 代表更新状态,数值越大,版本越新。
NM 004304.4 表示mRNA 序列,NM表示mRNA,004304是编号,后面的.4 代表更新状态,数值越大,版本越新。
第二个红色框中:CCDS33172.1 表示CDS序列,CCDS表示CDS,命名规则同上。
第三个红色框中:NC 000002.12(29,192,774...29,921,611) 表示完整的基因序列,NC表示基因组。
括号中的数字表示这个基因的碱基长度,从29,192,774bp到29,921,611bp。
要下载蛋白序列、mRNA 序列、CDS序列、基因序列,直接点击红色框中的链接即可。
5.这里下载基因序列,点击后出现如下界面:点击右边的Send complete record file format(有好几种格式)Create File。
常用的是FASTA 格式和GenBank 格式。
GenBank 格式保存的信息更全面。
至此就把ALK基因的序列下载了。
基因表达数据库

5.数据分析
“GEO datasets”提供了四种数据分析工具,分别是 “find genes” “compare 2 sets of samples” “cluster heatmaps” “experiment design and value distribution” 。 “find genes” 通过输入基因名称或符号直接定位到 “profiles” 中的相关基因。 “compare 2 sets of samples”用 以区别存在显著表达差异的两组样本,特别是比较属于不同 实验的样本。 “Cluster heat maps”工具提供了分层和 Kmeans 聚类分析方法,可以选择、 扩增、 下载大量感兴趣的 簇,并制成线图
的信息。一个平台含有多个提交者提交的样本。平台的命名 规则为 “GPL+n(n 代表数字)” 。样本,关于被检查的 mRNA 样本,实验条件和实验产生的基因表达测量数据信息。 一个样本必须涉及一个平台,可能会包括在许多系列之中。 样本的命名规则为: “GSM+n” 。
系列,样本收集,样本是如何相关的,如何排序的,分 析是如何进行的,和聚类数据是如何获得的信息。 系列含有 数据的摘要信息。 系列的命名规则为 “GSE+n” 。GEO 的原 始数据被放置在平台,样本和系列这三个数据库中;根据原 始数据观测角度的不同,又将这些数据整理并分置两个不同 的数据库中:数据集(datasets)和表达图谱(profile);数 据集以 “实验” 的角度存储了所有的元数据,表达图谱从 “基因” 的角度存储了单个基因表达的数据资料。
同时提供向NCBI 的其他数据库的链 接,如 PubMed, Epigenomics、 SRA 和 GEO Profiles 等。GEO 表达图谱 (profiles)的搜索结果以 “基因”角度列示,用图片的形式 展现一个基因对所有样本的表达水平,搜索结果中的实验条 件方便我们观察一个基因在不同条件下表达水平的差异。搜 索结果可利用 “my NCBI→collection” 进行在线保存。
如何在论文中正确使用基因

如何正确使用基因/蛋白质的名称格式(大写与斜体的使用)(美国LetPub编辑 : SCI论文写作系列6)1 一般规则·在你的文章中使用大家都认可的基因/蛋白质的名称和符号(见下文)·有时认可的基因/蛋白质的名称或符号已经不再有效。
这种情况下,第一次提到的基因/蛋白质,需要先列出经批准的名称,然后再添加括号说明(以前被称为XXX)。
例如,“我们使用抗POU5F1蛋白的抗体(以前称为OCT-4)...”。
注意使用正确的符号,而不是以往的名称。
2 不同物种有不同的规则1) 小鼠/ 大鼠/ 鸡(1)一般命名规则(适用于小鼠,大鼠和鸡):·基因的全名不用斜体,也不用希腊字母例如:insulin-like growth factor 1(胰岛素样生长因子1)·基因符号不用希腊字母和连字符,使用斜体,第一个字母大写,其余小写例如:Igf1 (斜体)(胰岛素样生长因子1)·蛋白质的名称和基因符号相同,但不用斜体,并全部大写例如:IGF1·mRNA和cDNA的基因符号和规定的格式例如:"... levels of Igf1 (italicized) mRNA increased when..."·首次提到的突变等位基因应该详细列出例如:Igf1tm1Arge/Igf1tm1Arge (italicized) is one of several knockout alleles of Igf1 (italicized)·所有字母和数字用斜体,等位基因的符号(tm1Arge)要用上标·经前面详细描述之后,敲除的纯合子可表示为Igf1-/- (所有字母均用斜体,并且- / -用上标);杂合子为Igf1 + / - 等。
(2)对于这些命名规定的详细信息,请参阅:·(小鼠,大鼠,鸡) /·(专门针对大鼠) /·MGI命名:/mgihome/nomen/index.shtml·基因的MGI快速指南:/mgihome/nomen/short_gene.shtml2) 人类/非人类灵长类动物/家畜/ 除了小鼠、大鼠、鱼、昆虫、苍蝇外默认的其他物种·完整的基因名称不用斜体和希腊字母例如:insulin-like growth factor 1(胰岛素样生长因子1)·基因符号不使用希腊字母和连字符,基因符号用斜体,所有的字母用大写例如:IGF1(斜体)·蛋白质的名称与基因符号相同,但不用斜体,并全部大写例如:IGF1·mRNA和cDNA的基因符号和规定的格式例如:"... levels of IGF1 (italicized) mRNA increased when..."更多关于此类物种基因(突变的等位基因)命名规则和符号的网站:/3) 鱼(适用于所有的鱼)·完整的基因名称用斜体表示,全部用小写字母,不要用希腊字母例如:cyclops(斜体)·基因符号用斜体,全部小写例如:cyc(斜体)·蛋白质命名与基因符号相同,但仅首字母大写,也不用斜体例如:Cyc更多关于此类物种基因(突变的等位基因)命名规则和符号的网站:/cgi-bin/webdriver?MIval=aa-ZDB_home.apg4) 其他有用的网站:·ExPASy(特殊蛋白质分析系统):是瑞士生物信息学研究所(SIB)提供的蛋白质组学服务系统,可对蛋白质序列和结构以及二维PAGE进行分析:( / )·OMIM -孟德尔人类遗传学数据库:(/entrez/query.fcgi?db=OMIM )·NCBI - Entrez Gene:提供了一个按序列和/或在NCBI's Map Viewer中查询基因的统一环境。
如何正确使用基因蛋白质的名称格式(大写与斜体的使用)

如何正确使用基因/蛋白质的名称格式(大写与斜体的使用)1 一般规则•在你的文章中使用大家都认可的基因/蛋白质的名称和符号(见下文)•有时认可的基因/蛋白质的名称或符号已经不再有效。
这种情况下,第一次提到的基因/蛋白质,需要先列出经批准的名称,然后再添加括号说明(以前被称为XXX)。
例如,“我们使用抗POU5F1蛋白的抗体(以前称为OCT-4)...”。
注意使用正确的符号,而不是以往的名称。
2 不同物种有不同的规则1) 小鼠 / 大鼠 / 鸡(1)一般命名规则(适用于小鼠,大鼠和鸡):•基因的全名不用斜体,也不用希腊字母例如:insulin-like growth factor 1(胰岛素样生长因子1)•基因符号不用希腊字母和连字符,使用斜体,第一个字母大写,其余小写例如:Igf1 (斜体)(胰岛素样生长因子1)•蛋白质的名称和基因符号相同,但不用斜体,并全部大写例如:IGF1•mRNA和cDNA的基因符号和规定的格式例如: "... levels of Igf1 (italicized) mRNA increased when..."•首次提到的突变等位基因应该详细列出例如:Igf1tm1Arge/Igf1tm1Arge (italicized) is one of several knockout alleles of Igf1 (italicized) •所有字母和数字用斜体,等位基因的符号(tm1Arge)要用上标•经前面详细描述之后,敲除的纯合子可表示为Igf1-/- (所有字母均用斜体,并且- / -用上标);杂合子为Igf1 + / - 等。
(2)对于这些命名规定的详细信息,请参阅:•(小鼠,大鼠,鸡) /•(专门针对大鼠) /•MGI命名:/mgihome/nomen/index.shtml•基因的MGI快速指南:/mgihome/nomen/short_gene.shtml2) 人类/非人类灵长类动物/家畜/ 除了小鼠、大鼠、鱼、昆虫、苍蝇外默认的其他物种•完整的基因名称不用斜体和希腊字母例如:insulin-like growth factor 1(胰岛素样生长因子1)•基因符号不使用希腊字母和连字符,基因符号用斜体,所有的字母用大写例如:IGF1(斜体)•蛋白质的名称与基因符号相同,但不用斜体,并全部大写例如:IGF1•mRNA和cDNA的基因符号和规定的格式例如:"... levels of IGF1 (italicized) mRNA increased when..."更多关于此类物种基因(突变的等位基因)命名规则和符号的网站:3) 鱼(适用于所有的鱼)•完整的基因名称用斜体表示,全部用小写字母,不要用希腊字母例如:cyclops(斜体)•基因符号用斜体,全部小写例如:cyc(斜体)•蛋白质命名与基因符号相同,但仅首字母大写,也不用斜体例如:Cyc更多关于此类物种基因(突变的等位基因)命名规则和符号的网站:/cgi-bin/webdriver?MIval=aa-ZDB_home.apg4) 其他有用的网站:•ExPASy(特殊蛋白质分析系统):是瑞士生物信息学研究所(SIB)提供的蛋白质组学服务系统,可对蛋白质序列和结构以及二维PAGE进行分析:( /) •OMIM -孟德尔人类遗传学数据库:(/entrez/query.fcgi?db=OMIM)•NCBI – Entrez Gene:提供了一个按序列和/或在NCBI's Map Viewer中查询基因的统一环境。
ANNOVAR注释基因突变位点

基因突变注释相关数据库简介
5.5 PROVEAN : 突变对蛋白生物功能影响预测。分值越小,危害越大。
/
5.6 CADD 对SNV和InDel等变异危害性进行打分的工具,它整合多种信息来注释变异位点
的功能;能够预测编码区变异(包括同义突变和非同义突变)的功能影响以及非 编码区变异的功能影响。得分结果中,10 表示score 排名在前10%,20 表示前 1%,30 表示前 0.1%。
CONTENTS
1 基因突变类型与注释方法 2 基因突变注释相关数据库简介
01
O
N
E
基因突变类型与注释方法
基因突变的类型
碱基替换
点突变
基因突变
静态突变
片段突变
移码突变 缺失 重复 重排
同义突变 无义突变 错义突变 终止密码突变
动态突变(三联密码子的重复次数的累加效应)
基因突变注释方法
基因突变基础注释:
于ANNOVAR软件二次开发的网页版,可以对变异位点进行功能注释 (包括但不限于:千人基因组、ESP6500、ExAC、dbsnp、clinvar、 COSMIC、dbNSFP[SIFT、Polyphen2、MutationTaster、 MutationAssessor、GERP++]),从而可以更加直观的展示基因组的 某种突变可能导致的影响,更进一步的,方便对全基因组、全外显子或 目标区域捕获测序的结果进行进一步的筛选。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
ncbi突变命名规则
(最新版)
目录
1.NCBI 的概述
2.突变命名规则的背景和重要性
3.NCBI 的突变命名规则的具体内容
4.突变命名规则的实际应用
5.总结
正文
【1.NCBI 的概述】
CBI(National Center for Biotechnology Information)是美国国立生物技术信息中心,负责收集、存储和传播生物学和医学研究所需的各种信息资源。
NCBI 成立于 1988 年,是美国国立卫生研究院(NIH)的一部分,其数据库和工具广泛应用于生物学、医学和药物研究等领域。
【2.突变命名规则的背景和重要性】
在生物学研究中,基因突变是引发遗传病和生物体表型差异的重要原因。
对基因突变进行准确、规范的命名,有助于科研人员、临床医生和药物研发人员更好地理解、诊断和治疗相关疾病。
因此,制定一套科学的突变命名规则具有重要意义。
【3.NCBI 的突变命名规则的具体内容】
CBI 的突变命名规则主要遵循以下原则:
(1)突变类型和位置:突变类型包括点突变、插入、缺失等;突变位置指明突变发生在基因的哪个位置,通常用密码子或氨基酸位置表示。
(2)参考序列:突变命名时需要指明参考序列,一般采用基因组参
考序列(GRCh)或蛋白质参考序列(Swiss-Prot)。
(3)等位基因和等位突变:对于某个基因的多个等位基因,需要分别命名。
对于同一等位基因的不同突变形式,可采用不同的命名方式表示。
(4)命名前缀和后缀:突变命名时需加上适当的前缀和后缀,如“c.”表示编码区突变,“g.”表示基因组突变,“p.”表示蛋白质突变等。
【4.突变命名规则的实际应用】
CBI 的突变命名规则在生物信息学领域具有广泛的应用,如在基因变异数据库(dbSNP)、突变数据库(ClinVar)和非编码变异数据库(ncvar)等数据库中,都可以看到按照该规则命名的突变。
这有助于科研人员、临床医生和药物研发人员更高效地获取和利用这些信息。
【5.总结】
总之,NCBI 的突变命名规则为生物学研究提供了一套科学、规范的命名体系,有助于提高基因突变信息的可读性和可利用性。
