Pathways enrichment for Gene
基于网络药理学探讨澳洲茄碱抗肿瘤机制

图 1 澳茄碱洲-肿瘤靶点基因匹配情况
恶性肿瘤严重危害人类健康,据统计全球每年 新发恶性肿瘤 1808 万例,死亡 956 万例,而中国分 别占新发的 23.7%和死亡的 30%[1]。中医辨治肿瘤经 验丰富,是我国肿瘤治疗重要组成部分。中药通过多 靶点、多途径、多效应抗肿瘤,在肿瘤防治中应用广 泛 。澳 洲 茄 碱(s olas onine ,SS)是 传 统 抗 癌 中 药 龙 葵 (Solanum nigrum L.)的主要抗癌成分,研究表明,澳
基金项目:浙江省医药卫生科技计划项目(No.2017KY519) 作者单位:浙江中医药大学附属第三医院肿瘤科(杭州 310005) 通信作者:高聚伟,Tel:15397158405;E-mail:gbyisheng@
洲茄碱通过抑制细胞增殖、诱导细胞凋亡、阻断细胞 周期等机制,在膀胱癌、肝癌、胃癌等多种癌肿中发 挥抗肿瘤效应[2-5]。澳洲茄碱抗肿瘤作用靶点及机制 目前未完全明确。因此,本研究利用网络药理学方 法,对澳洲茄碱抗肿瘤作用靶点及机制进行研究,为 澳洲茄碱进一步实验研究和临床使用提供依据,现 报道如下。 1 资料与方法 1.1 澳洲茄碱药物靶点筛选 使用 TCMSP 数据库 (https:///tcmsp.php)获得澳洲茄碱 mol2 结
浙江中西医结合杂志 2021 年第 31 卷第 7 期 ZJITCWM(Vol.31 No.7 2021)
611
图 3 PPI 关系网络
鸡胚心脏组织转录组数据鉴定雪域白鸡高原低氧适应性关键基因

畜牧兽医学报 2023,54(10):4154-4163A c t a V e t e r i n a r i a e t Z o o t e c h n i c a S i n i c ad o i :10.11843/j.i s s n .0366-6964.2023.10.014开放科学(资源服务)标识码(O S I D ):鸡胚心脏组织转录组数据鉴定雪域白鸡高原低氧适应性关键基因陈雪娇2,刘会杰1,臧 蕾1,冯 静1,鹏 达1*,张 浩2*(1.西藏自治区农牧科学院畜牧兽医研究所,拉萨850009;2.中国农业大学动物科学技术学院,高原畜禽遗传资源研究中心,北京100193))摘 要:旨在通过分析白来航(W h i t e L e g h o r n ,W L )和雪域白鸡(X u e yu W h i t e c h i c k e n ,X YW )在低氧条件下孵化时胚胎心脏组织的基因表达差异,挖掘鸡胚低氧适应候选基因㊂本研究在高海拔环境孵化雪域白鸡和白来航鸡种蛋,采集孵化第16天胚胎心脏组织,提取R N A ,进行转录组测序(R N A -s e q),筛选差异表达基因(D E G s ),并对其进行荧光定量P C R (q R T -P C R )验证㊁功能富集分析和构建转录因子-靶基因调控网络,鉴定与雪域白鸡低氧适应相关的候选基因㊂在雪域白鸡和白来航鸡的心脏组织之间筛选到253个D E G s ,随机选择5个D E G s 进行q R T -P C R 验证,表达趋势与测序结果一致㊂功能富集分析表明,D E G s 主要涉及心血管发育和心脏功能相关的生物学过程和信号通路㊂将D E G s 和已知的鸡转录因子对比,筛选到F O X P 2和HO X A 2,参与调控血管生成㊁心肌收缩等,对雪域白鸡胚胎低氧适应起关键作用㊂本研究通过鸡胚心脏组织转录组数据分析,鉴定到T E NM 2㊁N O G ㊁S M O C 1㊁C C B E 1等鸡胚高原低氧适应候选基因,为解析雪域白鸡高原低氧适应分子机制提供理论依据㊂关键词:雪域白鸡;白来航鸡;低氧适应;心脏;转录组中图分类号:S 831.2 文献标志码:A 文章编号:0366-6964(2023)10-4154-10收稿日期:2023-05-18基金项目:西藏自然科学基金项目(X Z 202101Z R 0061G );国家现代农业产业技术体系(C A R S -40)作者简介:陈雪娇(1998-),女,河北保定人,硕士生,主要从事家禽遗传育种研究,E -m a i l :c h e n x u e ji a o 0805@126.c o m *通信作者:鹏 达,主要从事鸡遗传育种研究,E -m a i l :2845545151@q q.c o m ;张 浩,主要从事动物遗传资源与功能基因组学研究,E -m a i l :z h a n gh a o 827@163.c o m T r a n s c r i p t o m e D a t a f r o m C h i c k e n E m b r y o H e a r t T i s s u e I d e n t i f i e d K e y Ge n e sf o r A l t i t u d e H y p o x i a A d a p t a t i o n i n X u e yu W h i t e C h i c k e n s C H E N X u e j i a o 2,L I U H u i j i e 1,Z A N G L e i 1,F E N G J i n g 1,P E N G D a 1*,Z H A N G H a o 2*(1.I n s t i t u t e o f A n i m a l S c i e n c e ,T i b e t A c a d e m y o f A g r i c u l t u r a l a n d A n i m a l H u s b a n d r y S c i e n c e s ,L h a s a 850009,C h i n a ;2.P l a t e a u A n i m a l a n d P o u l t r y G e n e t i c R e s o u r c e s R e s e a r c h C e n t e r ,C o l l e g e o fA n i m a l S c i e n c e a n d T e c h n o l o g y ,C h i n a A g r i c u l t u r a l U n i v e r s i t y ,B e i j i n g 100193,C h i n a )A b s t r a c t :T h e a i m o f t h i s s t u d y w a s t o i d e n t i f y c a n d i d a t e g e n e s f o r h y p o x i a a d a pt a t i o n i n c h i c k e n e m b r y o s b y a n a l y z i n g t h e g e n e e x p r e s s i o n c h a n g e s i n e m b r y o n i c h e a r t t i s s u e s o f W h i t e L e g h o r n (W L )a n d X u e y u W h i t e (X YW )c h i c k e n s i n c u b a t e d u n d e r h y p o x i c c o n d i t i o n s .T h e e g gs w e r e i n -c u b a t e d a t h i g h a l t i t u d e .T h e h e a r t t i s s u e s w e r e c o l l e c t e d f r o m e m b r y o s a t d a y 16o f i n c u b a t i o n ,a n d t h e R N A s w e r e e x t r a c t e d f o r R N A s e q u e n c i n g (R N A -s e q ).D i f f e r e n t l y e x p r e s s e d g e n e s (D E G s )w e r e s c r e e n e d ,w h i c h w e r e v a l i d a t e d u s i n g f l u o r e s c e n c e q u a n t i t a t i v e P C R (qR T -P C R ).T h e f u n c t i o n a l e n r i c h m e n t a n a l y s i s a n d a t r a n s c r i p t i o n f a c t o r -t a r g e t g e n e r e g u l a t o r y ne t w o r k w a s c o n s t r u c t e d t of u r t h e r s c r e e n c a n d i d a t eg e n e s r e l a t e d t oh y p o xi a a d a pt a t i o n i n X YW.T h e 25310期陈雪娇等:鸡胚心脏组织转录组数据鉴定雪域白鸡高原低氧适应性关键基因D E G s w e r e s c r e e n e d i n h e a r t t i s s u e s b e t w e e n X YW a n d W L,a n d5D E G s w e r e r a n d o m l y s e l e c t e d f o r q R T-P C R v a l i d a t i o n.T h e i r e x p r e s s i o n t r e n d w a s c o n s i s t e n t w i t h t h e s e q u e n c i n g r e s u l t s.E n r i c h m e n t a n a l y s i s s h o w e d t h a t D E G s w e r e m a i n l y e n r i c h e d i n b i o l o g i c a l p r o c e s s e s a n d s i g n a l i n g p a t h w a y s r e l a t e d t o c a r d i o v a s c u l a r d e v e l o p m e n t a n d c a r d i a c f u n c t i o n.C o m p a r i n g D E G s w i t h k n o w n c h i c k e n t r a n s c r i p t i o n f a c t o r s,F O X P2a n d HO X A2w e r e s c r e e n e d,w h i c h r e g u l a t e d a n g i o-g e n e s i s,m y o c a r d i a l c o n t r a c t i o n a n d o t h e r f u n c t i o n s,a n d p l a y e d a k e y r o l e i n h y p o x i a a d a p t a t i o n i n X YW e m b r y o s.T h r o u g h t h e a n a l y s i s o f t r a n s c r i p t o m i c d a t a,T E NM2,N O G,S M O C1, C C B E1a n d o t h e r c a n d i d a t e g e n e s w e r e i d e n t i f i e d f o r p l a t e a u h y p o x i a a d a p t a t i o n i n c h i c k e n e m b r y o s,w h i c h p r o v i d e a t h e o r e t i c a l b a s i s f o r a n a l y z i n g t h e m o l e c u l a r m e c h a n i s m o f p l a t e a u h y p o x i a a d a p t a t i o n i n X YW.K e y w o r d s:X u e y u W h i t e c h i c k e n;W h i t e L e g h o r n c h i c k e n;h y p o x i a a d a p t a t i o n;h e a r t;t r a n s c r i p-t o m e*C o r r e s p o n d i n g a u t h o r s:P E N G D a,E-m a i l:2845545151@q q.c o m;Z H A N G H a o,E-m a i l: z h a n g h a o827@163.c o m雪域白鸡,原名拉萨白鸡,于2020年通过国家畜禽遗传资源委员会新品种审定,并正式命名为 雪域白鸡 ㊂雪域白鸡是由白来航鸡和藏鸡杂交选育而来,兼具藏鸡的高原适应性和白来航的高产蛋性能,属于高海拔地区轻型良种蛋鸡品种[1],为西藏养鸡业带来了较高的经济效益㊂低氧适应研究一直深受人们的关注㊂有关低氧感应过程的研究成果获得了2019年的诺贝尔医学或生理学奖㊂低氧与人类的一些疾病紧密相关,例如心脑血管疾病㊁缺血性损伤㊁高原反应等[2-3],还是肿瘤微环境的重要指标[4]㊂世居青藏高原等高海拔地区的人类和动物,经过多年的自然选择和遗传变异,形成了独特的生理特征,能够耐受高寒㊁低氧㊁低压的环境[5-6]㊂鸟类的生殖方式是卵生,胚胎体外发育,对氧气含量的变化非常敏感[7]㊂已有研究证明,胚胎期是低氧适应的关键时期[8]㊂雪域白鸡经过多年的选育,已经适应了高原低氧的环境,在海拔3780m的环境条件下受精蛋孵化率高达81.33%,而北京油鸡仅为1.00%[9]㊂心脏是鸡胚适应低氧环境的关键器官,低海拔鸡种在受到低氧刺激时,心脏发育异常,心室壁变薄,心室容积增大,不利于泵血,而青藏高原本土鸡种藏鸡的心脏发育正常[10-11]㊂本研究选取低海拔鸡种白来航鸡作为对照,将种蛋在高海拔地区(西藏拉萨)进行孵化,采集孵化第16天的鸡胚心脏组织进行转录组测序,鉴定雪域白鸡适应高原低氧环境的关键基因及其调控途径㊂1材料与方法1.1试验材料白来航和雪域白鸡的种蛋均在西藏拉萨(平均海拔高度3650m)进行孵化,温度37.8ħ,相对湿度55%,每天翻蛋12次㊂选取孵化第16天的白来航鸡胚10个(W L-D16,n=10)和雪域白鸡胚10个(X YW-D16,n=10),采集心脏组织,迅速放置于液氮速冻,然后置于-80ħ冰箱进行保存㊂1.2总R N A提取及质控取适量心脏组织,研磨棒研磨后,使用T r i z o l 法提取鸡胚心脏总R N A,微量分光光度计N a n o-D r o p2000(T h e r m o,美国)检测其O D260n m/O D280n m 值鉴定R N A样品浓度㊂使用A g i e n t2100/L a b C h i pG X检测R N A的完整性㊂1.3文库构建及测序样品检测合格后,进行文库构建,每组测序文库设3个生物学重复㊂用带有O l i g o(d T)的磁珠富集m R N A;加入F r a g m e n t a t i o n B u f f e r将m R N A进行随机打断;以m R N A为模板,合成第一条c D N A链及二链,并进行c D N A纯化;对纯化的双链c D N A进行末端修复㊁加A尾,并连接测序接头;然后用A M P u r e X P b e a d s选择380b p左右的片段;最后通过P C R得到c D N A测序文库㊂使用Q u b i t3.0荧光定量仪库对文库进行初步定量,浓度需达到1n g㊃μL-1以上,随后用Q s e p400高通量分析系统对文库的插入片段进行检测㊂文库质检合格后,使用I l l u m i n a N o v a S e q6000测序平台进行P E150模式测序㊂5514畜 牧 兽 医 学 报54卷1.4 测序数据处理及差异表达基因筛选对原始数据进行质控,去除含有接头和低质量的r e a d s 得到c l e a n r e a d s ㊂使用H I S A T 2软件将c l e a n r e ad s 与参考基因组进行快速精确的比对㊂利用S t r i n g t i e 对m a p pe d r e a d s 进行组装和定量,采用F P KM (f r ag m e n t s p e r k i l o b a s e o f t r a n s c r i pt p e r M i l l i o n f r a g m e n t s m a p pe d )进行标准化[12],斯皮尔曼相关系数r (S pe a r m a n 's c o r r e l a t i o n c o ef f i c i e n t )作为评估样本间基因表达重复性的指标㊂以白来航为对照组,采用D E S e q2进行基因表达量差异分析[13],将F o l d C h a n ge ȡ2且F D R <0.05作为筛选标准㊂1.5 差异表达基因的功能分析对筛选到的D E G s 进行功能富集分析,G e n eO n t o l o g y (G O )在百迈克在线网站(h t t ps :ʊi n t e r n a -t i o n a l .b i o c l o u d .n e t /)完成,K E G G (K y o t o E n c yc l o -pe d i a of G e n e s a n d G e n o m e s )通路分析使用K O -B A S 在线网站(h t t p :ʊb i o i n f o .o r g/k o b a s /)完成㊂使用R 包g g p l o t 2进行G O 和K E G G 富集分析可视化㊂1.6 转录因子筛选和靶基因预测在线网站A n i m a l T F D B (h t t p:ʊb i o i n f o .l i f e .h u s t .e d u .c n /A n i m a l T F D B 4/#/)可下载鸡的转录因子(T F )列表,与筛选的D E G s 进行对比,找到测序数据中差异表达的转录因子㊂在线网站J A S -P A R (h t t p s :ʊj a s p a r .g e n e r e g.n e t /)下载转录因子的基序(m o t i f ),选定D E G s 转录起始位点(t r a n -s c r i p t i o n s t a r t s i t e ,T S S )上游5000b p 为启动子序列,最后在M E M E (h t t p s :ʊm e m e -s u i t e .o r g/m e m e /d o c /m e m e .h t m l)网站对基序和启动子序列进行靶向预测,建立T F -t a r ge t s 网络㊂1.7 实时荧光定量P C R (qR T -P C R )验证选取5个D E G s ,肌动蛋白γ2(a c t i n γ2,A C T G 2)㊁钾钙激活通道亚家族M α1(po t a s s i u m c a l c i u m -a c t i v a t e d c h a n n e l s u b f a m i l y M α1,K C -NMA 1)㊁叉头框P 2(f o r k h e a d b o x P 2,F O X P 2)㊁同源框A 2(h o m e o b o x A 2,H O X A 2)㊁肌动蛋白α2(a c t i n α2,A C T A 2),在N C B I 网站设计引物(表1),引物由生工生物工程(上海)股份有限公司合成㊂选取G A P DH 为内参基因,对白来航鸡和雪域白鸡样本进行q R T -P C R 验证,以检测测序结果及数据分析的准确性㊂采用2-ΔΔC T 法计算基因相对表达量㊂采用G r a ph P a d P r i s m 9.5.1软件作图㊂表1 q R T -P C R 引物T a b l e 1 q R T -P C R p r i m e r s基因G e n e引物序列(5'ң3')P r i m e r s e qu e n c e 扩增长度/b pA m pl i c o n s i z e A C T A 2F :A C T C T G C T G A C T G A A G C A C C145R :T C C C T G T A G T A C G C C C A G A AA C T G 2F :G T G C G A C A T C G A C A T C C G T A A241R :C T C G T C G T A C T C G G G T T T G C K C NM A 1F :A A G C A C T G A T T G C C G A G G A A 244R :C G T T T A G T G C A T T G G C T G G G H O X A 2F :C C C T C G A G G T C C C C T C T T T A 166R :A A G T C A A T C G T G G T G A G C G TF O X P 2F :TG C A G A G G A T G A A G A C T G T C C 173R :G T G G A G T G G T T C T G A A G G T C A2 结 果2.1 转录组数据白来航和雪域白鸡共6个样品的转录组测序数据经过质量控制,共得到38.53G b c l e a n d a t a,每个样品的c l e a n d a t a 均达到了5.97G b 及以上,Q 30碱基百分比在92.85%及以上㊂将c l e a n r e a d s 与鸡参考基因组C R C g7b 进行比对,比对率在92.01%到93.64%之间(表2)㊂在白来航和雪域白鸡胚胎心脏组织中共鉴定20291个阳性表达基因,其中共同表达的基因18258个㊂2.2 差异表达基因及功能分析白来航与雪域白鸡胚胎心肌组织中筛选到253个D E G s ,其中在雪域白鸡中122个基因显著上调,131个基因显著下调(图1)㊂差异表达的253个基因主要富集于心肌组织发育调节㊁心肌细胞增殖㊁651410期陈雪娇等:鸡胚心脏组织转录组数据鉴定雪域白鸡高原低氧适应性关键基因表2 测序样品与参考基因组的序列比对T a b l e 2 S e q u e n c e a l i g n m e n t b e t w e e n t h e s e q u e n c e d s a m pl e s a n d t h e r e f e r e n c e g e n o m e 样本名称S a m pl e I D 碱基总数B a s e s u mG C 含量/%G C c o n t e n tQ 30/%总读数T o t a l R e a d s映射读数M a p pe d R e a d s W L -D 16-1658066736449.5593.594401928840877665(92.86%)W L -D 16-2698055905249.4893.834669339243722541(93.64%)W L -D 16-3613569012249.2693.424105360038086058(92.77%)X YW -D 16-159********49.2493.113995901836764412(92.01%)X YW -D 16-2627240084850.5592.854195505238831281(92.55%)X YW -D 16-3658771667649.0194.554405787440890429(92.81%)W L -D 16-1~3.白来航鸡孵化16天样本;X YW -D 16-1~3.雪域白鸡孵化16天样本;Q 30.原始数据中P h r e d 数值大于30的碱基数量占总碱基数量的百分比(P h r e d .用来预测碱基测序错误率)W L -D 16-1-3.W h i t e L e g h o r n c h i c k e n h a t c h i n g s a m p l e s f o r 16d a y s ;X YW -D 16-1-3.X u e y u W h i t e c h i c k e n h a t c h i n g s a m pl e s f o r 16d a y s ;Q 30.T h e p e r c e n t a ge of t h e n u m b e r o f b a s e s w i t h P h r e d v a l u e sg r e a t e r th a n 30i n t h e t o t a l n u m b e r o f b a s e s i n t h e o r i g i n a l d a t a (P h r e d .U s e d t o p r e d i c t b a s e s e q u e n c i n g er r o r r a t e)灰色是表达差异不显著的基因;红色是表达上调的基因;蓝色是表达下调的基因N o n -d i f f e r e n t i a l l y e x p r e s s e d g e n e s a r e s h o w e d i n g r e y c o l o r ;r e d s h o w u p r e g u l a t e d g e n e s ;b l u e s h o w d o w n r e gu l a t e d g e n e s 图1 差异表达基因的火山图F i g .1 V o l c a n i c m a p o f d i f f e r e n t i a l l y e x pr e s s e d g e n e s 心肌组织生长㊁肌原纤维㊁细胞外基质㊁苹果酸脱氢酶(脱羧)(N A D P+)活性㊁MA P 激酶磷酸酶活性㊁钙释放通道活性和钙离子结合等G O 条目(图2)㊂利用K O B A S 数据库分析D E G s 功能,发现它们(T E NM 2㊁N O G ㊁S M O C 1㊁C C B E 1㊁E N D 2㊁A C T G 2㊁和MMP 2等)主要参与血管平滑肌收缩㊁MA P K 信号通路㊁血管内皮生长因子信号通路和心肌收缩等途径,这些途径可能参与心脏形态发生和功能㊂此外D U S P 5㊁U P B 1㊁L P I N 2㊁P L A 2G 4A和P D G F C 等差异表达基因还参与多种代谢途径,包括淀粉和蔗糖代谢㊁泛酸和辅酶A 的生物合成以及甘油磷脂代谢等(图3),这些能量代谢途径可能为机体生存和心脏起搏提供能量㊂2.3 转录因子筛选和靶基因网络构建经过筛选,差异表达基因中含有2个转录因子,分别是F O X P 2和HO X A 2㊂由于m o t i f 的每个位点碱基分布频率不同,转录因子和靶基因可能存在一个或多个结合位点(表3)㊂通过将m o t i f 与启动子序列比对,构建了转录因子与靶基因网络(图4),值得注意的是,其中一些靶基因(MMP 2㊁A C T A 2㊁A C T G 2和K C NMA 1等)的功能富集在血管内皮生长因子信号通路㊁应对低氧和血管生成等途径,提7514畜 牧 兽 医 学 报54卷图2 差异表达基因的G O 功能注释F i g .2 G O f u n c t i o n a l a n n o t a t i o n o f d i f f e r e n t i a l l y e x pr e s s e d g e n es 图3 差异表达基因的K E G G 通路富集分析F i g .3 K E G G p a t h w a y e n r i c h m e n t a n a l y s i s o f d i f f e r e n t i a l l y e x pr e s s e d g e n e s 示与心脏发育和功能有关(图5)㊂2.4 差异表达基因验证选取5个差异表达基因,取白来航和雪域白鸡样品进行q R T -P C R 分析,通过l o g 2(f o l d c h a ng e )对差异倍数进行转换㊂结果如图6所示,q R T -P C R 结果与R N A -s e q 测序结果表达趋势一致,表明转录85149514 10期陈雪娇等:鸡胚心脏组织转录组数据鉴定雪域白鸡高原低氧适应性关键基因表3转录因子与部分靶基因结合位点预测T a b l e3P r e d i c t e d b i n d i n g s i t e s o f t r a n s c r i p t i o n f a c t o r s t o s o m e t a r g e t g e n e sF O X P2m o t i f HO X A2m o t i fK C NM A1(C h r6)-4187T A G T A A A C A A T-4177;-2853G C T C A T T A C G-2844-2169G T G T A A A C A C G-2159;-2647A T G T A A A C A G A-2637;-2252C G G T A A A C A C A-2242;-1771C A A T A A A C A A A-1761A C T A2(C h r6)-4300G A G A A A A C A C A-4290;-1764A G G A A A A C A A A-1754;-677T A A T A A A C A A C-667A C T G2(C h r22)-2095A A G A A A A C A A G-2085;-4729C G A T A A A C A G G-4719+1表示转录起始位点;符号"-"指转录起始位点的上游;C h r6表示基因位于6号染色体t r a n s c r i p t i o n s t a r t s i t e(T S S)i s d e f i n e d a s+1;t h e s y m b o l - m e a n s t h e u p s t r e a m o f T S S;C h r6i n d i c a t e s t h a t t h e g e n e i s l o c a t e d o n c h r o m o s o m e 6红色三角形代表转录因子;蓝色方形代表靶基因(差异表达基因)T h e r e d t r i a n g l e s r e p r e s e n t t r a n s c r i p t i o n f a c t o r s;T h e b l u e s q u a r e s r e p r e s e n t t a r g e t g e n e s(D E G s)图4转录因子-靶基因预测网络F i g.4T r a n s c r i p t i o n f a c t o r-t a r g e t g e n e p r e d i c t i o n n e t w o r k畜 牧 兽 医 学 报54卷蓝色圆形代表转录因子;绿色三角形代表靶基因(差异表达基因);橙色方形代表通路B l u e c i r c l e s r e p r e s e n t t r a n s c r i p t i o n f a c t o r s ;G r e e n t r i a n g l e s r e p r e s e n t t a r g e t g e n e s (D E G s );T h e o r a n g e s q u a r e s r e pr e s e n t t h e p a t h w a ys 图5 转录因子与部分靶基因及通路预测网络F i g .5 T r a n s c r i p t i o n f a c t o r s a n d p a r t i a l t a r g e t g e n e s a n d p a t h w a ys p r e d i c t i o n n e t w o rk 图6 q R T -P C R 基因表达量结果F i g .6 q R T -P C R g e n e e x pr e s s i o n r e s u l t s 组测序结果的真实性和准确性㊂3 讨 论氧气是决定胚胎正常发育的关键因素之一,包括心脏发生[14]㊂心脏发生是一个复杂而精妙的过程,很容易受到自身基因表达变化和外界环境因素的影响而发育异常[15-16]㊂有研究指出,胎儿在母体子宫内的异常缺氧会对心脏发育产生不利影响,改变心肌结构,导致心功能下降[17]㊂长期暴露于高海拔地区提高了牦牛对低氧的生理反应,较大的心脏有利于它们适应低氧环境[18]㊂低氧孵化第16天的藏鸡胚胎心脏组织表达谱显示,藏鸡与低地鸡种差异表达基因主要为F G F R 1㊁C T G F ㊁A D AM 9㊁J PH 2㊁S A T B 1㊁B MP 4和H Y A L 1等,这些基因可能参与心肺系统发育[19]㊂本研究对高海拔孵化第16天的白来航鸡和雪域白鸡胚胎心脏组织进行基因表达谱分析,鉴定出的差异表达基因主要为T E NM 2㊁N O G ㊁S M O C 1㊁C C B E 1㊁E N D 2㊁A C T G 2㊁和MMP 2等,这些基因富集在心脏发育和血管发育等通路㊂这些结果表明,鉴定到的差异基因可能参与61410期陈雪娇等:鸡胚心脏组织转录组数据鉴定雪域白鸡高原低氧适应性关键基因鸡胚心血管发育和功能,是研究雪域白鸡适应低氧环境的候选基因㊂氧气与能量代谢有紧密的关系,机体生存和运动都离不开A T P供能,而A T P的产生依赖于氧气参与的氧化磷酸化[20]㊂因此当机体处于低氧环境时,能量代谢也会受到影响㊂先前的研究将高海拔地区的代谢适应归因于肌肉氧化能力的降低,其中乳酸脱氢酶(L D H)是厌氧糖酵解的关键酶,催化丙酮酸和乳酸之间的转化,在能量代谢过程中起关键作用[21]㊂高原牦牛背最长肌中的L D H活性较高,能利用有限的氧气促进碳水化合物的利用从而释放能量[18]㊂还原性辅酶N A D H在生化代谢反应中发挥重要作用,例如糖酵解和三羧酸循环等[22]㊂本研究鉴定的差异表达基因(D U S P5㊁U P B1㊁L P I N2㊁P L A2G4A和P D G F C等)也在能量代谢途径有富集,包括苹果酸脱氢酶(N A D+)活性和丙酮酸代谢等㊂表明能量代谢方面的差异可能是雪域白鸡适应低氧环境的原因㊂转录因子指能够以序列特异性方式结合D N A 并且调节转录的蛋白质[23]㊂转录因子与特异性D N A结合通常概括为 基序 (m o t i f),可用于扫描较长序列(例如启动子)以鉴定潜在的结合位点,转录因子在基因的启动子区有一个或多个结合位点[24]㊂转录因子与启动子结合,招募R N A聚合酶或其他因子,促进或者抑制基因的转录,从而调控生物功能[25]㊂低氧研究的明星基因H I F-1α,作为转录因子激活调控的靶基因表达[26-28]㊂血管内皮生长因子(v a s c u l a r e n d o t h e l i a l g r o w t h f a c t o r,V E G F)是H I F的靶基因,具有促进血管通透性增加㊁细胞外基质变性㊁血管内皮细胞迁移㊁增殖和血管形成等作用,缺氧条件下H I F-1可以调节V E G F的表达[29-30]㊂本研究鉴定了2个重要的转录因子F O X P2和H O X A2,可以靶向调控K C NM A1㊁C A C N G4㊁S C N4B㊁WN T2B㊁MM P2㊁C C B E1㊁A C T G2㊁A C T A2㊁P L A2G4A和T E NM2等基因表达㊂其中MMP2属于基质金属蛋白酶家族,低氧可以刺激MMP2的表达上调[31],参与多种生物学功能过程,包括血管平滑肌生长㊁增殖㊁迁移㊁松弛㊁血管生成和细胞凋亡等,与心血管发育密切相关[32-34]㊂A C T A2基因编码α-平滑肌肌动蛋白,通常在促进血管运动和收缩的血管平滑肌细胞中表达,参与血管收缩和血压稳态[35-36]㊂这些基因的功能均与心血管发育相关,参与心脏发育和功能相关通路㊂因此,本研究认为在低氧适应过程中,这些转录因子可能通过调控靶基因的转录表达而发挥作用,保证雪域白鸡胚胎期心脏正常发育,泵血功能正常,从而适应低氧环境㊂4结论雪域白鸡是西藏自主培育的品种,本研究通过高海拔孵化的鸡胚心脏转录组测序,发现了差异表达基因富集在心脏发育㊁血管发育和能量代谢途径,鉴定了转录因子F O X P2和HO X A2,它们可能通过调控靶基因的表达参与血管生成和心肌收缩等途径,为进一步研究鸡高原低氧适应分子机制提供了基础㊂参考文献(R e f e r e n c e s):[1]单增群佩.拉萨白鸡选育历程的回顾[J].西藏农业科技,2001,23(3):57-58.D A N Z Q P.A r e v i e w o f t h e s e l e c t i o n a n d b r e e d i n gp r o c e s s o f L h a s a w h i t e c h i c k e n[J].T i b e t J o u r n a l o fA g r i c u l t u r a l S c i e n c e s,2001,23(3):57-58.(i n C h i n e s e)[2] N E C HA E V A M V.P h y s i o l o g i c a l r e s p o n s e s t o a c u t ec h a n g e s i n t e m p e r a t u r e a nd o x y ge n a t i o n i n b i r d a n dr e p t i l e e m b r y o s[J].R e s p i r P h y s i o l N e u r o b i o l,2011,178(1):108-117.[3]张浩,吴常信,强巴央宗,等.氧气对低地鸡蛋胚胎死亡和孵化率的影响[J].畜牧兽医学报,2006,37(2):112-116.Z H A N G H,WU C X,Q I A N G B A Y Z,e t a l.I n f l u e n c e so f o x y g e n o n e m b r y o n i c m o r t a l i t y a n d h a t c h a b i l i t y o fc h i c k e n e g g s[J].A c t a V e t e r i n a r i a e t Z o o t e c h n i c a S i n i c a,2006,37(2):112-116.(i n C h i n e s e)[4]冯静,袁经纬,臧蕾,等.高海拔环境下不同品种鸡种蛋孵化效果研究[J].畜牧与饲料科学,2018,39(8):13-15.F E NG J,Y U A N J W,Z A N G L,e t a l.S t u d y o n t h eh a t c h i n g e f f e c t o f d i f f e r e n t s p e c i e s o f c h i c k e n e g g s i nh i g h a l t i t u d e a r e a[J].A n i m a l H u s b a n d r y a n d F e e dS c i e n c e,2018,39(8):13-15.(i n C h i n e s e)[5] Z HA N G H,B U R G G R E N W W.H y p o x i c l e v e l a n dd u r a t i o n d i f fe r e n t i a l l y af f e c t e m b r y o n i c o rg a n s y s t e md e v e l o p m e n t o f t h e c h i c k e n(G a l l u s g a l l u s)[J].P o u l t S c i,2012,91(12):3191-3201.[6]苟文钰.鸡胚胎低氧适应表型及心脏组织差异表达基因鉴定[D].北京:中国农业大学,2015.G O U W Y.I d e n t i f i c a t i o n o f h y p o x i c a d a p t a t i o n1614畜牧兽医学报54卷p h e n o t y p e s a n d t h e d i f f e r e n t i a l e x p r e s s i o n g e n e s o fm y o c a r d i a l t i s s u e i n c h i c k e n[D].B e i j i n g:C h i n aA g r i c u l t u r a l U n i v e r s i t y,2015.(i n C h i n e s e)[7] P E R T E A M,P E R T E A G M,A N T O N E S C U C M,e ta l.S t r i n g T i e e n ab l e s i m p r o v e d r ec o n s t r u c t i o n o f at r a n s c r i p t o m e f r o m R N A-s e q r e a d s[J].N a tB i o t e c h n o l,2015,33(3):290-295.[8] L O V E M I,H U B E R W,A N D E R S S.M o d e r a t e de s t i m a t i o n of f o l d c h a ng e a n d d i s p e r s i o n f o r R N A-s e qd a t a w i t h D E Se q2[J].G e n o m e B i o l,2014,15(12):550.[9] Y U B Q,WA N G X,S O N G Y T,e t a l.T h e r o l e o fh y p o x i a-i n d u c i b l e f a c t o r s i n c a r d i o v a s c u l a r d i s e a s e s[J].P h a r m a c o l T h e r,2022,238:108186. [10] P E N A E,E L A L A M S,S I Q U E S P,e t a l.O x i d a t i v es t r e s s a n d d i s e a s e s a s s o c i a t e d w i t h h i g h-a l t i t u d e e x p o s u r e[J].A n t i o x i d a n t s(B a s e l),2022,11(2):267. [11] L I Y,Z HA O L,L I X F.H y p o x i a a n d t h e t u m o rm i c r o e n v i r o n m e n t[J].T e c h n o l C a n c e r R e s T r e a t,2021,20,d o i:10.1177/15330338211036304.[12] S I MO N S O N T S,Y A N G Y Z,HU F F C D,e t a l.G e n e t i c e v i d e n c e f o r h i g h-a l t i t u d e a d a p t a t i o n i n t i b e t[J].S c i e n c e,2010,329(5987):72-75. [13] Q I X B,Z HA N G Q,H E Y X,e t a l.T h et r a n s c r i p t o m i c l a n d s c a p e o f y a k s r e v e a l s m o l e c u l a rp a t h w a y s f o r h i g h a l t i t u d e a d a p t a t i o n[J].G e n o m eB i o l E v o l,2019,11(1):72-85.[14] G I U S S A N I D A.B r e a t h o f l i f e:h e a r t d i s e a s e l i n k t od e v e l o p m e n t a l h y p o x i a[J].C i r c u l a t i o n,2021,144(17):1429-1443.[15] S M I T H K L M,S W I D E R S K A A,L O C K M C,e t a l.C h r o n i c d e v e l o p m e n t a l h y p o x i a a l t e r s m i t o c h o n d r i a lo x i d a t i v e c a p a c i t y a n d r e a c t i v e o x y g e n s p e c i e sp r o d u c t i o n i n t h e f e t a l r a t h e a r t i n a s e x-d e p e n d e n tm a n n e r[J].J P i n e a l R e s,2022,73(3):e12821. [16] M E N E N D E Z-MO N T E S I,E S C O B A R B,G OM E Z MJ,e t a l.A c t i v a t i o n o f a m i n o a c i d m e t a b o l i c p r o g r a m i nc a rd i a c H I F1-a l p h a-def i c i e n t m i c e[J].i S c i e n c e,2021,24(2):102124.[17] P A T T E R S O N A J,Z HA N G L.H y p o x i a a n d f e t a lh e a r t d e v e l o p m e n t[J].C u r r M o l M e d,2010,10(7):653-666.[18] A Y A L E W W,C HU M,L I A N G C N,e t a l.A d a p t a t i o n m e c h a n i s m s o f y a k(B o s g r u n n i e n s)t oh i g h-a l t i t u d e e n v i r o n m e n t a l s t r e s s[J].A n i m a l s(B a s e l),2021,11(8):2344.[19] Z HA N G Q,G O U W Y,WA N G X T,e t a l.G e n o m er e s e q u e n c i n g i d e n t i f i e s u n i q u e a d a p t a t i o n s o f T i b e t a nc h i c k e n s t o h y p o x i a a nd h i g h-d o se u l t r a v i o l e tr a d i a t i o n i n h i g h-a l t i t u d e e n v i r o n m e n t s[J].G e n o m eB i o l E v o l,2016,8(3):765-776.[20] R A D H A K R I S H N A N K,L A M A N N A J C,C A B R E R A ME.A q u a n t i t a t i v e s t u d y o f o x y g e n a s a m e t a b o l i cr e g u l a t o r[M]ʊD U N N J F,S WA R T Z H M.O x y g e nT r a n s p o r t t o T i s s u e X X I V.B o s t o n:S p r i n g e r,2003:547-554.[21] M A R K E R T C L.L a c t a t e d e h y d r o g e n a s e.B i o c h e m i s t r ya n d f u n c t i o n o f l a c t a t e d e h y d r o g e n a s e[J].C e l l B i o c h e mF u n c t,1984,2(3):131-134.[22] W I L S O N D F,MA T S C H I N S K Y F M.M e t a b o l i ch o m e o s t a s i s:o x i d a t i v e p h o s p h o r y l a t i o n a n d t h em e t a b o l i c r e q u i r e m e n t s o f h i g h e r p l a n t s a n d a n i m a l s[J].J A p p l P h y s i o l,2018,125(4):1183-1192.[23] L AM B E R T S A,J O L MA A,C AM P I T E L L I L F,e ta l.T h e h u m a n t r a n s c r i p t i o n f a c t o r s[J].C e l l,2018,172(4):650-665.[24] T O G N O N M,G I U G N O R,P I N E L L O L.A s u r v e y o na l g o r i t h m s t o c h a r a c t e r i z e t r a n s c r i p t i o n f a c t o rb i n d i n gs i t e s[J].B r i e f B i o i n f o r m,2023,24(3):b b a d156.[25] D E MA T T O S K,V I G E R R S,T R E M B L A Y J J.T r a n s c r i p t i o n f a c t o r s i n t h e r e g u l a t i o n o f l e y d i g c e l lg e n e e x p r e s s i o n a n d f u n c t i o n[J].F r o n t E n d o c r i n o l(L a u s a n n e),2022,13:881309.[26] X U R F,WA N G F,Y A N G H Q,e t a l.A c t i o n s i t e sa n d c l i n i c a l a p p l i c a t i o n o f H I F-1αi n h ib i t o r s[J].M o l e c u l e s,2022,27(11):3426.[27] P E N G X F,G A O H,X U R,e t a l.T h e i n t e r p l a yb e t w e e n H I F-1αa n d n o nc od i n g R N A s i n c a n ce r[J].JE x p C l i n C a n c e r R e s,2020,39(1):27.[28] WU F,T O N G D D,N I L,e t a l.H I F-1αs u p p r e s s e sm y e l o m a p r o g r e s s i o n b y t a r g e t i n g M c l-1[J].I n t JC l i n E x p P a t h o l,2020,13(7):1483-1491.[29] T A N G K C,B R E E N E C,WA G N E R H,e t a l.H I Fa n d V E G F r e l a t i o n s h i p s i n r e s p o n s e t o h y p o x i a a n ds c i a t i c n e r v e s t i m u l a t i o n i n r a t g a s t r o c n e m i u s[J].R e s p i r P h y s i o l N e u r o b i o l,2004,144(1):71-80. [30] S A D E G H I F,K A R D A R G A,B O L O U R I M R,e t a l.O v e r e x p r e s s i o n o f b H L H d o m a i n o f H I F-1f a i l e d t oi n h i b i t t h e H I F-1t r a n s c r i p t i o n a l a c t i v i t y i n h y p o x i a[J].B i o l R e s,2020,53(1):25.[31] Z E N G W,S U J,WU L,e t a l.C D147p r o m o t e s m e l a n o m ap r o g r e s s i o n t h r o u g h h y p o x i a-i n d u c e d MM P2a c t i v a t i o n[J].C u r r M o l M e d,2014,14(1):163-173.[32] R A F F E T T O J D,K H A L I L R A.M a t r i x m e t a l l o p r o t e i n a s e sa n d t h e i r i n h ib i t o r s i n v a sc u l a r r e m ode l i n g a n d261410期陈雪娇等:鸡胚心脏组织转录组数据鉴定雪域白鸡高原低氧适应性关键基因v a s c u l a r d i s e a s e[J].B i o c h e m P h a r m a c o l,2008,75(2):346-359.[33] S I E F E R T S A,S A R K A R R.M a t r i x m e t a l l o p r o t e i n a s e s i nv a s c u l a r p h y s i o l o g y a n d d i s e a s e[J].V a s c u l a r,2012,20(4):210-216.[34] WA N G X,K HA L I L R A.M a t r i x m e t a l l o p r o t e i n a s e s,v a s c u l a r r e m o d e l i n g,a n d v a s c u l a r d i s e a s e[J].A d vP h a r m a c o l,2018,81:241-330.[35] Y U A N S M.α-s m o o t h m u s c l e a c t i n a n d A C T A2g e n ee x p r e s s i o n s i n v a s c u l o p a t h i e s[J].B r a z J C a r d i o v a s cS u r g,2015,30(6):644-649.[36] K AW A,K AW K,HO S T E T L E R E M,e t a l.E x p a n d i n g A C T A2g e n o t y p e s w i t h c o r r e s p o n d i n gp h e n o t y p e s o v e r l a p p i n g w i t h s m o o t h m u s c l ed y s f u n c t i o n s y n d r o m e[J].A m J Me d G e n e t P a r t A,2022,188(8):2389-2396.(编辑郭云雁)3614。
基于网络药理学与分子对接技术探讨小儿清热止咳口服液治疗小儿发热作用机制

武 薇1 徐梓轩1. 承德医学院,2. 河北省药品医疗器械检验研究院,3. 河北省神经损伤与修复重点实验室,【摘要】目的:物过程和通路,选出小儿清热止咳口服液的活性成分,关靶点取交集,软件中构建“小儿清热止咳口服液成分蛋白相互作用网并得到关键靶点。
通过Cytoscape 3.7.2小儿清热止咳口服液是由麻黄、炒苦杏仁、石膏、甘草、黄芩、板蓝根、北豆根七味中药制成的复方制剂,原方来自我国汉代名医张仲景的《伤寒杂病论》的麻杏石甘汤[1]。
具有清热宣肺,平喘,利咽的功效。
用于小儿外感风热所致的感冒,症见发热恶寒、咳嗽痰黄、气促喘息、口干音哑、咽喉肿痛[2]。
小儿清热止咳口服液在治疗小儿发热方面具有优势,可充分利用药物的不同性能、功用,而进行动态的、个性化的、切中病证之本源的综合治疗[3]。
但其确切机制尚不清晰,因此本文基于网络药理学及分子对接技术,筛选出小儿清热止咳口服液有效活性成分和作用靶点,构建“中药-活性成分-靶点”、靶蛋白相互作用、靶点功能通路等多层次生物信息网络,为小儿清热止咳口服液用于治疗小儿发热提供理论依据[4]。
本研究的思路如Fig.1所示。
1 方法1.1 药物活性成分的获取及筛选中药系统药理学数据库与分析平台TCMSP (Traditional Chinese Medicine Systems Pharmacology Database and Analysis Platform)(http:/// tcmsp.php)搜寻小儿清热止咳口服液的7味中药(麻黄、炒苦杏仁、板蓝根、北豆根、甘草、黄芩、石膏)的化学成分。
根据药动学性质参数,包括口服生物利用度(oral bioavailability,OB)、类药性(drug-likeness,DL)、血脑屏障(blood-brainbarrie,BBB)分别对每个药物进行筛选以得到活性成分,因小儿清热口服液是口服方剂,因此OB≥30%;由于药物在体内需要依靠靶器官和靶细胞的生理功能特性,通过复杂的生物学过程,最终与相应靶点结合,才能发挥其理想的生物学效应,活性成分必须具有较高的类药性,因此设置DL≥0.18为筛【ABSTRACT】Objective:By constructing the interaction networks between the compositions of Xiao’er Qingre Zhike Koufuye and target networks and proteins,we conducted the enrichment analysis to determine the biological processes and involved pathways,we also studied the potential action mechanism of fever in children. Methods:The active components of Xiao’er Qingre Zhike Koufuye were selected and the target points were predicted by searching TCMSP database,and the potential action targets were obtained by crossing Genecards database with Drug Bank database of the fever-related targets. After processing the comparison results,the targets were input into Cytoscape3.7.2 software to build the “Xiao’er Qingre Zhike Koufuye ingredients-compounds-fever targets” network,and the String database and Cytoscape3.7.2 database were used to construct the protein interaction network and to obtain the key targets. The GO bio-function and KEGG pathway enrichment analysis were later performed through the DAVID v6.8 database and the enrichment results were visualized with Cytoscape3.7.2. Finally,the molecular docking of the key compounds and the targets were performed. Results:160 active components,584 active component targets,and 4 137 known fever in children-related target genes were obtained. The interaction of disease genes and drug targets yielded 346 intersection targets,and 18 core targets were obtained by the protein interaction network and topological analysis. Enrichment analysis showed that the main pathways focused on neuroactive ligand-receptor-related pathways,immune inflammation pathways,and other pathways such as neurotoxin addiction and viral infection. Further screening of core targets and compounds for molecular docking showed that Casp3,CREB1,Fos,IL-1B,IL-6,INS,Jun,and MYC as core target proteins could stably bind to small molecules such as D-norpseudoephedrine,beta-sitosterol,matrine and ephedrine. Conclusion:Through network pharmacology and molecular docking,our study found the possible active ingredients of Xiao’er Qingre Zhike Koufuye and the potential targets of fever in children and predicted the key ways to treat fever in children. It provided a theoretical basis for explaining the action mechanism of Xiao’er Qingre Zhike Koufuye in children and new methods for further studying the molecular action mechanism. Besides,it supplied a new idea for drug development of children fever.【KEY WORDS】 Xiao’er Qingre Zhike Koufuye;fever in children;network pharmacology;molecular docking technology;由于治疗发热的药物成分需要通过血脑屏障才能对机体发挥作用,因此设定BBB≥0.30。
蛹虫草转录组分析及类胡萝卜素生物合成相关基因的挖掘

蛹虫草转录组分析及类胡萝卜素生物合成相关基因的挖掘娄海伟1,2,赵 玉1,赵 逸2,林俊芳2,3,*,赵仁勇1,叶志伟2,3,郭丽琼2,3,*(1.河南工业大学粮油食品学院,河南 郑州450001;2.华南农业大学食品学院,广东 广州510642;3.广东省微生态制剂工程技术研究中心,广东 广州510642)摘 要:基于培养基显著影响蛹虫草类胡萝卜素的生成,采用高通量测序技术探究不同培养基培养的蛹虫草菌丝(CM10_RL和CM10_WL)的转录组差异,经生物信息学分析挖掘蛹虫草类胡萝卜素的生物合成相关基因。
结果表明,样品CM10_WL的类胡萝卜素含量显著高于样品CM10_RL的类胡萝卜素含量;相对于样品CM10_RL,在样品CM10_WL中检测到318 个上调表达基因和618 个下调表达基因。
基因本体论功能富集结果表明差异表达基因(differentially expressed gene,DEG)主要富集在代谢过程、细胞膜、催化活性条目中。
京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集结果表明DEG主要富集在代谢途径。
蛹虫草转录本与KEGG数据库中的类胡萝卜素生物合成途径比对结果表明基因CCM_06728和CCM_09155参与类胡萝卜素的生物合成。
本研究为进一步阐明蛹虫草类胡萝卜素的生物合成途径奠定基础。
关键词:蛹虫草;高通量测序;转录组;色素;类胡萝卜素;差异表达基因Transcriptomic Analysis of Cordyceps militaris and Mining of Genes Involved in Carotenoid Biosynthesis LOU Haiwei1,2, ZHAO Yu1, ZHAO Yi2, LIN Junfang2,3,*, ZHAO Renyong1, YE Zhiwei2,3, GUO Liqiong2,3,*(1. College of Food Science and Engineering, Henan University of Technology, Zhengzhou 450001, China;2.College of Food Science, South China Agricultural University, Guangzhou 510642, China;3. Research Center for Micro-Ecological Agent Engineering and Technology of Guangdong Province, Guangzhou 510642, China)Abstract: Culture medium composition significantly affects carotenoid biosynthesis in Cordyceps militaris. In this study, high-throughput sequencing technology was used to investigate the transcriptomic differences of C. militaris mycelia (CM10_RL and CM10_WL) cultured with different media. The genes related to the biosynthesis of carotenoids in C. militaris were identified by bioinformatics analysis. The results showed that the carotenoid content of CM10_WL was significantly higher than that of CM10_RL. A total of 318 up-regulated genes and 618 down-regulated genes were found in CM10_WL compared with CM10_RL. Gene Ontology (GO) analysis indicated that the differentially expressed genes (DEGs) were mainly classified into the “metabolic process”, “membrane” or “catalytic activity” terms. Kyoto Encyclopedia of Genes and Genomes (KEGG) pathway enrichment analysis suggested that these DEGs were mainly enriched in “metabolic pathways”.Therefore, all the transcripts derived from RNA-seq data were searched against the carotenoid biosynthetic pathway in the KEGG database, and the results showed that the CCM_06728 and CCM_09155 genes were involved in the biosynthesis of carotenoids. This study lay a foundation for further elucidating the biosynthetic pathway of carotenoids in C. militaris.Keywords: Cordyceps militaris; high-throughput sequencing; transcriptome; pigment; carotenoid; differentially expressed geneDOI:10.7506/spkx1002-6630-20200125-269中图分类号:TS201.3 文献标志码:A 文章编号:1002-6630(2021)06-0150-07收稿日期:2020-01-25基金项目:广东省重点领域研发计划项目(2018B020205001;2018B020205003);广东省现代农业食用菌产业技术体系项目(2019KJ103);国家自然科学基金面上项目(31772373;31572178);河南工业大学高层次人才科研基金项目(2020BS001);国家自然科学基金-河南省联合基金重点项目(U1604234)第一作者简介:娄海伟(1983—)(ORCID: 0000-0003-0987-0400),男,博士,研究方向为天然产物活性成分。
肾透明细胞癌舒尼替尼耐药的分子标志物筛选及相关生物学功能分析

第50卷第2期第142页2021年4月 华中科技大学学报(医学版)A c t aM e dU n i vS c iT e c h n o lH u a z h o n gV o l .50 N o .2 P .142A pr . 2021*国家自然科学基金资助项目(N o .81927807)#共同第一作者刘郴郴,男,1995年生,医学硕士,E -m a i l :l i u c c 1325@126.c o m宋正帅,男,1988年生,医学博士,E -m a i l :867703966@q q.c o m ә通讯作者,C o r r e s p o n d i n g a u t h o r ,E -m a i l :x z h a n g@h u s t .e d u .c n 肾透明细胞癌舒尼替尼耐药的分子标志物筛选及相关生物学功能分析*刘郴郴1,2#, 宋正帅3#, 章小平1,2ә华中科技大学同济医学院附属协和医院1泌尿外科2泌尿外科研究所,武汉 4300223华中科技大学同济医学院附属武汉中心医院泌尿外科,武汉 430014摘要:目的 通过公共数据库,利用生物信息学方法分析对舒尼替尼敏感或耐药的肾透明细胞癌(c c R C C )样本间的基因表达差异,筛选出舒尼替尼耐药相关分子并探讨介导获得性耐药的潜在信号通路和分子机制㊂方法 从G E O 数据库中下载对舒尼替尼敏感或耐药的c c R C C 基因表达谱,利用G E O 2R 在线工具对不同表达水平基因进行筛选得到差异表达基因,构建差异基因的蛋白-蛋白互作网络,通过蛋白互作网络中的高连通性确定枢纽基因㊂对表达水平上调基因㊁下调基因以及枢纽基因进行分子功能㊁信号通路以及基因集富集分析(G S E A ),发掘耐药的潜在机制㊂基于T C G A 数据库,分析枢纽基因表达水平和c c R C C 患者总生存期及无病生存期之间的关系,并利用C o x 回归模型探讨靶基因和肾癌患者各种临床病理参数的相关性㊂结果 筛选得到的舒尼替尼耐药样本差异表达基因中有上调基因38个,下调基因57个及10位枢纽基因,G O /K E G G 分析提示上调基因及前10位枢纽基因的生物过程富集在Ⅰ型干扰素信号通路㊁对病毒的防御反应;分子功能涉及2'-5'-o l i g o a d e n y l a t e 合成酶活性㊁双链R N A 结合㊁转移酶活性;信号通路富集在甲型流感㊁麻疹㊁单纯疱疹病毒感染和肠道免疫网络中产生I g A ;G S E A 结果显示舒尼替尼耐药组富集在复制应激㊁D N A 感应㊁干扰素激活等通路㊂生存分析表明I L 6㊁I S G 15㊁O A S L 高表达预示更差的生存期;I F I T 3㊁I S G 15㊁O A S L 的R O C 曲线下面积为0.7362~0.8914,且在肾癌组织中的表达明显上调,提示其具有潜在诊断价值,其中I S G 15的表达与c c R C C 的各种临床病理参数相关,可能促进肾癌的进展和转移㊂结论 I S G 15㊁I F I T 3㊁O A S L 是预测c c R C C 舒尼替尼耐药的潜在分子标志物,其中I S G 15的表达水平与c c R C C 的临床病理特征相关,是c c R C C 的独立预后因素,有望成为治疗c c R C C 的重要靶点㊂关键词:肾透明细胞癌; 舒尼替尼耐药; 分子标志物; 生物信息学中图分类号:R 737.11 D O I :10.3870/j.i s s n .1672-0741.2021.02.002S c r e e n i n g ofN e wB i o m a r k e r s f o r S u n i t i n i bR e s i s t a n c e i nC l e a rC e l lR e n a l C e l l C a r c i n o m a a n dA n a l y s i s o fR e l a t e dB i o l o gi c a l F u n c t i o n s L i uC h e n c h e n 1,2#,S o n g Z h e n g s h u a i 3#,Z h a n g X i a o p i n g1,2ә1D e p a r t m e n t o f U r o l o g y ,2I n s t i t u t e o f U r o l o g y ,U n i o n H o s p i t a l ,T o n g j iM e d i c a lC o l l e ge ,H u a z h o n g U n i v e r s i t y of S c i e n c e a n dT e c h n o l og y ,W u h a n430022,C h i n a 3D e p a r t m e n t o f U r o l o g y ,T h e C e n t r a lH o s p i t a l o f W u h a n ,T o n g j iM e d i c a lC o l l e ge ,H u a z h o n g U n i v e r s i t y of S c i e n c e o f T e c h n o l og y ,W u h a n430014,C h i n a A b s t r a c t O b je c t i v e T oa n a l y z et h e g e n e so fd if f e r e n t i a le x p r e s s i o nb e t w e e ns u n i t i n i b -s e n s i t i v ea n ds u n i t i n i b -r e s i s t a n t c c R C Cs a m p l e s b y b i o i n f o r m a t i c s a n a l y s i s b a s e d o n p u b l i c d a t a b a s e s a n d s c r e e n o u t r e l a t e dm o l e c u l e s t o e x p l o r e p o t e n t i a l s ig n a -l i n gp a th w a y s a n d m o l e c u l a rm e c h a ni s m s t h a tm e d i a t ea c q u i r e dd r u g r e s i s t a n c e .M e t h o d s T h e g e n ee x pr e s s i o n p r o f i l e sw e r e d o w n l o a d e d f r o mt h eG E Od a t a b a s e ,a n d t h e d i f f e r e n t i a l l y e x p r e s s e d g e n e sw e r e s c r e e n e du s i n g th eG E O 2R .T h e n t h e p r o t e i n -p r o t e i n i n t e r a c t i o nn e t w o r k so f t h ed i f f e r e n t i a l l y e x p r e s s e d g e n e sw e r ec o n s t r u c t e d ,a n dt h eh u b g e n e sw e r e i d e n t i f i e db y th e h i g hc o n n e c t i v i t y i n t h e p r o t e i n i n t e r a c t i o nn e t w o r k s .T h em o l e c u l a r f u n c t i o n ,s i g n a l i n gp a t h w a y a n d g e n e s e t e n r i c h m e n t a n a l y-s i sw e r e c a r r i e do u t f o r u p -r e g u l a t e d g e n e s ,d o w n -r e g u l a t e d g e n e s a n dh u b g e n e s t o e x p l o r e t h e p o t e n t i a lm e c h a n i s mo f d r u g re -s i s t a n c e .T h e r e l a t i o n s h i p b e t w e e nh u b g e n e e x p r e s s i o n l e v e l s a n d l if e t i m e o f c c R C C p a t i e n t sw a s a n a l yz e db a s e do n t h eT C G A d a t a b a s e .F i n a l l y ,t h e c o r r e l a t i o nb e t w e e n t h e s c r e e n e d g e n e s a n dv a r i o u s c l i n i c o p a t h o l o gi c a l f a c t o r so f c c R C C p a t i e n t sw a se x -p l o r e db y C o x r e g r e s s i o nm o d e l .R e s u l t s T h e r ew e r e 38u p -r e g u l a t e d g e n e s ,57d o w n -r e gu l a t e d g e n e s a n d10h u b g e n e s i n t h e d r u g -r e s i s t a n t s a m p l e s ,a n dG O /K E G Ga n a l y s i s i n d i c a t e d t h a t t h eu p -r e gu l a t e d g e n e s a n d t h e f i r s t 10h u b g e n e sw e r e e n r i c h e di n t h e t y p e I i n t e r f e r o n s i g n a l i n g p a t h w a y a n d t h e d e f e n s e r e s p o n s e t o t h e v i r u s.T h em o l e c u l a r f u n c t i o n s i n v o l v e d2'-5'-o l i g o a d e-n y l a t e s y n t h e t a s ea c t i v i t y,d o u b l e-s t r a n d e d R N A b i n d i n g a n dt r a n s f e r a s ea c t i v i t y.A n dt h es i g n a l i n gp a t h w a y so f t h o s e g e n e s w e r e e n r i c h e d i n i n f l u e n z aA,m e a s l e s,h e r p e s s i m p l e x i n f e c t i o n s,a n d i n t e s t i n a l i m m u n e n e t w o r k s t o p r o d u c e I g A.G S E Ar e s u l t s s h o w e d t h a tt h es u n i t i n i b-r e s i s t a n t g r o u p w a se n r i c h e di nr e p l i c a t i o ns t r e s s,D N A s e n s i n g,a n di n t e r f e r o na c t i v a t i o n p a t h-w a y s.S u r v i v a l a n a l y s i s s h o w e d t h a t h i g he x p r e s s i o n l e v e l so f I L6,I S G15a n dO A S L p r e d i c t e d p o o r e r s u r v i v a l.T h e a r e au n d e r t h eR O Cc u r v e s o f I F I T3,I S G15a n dO A S Lw e r e0.7362-0.8914,a n d t h e e x p r e s s i o n o f t h o s e g e n e sw a s s i g n i f i c a n t l y u p-r e g u-l a t e d i n c c R C Ct i s s u e s,s u g g e s t i n g t h e i r p o t e n t i a l d i a g n o s t i c v a l u e.T h e e x p r e s s i o n l e v e l o f I S G15c o r r e l a t e dw i t h v a r i o u s c l i n i c o-p a t h o l o g i c a l p a r a m e t e r s o f c c R C Ca n dm i g h t p r o m o t e c c R C C p r o g r e s s i o na n dm e t a s t a s i s.C o n c l u s i o n I S G15,I F I T3,a n dO A S L a r e e f f e c t i v e b i o m a r k e r s f o r p r e d i c t i n g s u n i t i n i b r e s i s t a n c e,i nw h i c h t h e e x p r e s s i o n l e v e l o f I S G15c o r r e l a t e sw i t h t h e c l i n i c a l a n d p a t h o l o g i c a l f e a t u r e s o f c c R C C,w h i c h i s e x p e c t e d t ob e a n i m p o r t a n t t a r g e t f o r t h e t r e a t m e n t o f c c R C C.K e y w o r d s c c R C C;s u n i t i n i b r e s i s t a n c e;b i o m a r k e r;b i o i n f o r m a t i c s肾癌是泌尿系统最常见的恶性肿瘤之一,其中又以肾透明细胞癌(c l e a r c e l l r e n a l c e l l c a r c i n o m a, c c R C C)占大多数(75%)[1-2]㊂约30%的肾癌患者在初诊时发现转移病灶以及30%的患者在行根治性肾切除术后发现复发是造成患者不良预后的主要原因[3-4]㊂目前针对进展期肾癌,随机临床试验证实靶向血管内皮生长因子(V E G F)等通路的抗血管生成药物,包括舒尼替尼㊁帕唑帕尼等,能够有效缓解疾病进展,并已得到广泛应用[5]㊂然而,目前仍缺乏能有效预测c c R C C靶向药物耐药性的分子标记物,因此,寻找和确认相应的分子标记物以预测疗效,探索耐药机制,是精准治疗的关键[1]㊂近年来,随着高通量芯片和生物信息学技术的发展,我们可以获取和分析肿瘤㊁肿瘤毗邻组织和正常组织在多种基因表达水平上的差异,从而发现肿瘤发展㊁侵袭㊁转移㊁复发以及靶向药物耐药等表型中的潜在分子机制㊂本研究基于T C G A㊁G E O等公共数据库,利用在线生物信息学工具和软件对数据进行筛选㊁分析㊁整理,得到c c R C C对舒尼替尼获得性耐药的分子标志物,为患者预后判断及治疗选择提供依据,并对其中潜在的信号通路及分子机制进行了探讨,为后续通过实验及临床数据进行进一步验证奠定了基础㊂1材料和方法1.1微阵列数据从G E O数据库中下载基因表达谱G S E76068㊂根据安捷伦G P L10558平台(I l l u m i n aH u m a n h t-12 v4.0表达芯片)提供的信息,G S E76068中共有24个样本,包括8个未处理的c c R C C样本,8个舒尼替尼敏感样本及8个舒尼替尼耐药样本㊂G S E76068中包含这24个样本的全基因组m R N A表达数据㊂1.2筛选差异表达基因(D E G s)利用基于R语言的G E O数据便捷在线分析工具G E O2R(h t t p s://w w w.n c b i.n l m.n i h.g o v/g e o/ G E O2r/),通过将 a d j.P<0.05 和 |l o g F C|>1 作为分界标准,得到舒尼替尼敏感样本和舒尼替尼耐药样本之间的D E G s㊂1.3蛋白-蛋白互作网络(P P I)及模块分析基于相应D E G s蛋白之间物理和功能联系,利用相互作用基因检索工具(S T R I N G,h t t p s:// s t r i n g-d b.o r g/c g i/n e t w o r k.p l)构建了P P I㊂同时根据最大交互数量ɤ5个,置信度ȡ0.4分的标准,用S T R I N G绘制并筛选出了10个枢纽基因㊂此外,利用分子复合体检测(M C O D E)插件,按照节点得分值=0.2,K值ȡ2,最大深度=100,在C y t o-s c a p e软件中构建了各模块的P P I网络㊂1.4D E G s和枢纽基因的G O和K E G G通路分析利用D A V I D(h t t p s://d a v i d.n c i f c r f.g o v/s u m-m a r y.j s p)数据库对D E G s和枢纽基因的基因本体(G O)和京都基因与基因组百科全书(K E G G)信号途径进行分析㊂D A V I D是一个免费开放的在线工具,为大规模的基因或蛋白质列表提供了系统全面的生物注释信息[6]㊂统计偏差的截止标准为P<0.05㊂1.5基因集富集分析(G S E A)为寻找c c R C C舒尼替尼耐药的潜在生物学功能和途径,进行了G S E A分析㊂16个c c R C C样本基因表达信息从G S E76068下载后,按舒尼替尼敏感和舒尼替尼耐药分为2组㊂选择A n n o t a t e d g e n e s e t s c2.c p.v6.2.s y m b o l s.g m t作为参考基因集,I l-l u m i n aH u m a n H T-12V4.0表达芯片作为芯片平台,并以P<0.05㊁F D R<0.25㊁基因大小ȡ100为分界标准㊂1.6枢纽基因的诊断㊁预后和临床病理价值分析从T C G A数据库中获取534例c c R C C患者总生存期(O S)和无病生存期(D F S)信息,以及患者年龄㊁性别㊁肿瘤T NM分期和G分级等临床信息(无特殊筛选标准),通过G r a p h P a dP r i s m6.0软件获得K a p l a n-M e i e r曲线,评估枢纽基因的预后价值㊂并通过受试者工作特征(R O C)曲线分析,确定这些枢纽基因对c c R C C的诊断价值㊂综合上述结果,重点评估靶基因I S G15的临床病理价值㊂㊃341㊃刘郴郴等.肾透明细胞癌舒尼替尼耐药的分子标志物筛选及相关生物学功能分析1.7统计学方法本研究采用G r a p h P a d P r i s m6.0(G r a p h P a d S o f t w a r e,I n c,U S A)和S P S S22.0(I B M S P S S,C h i-c a g o,I L)为统计分析软件,各组数据以平均值ʃ标准差表示,以P<0.05为差异具有统计学意义㊂采用非配对t检验分析c c R C C与正常肾组织之间枢纽基因表达的差异;通过M a n n-W h i t n e y检验统计目标基因和肾癌患者临床病理参数之间的联系;通过K a p l a n-M e i e r曲线和对数秩检验计算基因表达水平与患者O S㊁D F S的相关性,再结合O S和患者性别㊁年龄㊁T NM分期等变量做单因素C o x分析,并将筛选到的显著相关变量进行多因素C o x比例风险回归分析㊂2结果2.1筛选D E G s在G S E76068中,共有8个舒尼替尼敏感和8个舒尼替尼耐药的c c R C C样本㊂以P<0.05㊁|l o g F C|ȡ1为阈值,通过G E O2R分析出D E G s,其中包括表达上调基因38个,表达下调基因57个㊂2.2枢纽基因筛选与P P I网络构建根据连接度由高到低确定了10个枢纽基因,包括I L6㊁M X1㊁I S G15㊁I F I T1㊁O A S1㊁O A S2㊁I F I T3㊁I F I27㊁R S A D2㊁O A S L,其连接度依次为22㊁17㊁16㊁15㊁15㊁15㊁15㊁15㊁14㊁14㊂有趣的是,我们发现一半的枢纽基因属于干扰素相关的D N A损伤抗性标志基因(I R D S)[7]㊂根据S T R I N G查询的蛋白信息,我们分别构建了所有D E G s和前10位枢纽基因的P P I网络(图1A㊁1B);此外,C y t o s c a p e中的M C O D E插件识别出P P I网络中的前3个重要模块(图1C~1E)㊂2.3功能富集分析本研究通过D A V I D在线平台完成了G O功能和K E G G通路富集的分析㊂表1㊁2显示了D E G s的前5位基因本体类别㊂在生物过程(B P)方面,上调的D E G s 主要富集在Ⅰ型干扰素信号通路㊁对病毒的防御反应;在分子功能(M F)方面,上调的D E G s主要涉及2'-5'-o l i g o a d e n y l a t e合成酶活性㊁双链R N A结合㊁转移酶活性㊂此外,细胞成分(C C)分析显示,它们与胞质㊁线粒体有关㊂而K E G G途径主要富集在甲型流感㊁麻疹㊁单纯疱疹病毒感染和肠道免疫网络中产生I g A(表3)㊂其中图2A㊁2B给出了这些D E G s的G O和K E G G通路富集图㊂为了更全面地了解这些D E G s,我们对前10位枢纽基因进行了G O功能和K E G G通路富集㊂有趣的是如表4㊁5㊁6所示,这些枢纽基因的B P和K E G G通路富集在Ⅰ型干扰素信号通路㊁对病毒的防御反应㊁对病毒的反应㊁甲型流感㊁麻疹㊁单纯疱疹病毒感染等方面,与上调的D E G s和第1位模块基因的结果基本一致㊂A:D E G s互作网络;B:枢纽基因互作网络;C:模块1;D:模块2;E:模块3图1差异表达基因㊁枢纽基因和3个模块的蛋白-蛋白相互作用网络分析F i g.1P r o t e i n-p r o t e i n i n t e r a c t i o nn e t w o r ko f t h eD EG s,t o p10h u b g e n e s a n d3m o d u l a r a n a l y s e s㊃441㊃华中科技大学学报(医学版)2021年4月第50卷第2期表1 与c c R C C 舒尼替尼耐药相关上调表达基因G O 分析T a b l e 1 G e n e o n t o l o g y a n a l y s i s o f u p r e g u l a t e dd i f f e r e n t i a l l y e x pr e s s e d g e n e s a s s o c i a t e dw i t h s u n i t i n i b r e s i s t a n c e 类别项目数量%P 值生物过程(B P )G O :0060337~t y p e Ⅰi n t e r f e r o n s i g n a l i n gp a t h w a y 1338.23<0.01G O :0051607~d e f e n s e r e s p o n s e t ov i r u s 1441.17<0.01G O :0009615~r e s po n s e t ov i r u s 1135.29<0.01G O :0045071~n e g a t i v e r e g u l a t i o no f v i r a l g e n o m e r e pl i c a t i o n 823.52<0.01G O :0035457~c e l l u l a r r e s p o n s e t o i n t e r f e r o n -a l p h a 38.82<0.01细胞成分(C C )G O :0005829~c y t o s o l 1338.230.003G O :0005737~c y t o pl a s m 1647.050.007G O :0005739~m i t o c h o n d r i o n720.580.016G O :0005741~m i t o c h o n d r i a l o u t e rm e m b r a n e 38.820.023分子功能(M F )G O :0001730~2'-5'-o l i g o a d e n y l a t e s y n t h e t a s e a c t i v i t y38.82<0.01G O :0003725~d o u b l e -s t r a n d e dR N Ab i n d i n g 38.820.005G O :0016740~t r a n s f e r a s e a c t i v i t y 38.820.012G O :0005515~p r o t e i nb i n d i n g 2367.640.014G O :0005525~G T Pb i n d i n g411.740.030表2 与c c R C C 舒尼替尼耐药相关下调表达基因G O 分析T a b l e 2 G e n e o n t o l o g y a n a l y s i s o f d o w n r e g u l a t e dd i f f e r e n t i a l l y e x pr e s s e d g e n e s a s s o c i a t e dw i t h s u n i t i n i b r e s i s t a n c e 类别项目数量%P 值生物过程(B P )G O :0072593~r e a c t i v e o x y g e n s p e c i e sm e t a b o l i c p r o c e s s 40.06<0.01G O :0007155~c e l l a d h e s i o n 80.12<0.01G O :0006885~r e gu l a t i o no f p H 30.05<0.01G O :0033574~r e s po n s e t o t e s t o s t e r o n e 30.050.002G O :0071356~c e l l u l a r r e s p o n s e t o t u m o r n e c r o s i s f a c t o r 40.060.002细胞成分(C C )G O :0005615~e x t r a c e l l u l a r s pa c e 130.19<0.01G O :0005576~e x t r a c e l l u l a r r e g i o n 110.160.002G O :0005578~p r o t e i n a c e o u s e x t r a c e l l u l a rm a t r i x 50.070.003G O :0045121~m e m b r a n e r a f t40.060.011G O :0005577~f i b r i n o g e n c o m p l e x 20.030.020分子功能(M F )G O :0005178~i n t e g r i nb i n d i n g 60.09<0.01G O :0050840~e x t r a c e l l u l a rm a t r i xb i n d i n g 40.06<0.01G O :0008201~h e p a r i nb i n d i n g60.09<0.01G O :0001968~f i b r o n e c t i nb i n d i n g 30.040.001G O :0070051~f i b r i n o g e nb i n d i n g20.030.007表3 与c c R C C 舒尼替尼耐药相关差异表达基因K E G G 分析T a b l e 3 K E G G p a t h w a y a n a l y s i s o f d i f f e r e n t i a l l y e x pr e s s e d g e n e s a s s o c i a t e dw i t h s u n i t i n i b r e s i s t a n c e 类别项目数量%P 值表达上调基因h s a 05164:I n f l u e n z aA 514.70<0.01h s a 05162:M e a s l e s411.760.001h s a 04672:I n t e s t i n a l i m m u n e n e t w o r k f o r I g A p r o d u c t i o n 38.820.003h s a 05168:H e r p e s s i m pl e x i n f e c t i o n 411.760.004h s a 04060:C y t o k i n e -c y t o k i n e r e c e p t o r i n t e r a c t i o n 411.760.009表达下调基因h s a 05144:M a l a r i a40.06<0.01h s a 04360:A x o n g u i d a n c e 40.060.001h s a 04510:F o c a l a d h e s i o n40.060.026h s a 04668:T N Fs i g n a l i n gp a t h w a y30.040.045h s a 04380:O s t e o c l a s t d i f f e r e n t i a t i o n30.040.064㊃541㊃刘郴郴等.肾透明细胞癌舒尼替尼耐药的分子标志物筛选及相关生物学功能分析A :上调D E G s 的G O 分析;B :下调D E G s 的G O 分析;C :DE G s 的K E G G 通路分析图2 D E G s 的G O 和K E G G 通路富集分析F i g.2G Oa n dK E G G p a t h w a y s e n r i c h m e n t a n a l y s i s o fD E G s 表4 与c c R C C 舒尼替尼耐药相关枢纽基因G O 分析T a b l e 4 G e n e o n t o l o g y a n a l ys i s o f h u b g e n e s a s s o c i a t e dw i t h c c R C Cs u n i t i n i b r e s i s t a n c e 类别项目数量%P 值生物过程(B P )G O :0060337~t y p eⅠi n t e r f e r o n s i g n a l i n gp a t h w a y1317.564.78E -17G O :0051607~d e f e n s e r e s p o n s e t ov i r u s 1621.623.46E -16G O :0009615~r e s p o n s e t ov i r u s 1216.211.45E -12G O :0045071~n e g a t i v e r e g u l a t i o no f v i r a l g e n o m e r e pl i c a t i o n 912.165.91E -12G O :0006955~i m m u n e r e s p o n s e 1013.518.44E -05细胞成分(C C )G O :0005615~e x t r a c e l l u l a r s p a c e 1621.053.33E -04G O :0005576~e x t r a c e l l u l a r r e g i o n 1722.367.24E -04G O :0005829~c y t o s o l 2431.576.01E -03G O :0005739~m i t o c h o n d r i o n 1317.107.85E -03G O :0005737~c y t o p l a s m 3242.101.24E -02分子功能(M F )G O :0008201~h e p a r i nb i n d i n g 79.098.35E -05G O :0005178~i n t e g r i nb i n d i n g 67.791.10E -04G O :0001730~2'-5'-o l i g o a d e n y l a t e s y n t h e t a s e a c t i v i t y33.891.19E -04G O :0050840~e x t r a c e l l u l a rm a t r i xb i n d i n g45.192.12E -04G O :0005515~p r o t e i nb i n d i n g5267.535.49E -03表5 与c c R C C 舒尼替尼耐药相关的枢纽基因K E G G 分析T a b l e 5 K E G G p a t h w a y a n a l ys i s o f h u b g e n e s a s s o c i a t e dw i t h c c R C Cs u n i t i n i b r e s i s t a n c e 项目数量%P 值h s a 05144:M a l a r i a512.821.38E -04h s a 05164:I n f l u e n z aA 615.382.45E -03h s a 04360:A x o n g u i d a n c e 512.824.95E -03h s a 05162:M e a s l e s512.825.83E -03h s a 04060:C y t o k i n e -c y t o k i n e r e c e p t o r i n t e r a c t i o n 615.388.01E -03h s a 05323:R h e u m a t o i d a r t h r i t i s 410.251.22E -02h s a 05168:H e r p e s s i m pl e x i n f e c t i o n 512.821.74E -02h s a 04668:T N Fs i g n a l i n gp a t h w a y410.252.01E -02h s a 04672:I n t e s t i n a l i m m u n en e t w o r k f o r I gA p r o d u c t i o n 37.702.73E -02h s a 04145:P h a go s o m e 410.255.11E -02㊃641㊃华中科技大学学报(医学版) 2021年4月第50卷第2期表6 模块1K E G G 通路分析T a b l e 6 K E G G p a t h w a y a n a l y s i s o f t o p1m o d u l e 项目P 值h s a 05164:I n f l u e n z aA <0.01h s a 05160:H e p a t i t i sC 0.003h s a 05162:M e a s l e s0.003h s a 05168:H e r p e s s i m pl e x i n f e c t i o n 0.0062.4 基因集富集分析为深入了解G S E 76068样本中导致舒尼替尼耐药的潜在生物学途径和机制,我们进行了G S E A 分析㊂根据截止标准F D R<0.25,P <0.05,显示了12个功能基因组(图3)㊂该结果表明,舒尼替尼耐药组富集在胞质D N A 感知通路㊁T o l l 样受体信号通路㊁R I G -I 受体信号通路㊁干扰素信号通路㊁激活A T R 应对复制应激㊁G 2/M 检查点等㊂P a r e n t a l :舒尼替尼敏感组;S u n i t i n i b -R :舒尼替尼耐药组;以P <0.05且F D R<0.25为显著富集图3 G S E 76068样本中舒尼替尼敏感组和舒尼替尼耐药组之间的基因组富集分析F i g.3G e n e s e t e n r i c h m e n t a n a l y s i s b e t w e e ns u n i t i n i b r e s p o n s e a n d s u n i t i n i b r e s i s t a n c e g r o u p o fG S E 76068s a m p l e s 2.5 K a pl a n -M e i e r 生存分析在K a pl a n -M e i e r 分析的基础上,分析了枢纽基因的表达与c c R C C 患者O S ㊁D F S 之间的关系㊂结果显示,I L 6(H R =2.360,l o g-r a n k P <0.01)㊁I S G 15(H R =1.902,l o g -r a n k P <0.01)㊁O A S L (H R =1.842,l o g-r a n k P <0.01)的高表达预示c c R C C 患者更差的O S ;I L 6(H R =1.387,l o g-r a n k P =0.0463)㊁I S G 15(H R =1.74,l o g -r a n k P =0.0052)㊁O A S L (H R =2.195,l o g -r a n k P =0.0001)的高表达预示c c R C C 患者更差的D F S (图4㊁图5)㊂而I F I T 1的表达和更好的O S (H R =0.5598,l o g-r a n k P =0.0001)和D F S (H R =0.6368,l o g-r a n k P =0.0112)相关㊂患者总生存期信息来自T C G A 数据库;A :I F I T 1;B :I L 6;C :I S G 15;D :O A S L图4 枢纽基因对肾癌患者总生存(O S)的预后价值评估F i g.4P r o g n o s i s a s s e s s m e n t o f o v e r a l l s u r v i v a l b y H u b g e n e s ㊃741㊃刘郴郴等.肾透明细胞癌舒尼替尼耐药的分子标志物筛选及相关生物学功能分析患者无病生存期信息来自T C G A 数据库;A :I F I T 1;B :I L 6;C :I S G 15;D :O A S L图5 枢纽基因对肾癌患者无病生存期(D F S)的预后价值评估F i g.5P r o g n o s i s a s s e s s m e n t o f d i s e a s e f r e e s u r v i v a l b y h u b g e n e 2.6 枢纽基因的受试者工作特征(R O C )曲线分析我们通过R O C 曲线来评估枢纽基因是否具有潜在诊断价值㊂如图6,所示基因可以完全区分c c R C C 和配对的正常组织,曲线下面积(A U C )为0.7362~0.8914㊂此结果表明,这些枢纽基因可能是c c R C C 患者的有效诊断生物标志物㊂2.7 枢纽基因在c c R C C 组织和正常肾组织中的表达利用T C G A 数据库对c c R C C 和正常组织中枢纽基因的表达水平进行评估㊂如图6,与正常组织相比,其中部分基因如I S G 15㊁I F I T 3㊁O A S L 在c c R C C 组织中的表达明显上调㊂基于I S G 15较好的诊断和预后价值,我们进一步研究了它和c c R C C 患者临床病理参数之间的相关性㊂2.8 I S G 15的表达与c c R C C 的各种临床病理参数相关对T C G A 数据库中534例包含较完整信息的c c R C C 样本数据(其中2例缺少T ㊁N 分期,4例缺少M 分期,10例缺少G 分级信息)分析显示,I S G 15m R N A 表达上调与较高的病理分期分级㊁远处转移有显著的相关性(图7),I S G 15的表达水平随着肿瘤病理分期分级升高有增高的趋势㊂患者临床病理资料见表7,依据I S G 15表达水平降序排列,取中位均分为I S G 15高表达和低表达两组㊂I S G 15的高表达与这些临床病理参数之间有显著的相关性,与上述结果一致㊂为了进一步探讨I S G 15的预后价值,我们评估了其表达水平和患者各种临床病理因素与O S 相关性㊂单因素和多因素C o x 比例风险回归分析表明,I S G 15高表达是c c R C C 患者O S 的独立风险因素,可作为独立预后指标(表8)㊂A~C :基因表达水平分析;D ~F :基因表达R O C 曲线分析;患者基因表达水平信息均来自T C G A 数据库;**P <0.01图6 枢纽基因在c c R C C 患者与正常人肾组织表达水平对比及R O C 曲线分析F i g.6E x p r e s s i o n l e v e l a n dR O Cc u r v e a n a l y s i s o f h u b g e n e s i n c c R C C v s .n o r m a l r e n a l t i s s u e s ㊃841㊃华中科技大学学报(医学版) 2021年4月第50卷第2期A :T 分期;B :淋巴结转移;C :远处转移;D :G 分级;**P <0.01图7 I S G 15m R N A 水平与c c R C C 患者多种临床病理参数相关F i g.7T h eh i g hm R N Ae x p r e s s i o no f I S G 15w a s c o r r e l a t e dw i t hv a r i o u s c l i n i c o p a t h o l o g i c a l p a r a m e t e r s 表7 c c R C C 患者I S G 15m R N A 表达水平与临床病理参数的关系T a b l e 7 A s s o c i a t i o nb e t w e e n I S G 15m R N Ae x p r e s s i o na n d c l i n i c o p a t h o l o g i c a l pa r a m e t e r s o f p a t i e n t sw i t h c c R C C 参数例数I S G 15低表达I S G 15高表达P 值A g e (y e a r s )0.293 <60245129116 ȡ60287137150G e n d e r0.523 F e m a l e 1889098M a l e 344176168Ts t a ge <0.01 T 1o rT 2341193148 T 3o rT 419173118Ns t a ge 0.623 N 0o rN X 516259257 N 11679Ms t a g e 0.003 M 0o rM X 451239212 M 1792752G g r a d e0.019G 1o rG 2242134108 G 3o rG 4282127155表8 I S G 15m R N A 表达水平与患者总生存期的单因素和多因素回归分析T a b l e 8 U n i v a r i a t e a n dm u l t i v a r i a t e a n a l y s e s o f I S G 15m R N Ae x pr e s s i o na n d p a t i e n t o v e r a l l s u r v i v a l 变量单因素分析H R95%C I P 值多因素分析H R95%C I P 值A g e (y e a r s ) <60(n =243)1.7861.304~2.445<0.011.51041.098~2.0860.011ȡ60(n =281)G e n d e rF e m a l e (n =183)0.9420.692~1.2820.703 M a l e (n =341)Ts t a ge T 1o rT 2(n =335)3.1032.293~4.201<0.011.5731.097~2.2550.014 T 3o rT 4(n =189)Ns t a ge N 0o rN X (n =508)3.8462.082~7.104<0.012.8321.269~4.4720.007 N 1(n =16)Ms t a g e M 0o rM X (n =446)4.2923.147~5.853<0.012.5141.759~3.593<0.01 M 1(n =78)G g r a d e G 1o rG 2(n =245)2.6161.867~3.666<0.011.6941.178~2.4360.004 G 3o rG 4(n =279)I S G 15L o w (n =262)1.9541.431~2.667<0.011.3961.011~1.9290.043H i gh (n =262)㊃941㊃刘郴郴等.肾透明细胞癌舒尼替尼耐药的分子标志物筛选及相关生物学功能分析3讨论c c R C C是最常见的肾癌亚型,酪氨酸激酶抑制剂(t y r o s i n ek i n a s e i n h i b i t o r,T K I)类靶向药物,如舒尼替尼仍是进展期c c R C C治疗的基石[2,8]㊂事实证明,由于个体差异及肿瘤异质性等因素,不管放化疗㊁靶向治疗还是免疫治疗,总存在着一部分患者先天不敏感或对持续治疗耐受㊂所以,研究c c R C C舒尼替尼耐药的分子标志物和功能途径对c c R C C患者的治疗方式选择及生存预后评估具有重要意义㊂本研究利用多种生物信息学工具发现了一组具有代表意义的分子标志物,有望成为诊断甚至治疗靶点应用于临床,此外我们还分析了其中枢纽基因的诊断和预后价值,并探讨了导致c c R C C舒尼替尼耐药的潜在信号通路㊂3.1D N A感知器-Ⅰ型干扰素通路-I R D S轴可能导致c c R C C获得性耐药到目前为止,已有大量文献报道肾癌舒尼替尼耐药相关机制,包括T K I s被溶酶体螯合㊁缺氧诱导因子(H I F)等关键基因的突变和修饰㊁血管生成替代途径(A K T/m T O R)的激活㊁肿瘤异质性和微环境等,但仍缺乏共识[9]㊂在本研究中,G S E A分析表明D N A损伤反应相关基因集(激活A T R应对复制应激和G2/M检查点)得到显著富集㊂同时,根据G O/K E G G分析,我们发现生物过程(Ⅰ型干扰素信号通路㊁对病毒的防御反应)㊁信号通路(甲型流感㊁麻疹和单纯疱疹病毒感染)和基因集(干扰素信号传导)均得到富集(图2A㊁2B㊁3D),最重要的是,G S E A结果表明胞质D N A感知通路㊁T o l l样受体信号通路㊁R I G-I受体信号通路等经典D N A感应通路均被显著富集㊂据此,我们认为肿瘤细胞通过靶向药物治疗,在复制应激等因素下导致D N A损伤,D N A损伤产物被D N A感应蛋白感知并激活了干扰素信号通路(如c G A S-S T I N G信号通路)㊂在过去的几十年中,研究发现Ⅰ型干扰素(I F N-Ⅰ)来源于各种免疫细胞或肿瘤细胞,作为调节数百种下游细胞因子转录的强大免疫调节因子,参与肿瘤免疫㊁感染调控和组织损伤等过程㊂一般来说,I F N-Ⅰ和Ⅰ型干扰素途径对抑制感染和杀伤肿瘤细胞尤为重要,因此在20多年前,大剂量干扰素被用于治疗转移性肾癌,但疗效不甚理想,而且由于它的副作用使其并没有被广泛应用㊂目前,一些临床前试验已经证明了I F N-Ⅰ定向治疗肿瘤的可取性,但更多的是需要与其他疗法,如靶向治疗㊁放化疗和新型免疫疗法联合使用,才能取得更好的效果㊂然而,近期研究结果表明,Ⅰ型干扰素通路在肿瘤进展和获得性耐药中起着至关重要的作用,尤其是在长期和持续暴露于I F N-Ⅰ的情况下,干扰素相关D N A损伤抗性标志基因(I R D S)明显上调并发挥促癌促耐药作用[10-14]㊂例如J A K抑制剂R u x-o l i t i n i b可以克服非小细胞肺癌对顺铂的耐药性[15]㊂此外,c G A S/R I G-I/T L R4等D N A感应蛋白作为明星分子,在免疫调节途径中对癌症的进展和治疗具有决定性的影响,但与Ⅰ型干扰素信号通路一样, D N A感应蛋白也具有两面性㊂有研究表明,D N A 感应蛋白D D X41是肿瘤启动因子同时也是放疗的不良预后因子[16];另一项研究调查了在基因毒药物作用下,T L R4通路的激活和下游产物的上调保护了残余肿瘤细胞,并促进了肿瘤的转移[17]㊂有趣的是本研究中我们筛选的大部分枢纽基因如I S G15㊁O A S L㊁I F I T3在舒尼替尼耐药样本中上调,均属于I R D S基因,且这个独特的基因子集正是D N A感应和Ⅰ型干扰素信号通路的下游基因㊂综上所述,我们认为在舒尼替尼的作用下,c c R C C中细胞核酸的积累被D N A感应蛋白所感知,并引发了Ⅰ型干扰素通路的激活,继而导致I R D S的上调,促进了肿瘤获得性耐药㊂3.2I R D S是c c R C C获得性耐药的潜在分子标志Ⅰ型干扰素启动了数百个干扰素下游基因的表达,涉及抗病毒㊁抗肿瘤和抗炎功能[18]㊂越来越多的研究表明,在抗病毒反应的第二阶段,在持续低剂量I F Nβ刺激下,未磷酸化的转录因子I S G F3(U-I S G F3)高表达,该转录因子进一步介导一个特殊的干扰素刺激基因子集(I S G s)上调,发挥抵抗D N A 损伤和延长抗病毒效果的作用[7,12,19-22]㊂耐人寻味的是,正是该基因子集中的一部分驱动了放化疗的获得性耐受,并被统称为干扰素相关D N A损伤耐药基因(I R D S),且已经作为预测乳腺癌㊁头颈部肿瘤等恶性肿瘤的放化疗预后因子应用于临床[7,10,12,23]㊂到目前为止,已经发现了49个以上的基因属于I R D S基因㊂从我们富集的生物信息学结果来看, I R D S基因在舒尼替尼耐药组中显著上调㊂如前所述,越来越多的证据表明,细胞D N A损伤产物被c G A S/R I G-I/T L R4等感应蛋白感知,然后激活干扰素通路,促进I R D S的表达[14,24-26]㊂随后,该轴的激活所引发的I R D S上调通过D N A损伤修复和其他分子机制促进了肿瘤的生存[13,27]㊂因此我们有理由相信该过程同时促进了c c R C C的获得性耐药,㊃051㊃华中科技大学学报(医学版)2021年4月第50卷第2期但还需要更多的实验证据㊂我们的研究为I R D S基因I S G15㊁O A S L㊁I F I T3可作为肾癌获得性耐药的有效分子标志物及预后指标提供了线索㊂3.3I S G15是c c R C C潜在的诊断和预后标志物I S G15,即干扰素刺激基因15,1987年作为泛素样蛋白(U b l s)被发现,在各种细胞活动如稳定蛋白㊁调节细胞周期㊁应激反应和信号转导中发挥了重要作用,类似于泛素化,其与蛋白结合的方式被称为I S G y l a t i o n,是未被充分开发的翻译后蛋白修饰途径(P T M s)之一㊂几十年来,对I S G15的研究一直集中在抗病毒复制方面㊂直到最近,研究人员发现I S G15在乳腺癌㊁前列腺癌和胰腺癌等肿瘤发病㊁进展和耐药中发挥了重要作用[7,13,28-30]㊂本研究首次探讨了I S G15在c c R C C中的表达和临床病理参数之间的联系㊂我们发现,与正常肾组织相比,I S G15在c c R C C组织中表达上调,并与c c R C C患者的不良预后相关;多变量回归分析表明,I S G15表达水平是c c R C C的独立预后因素㊂基于此,我们认为I S G15能作为一个潜在的分子标志物用于c c R C C 患者的诊断和预后判断㊂参考文献[1] D iM a r t i n oS,D eL u c aG,G r a s s iL,e ta l.R e n a l c a n c e r:n e wm o d e l s a n d a p p r o a c h f o r p e r s o n a l i z i n g t h e r a p y[J].JE x p C l i nC a n c e rR e s,2018,37(1):217.[2]S i e g e lR L,M i l l e rK D,J e m a lA.C a n c e r s t a t i s t i c s,2019[J].C AC a n c e r JC l i n,2019,69(1):7-34.[3]M o t z e rRJ,B u k o w s k iR M,F i g l i nR A,e t a l.P r o g n o s t i cn o-m o g r a mf o rs u n i t i n i bi n p a t i e n t s w i t h m e t a s t a t i cr e n a lc e l lc a r c i n o m a[J].C a n c e r,2008,113(7):1552-1558.[4]N e r i c hV,H u g u e sM,P a i l l a rMJ,e t a l.C l i n i c a l i m p a c t o f t a r-g e t e d t h e r a p i e s i n p a t i e n t sw i t hm e t a s t a t i c c l e a r-c e l l r e n a l c e l lc a r c i n o m a[J].O n c oT a r g e t sT h e r,2014,7:365-374.[5]M o t z e rRJ,M c c a n nL,D e e nK.P a z o p a n i bv e r s u s s u n i t i n i b i nr e n a l c a n c e r[J].N e wE n g l JM e d,2013,369(20):1968-1970.[6]K a n e h i s a M,G o t oS,H a t t o r iM,e t a l.F r o m g e n o m i c s t oc h-e m i c a l g e n o m i c s:n e w d e v e l o p m e n t si n K E G G[J].N u c l e i cA c i d sR e s,2006,34(D a t a b a s e i s s u e):D354-D357.[7]W e i c h s e l b a u mRR,I s h w a r a nH,Y o o nT,e t a l.A n i n t e r f e r o n-r e l a t e d g e n e s i g n a t u r e f o rD N Ad a m a g e r e s i s t a n c e i s a p r e d i c-t i v em a r k e r f o r c h e m o t h e r a p y a n dr a d i a t i o n f o rb r e a s t c a n c e r[J].P N A S,2008,105(47):18490-18495.[8]A t k i n sM B,T a n n i rN M.C u r r e n t a n d e m e r g i n g t h e r a p i e s f o rf i r s t-l i n e t r e a t m e n t o fm e t a s t a t i c c l e a r c e l l r e n a l c e l l c a r c i n o m a[J].C a n c e rT r e a tR e v,2018,70:127-137.[9]M a k h o vP,J o s h i S,G h a t a l i aP,e ta l.R e s i s t a n c et os y s t e m i ct h e r a p i e s i nc l e a rc e l lr e n a lc e l lc a r c i n o m a:m e c h a n i s m sa n dm a n a g e m e n t s t r a t e g i e s[J].M o lC a n c e rT h e r,2018,17(7):1355-1364.[10] K h o d a r e vN N,B e c k e t tM,L a b a y E,e t a l.S T A T1i so v e r e x-p r e s s e d i n t u m o r s s e l e c t e d f o r r a d i o r e s i s t a n c e a n d c o n f e r s p r o-t e c t i o n f r o mr a d i a t i o n i n t r a n s d u c e d s e n s i t i v e c e l l s[J].P N A S,2004,101(6):1714-1719.[11] K h o d a r v eNN,M i n nAJ,E f i m o v aEV,e t a l.S i g n a l t r a n s d u c-e r a n d a c t i v a t o r of t r a n s c r i p t i o n1r eg u l a t e s b o th c y t o t o xi c a n dp r o s u r v i v a l f u n c t i o n s i n t u m o r c e l l s[J].C a n c e rR e s,2007,67(19):9214-9220.[12] D u a r t eC W,W i l l e y CD,Z h i D,e t a l.E x p r e s s i o ns i g n a t u r eo fI F N/S T A T1s i g n a l i n gg e n e s p r e d i c t s p o o rs u r v i v a lo u t c o m ei n g l i o b l a s t o m am u l t i f o r m e i nas u b t y p e-s p e c i f i cm a n n e r[J].P L o SO n e,2012,7(1):e29653.[13] B o e l e n s M C,W u TJ,N a b e tB Y,e ta l.E x o s o m et r a n s f e rf r o ms t r o m a l t ob r e a s t c a n c e rc e l l sr eg u l a t e s th e r a p y r e si s t-a n c e p a t h w a y s[J].C e l l,2014,159(3):499-513.[14] B u d h w a n iM,M a z z i e r i R,D o l c e t t i R.P l a s t i c i t y o f t y p e I i n t e r-f e r o n-m e d i a t e dr e s p o n s e s i nc a n c e rt h e r a p y:f r o m a n t i-t u m o ri m m u n i t y t o r e s i s t a n c e[J].F r o n tO n c o l,2018,8:322.[15]H u Y,H o n g Y,X u Y,e ta l.I n h i b i t i o no f t h eJ A K/S T A Tp a t h w a y w i t h r u x o l i t i n i b o v e r c o m e sc i s p l a t i n r e s i s t a n c ei nn o n-s m a l l-c e l l l u n g c a n c e r N S C L C[J].A p o p t o s i s,2014,19(11):1627-1636.[16] L a e n g l e J,S t i f t J,B i l e c zA,e t a l.D N Ad a m a g e p r e d i c t s p r o g-n o s i s a n d t r e a t m e n t r e s p o n s e i n c o l o r e c t a l l i v e rm e t a s t a s e s s u-p e r i o r t o i m m u n o g e n i cc e l ld e a t ha n d Tc e l l s[J].T h e r a n o s-t i c s,2018,8(12):3198-3213.[17] R a nS.T h e r o l eo fT L R4i nc h e m o t h e r a p y-d r i v e n m e t a s t a s i s[J].C a n c e rR e s,2015,75(12):2405-2410.[18] C h e o n H,H o l v e y-b a t e sE G,S c h o g g i n sJ W,e ta l.I F Nβ-d e-p e n d e n t i n c r e a s e s i nS T A T1,S T A T2,a n dI R F9m e d i a t er e-s i s t a n c e t ov i r u s e sa n d D N A d a m a g e[J].E M B OJ,2013,32(20):2751-2763.[19] W a n g W,Y i nY,X uL,e t a l.U n p h o s p h o r y l a t e d I S G F3d r i v e sc o n s t i t u t i v e e x p r e s s i o no f i n t e r f e r o n-s t i m u l a t ed ge n e s t o p r o-t e c t a g a i n s tv i r a l i n f e c t i o n s[J].S c iS i g n a l,2017,10(476):e a a h4248.[20] C h e o nH,H o l v e y-b a t e sEG,S t a r kGR.A b s t r a c t485:C h r o n-i c e x p o s u r et oal o w d o s eo f i n t e r f e r o n-βi n c r e a s e s l e v e l so fD N Ad a m a g e r e s i s t a n c e s i g n a t u r e g e n e s t h r o u g hu n p h o s p h o-r y l a t e d I S G F3[J].C a n c e rR e s,2014,74(19S u p p l):485.[21] C h e o nH,S t a r kG R.U n p h o s p h o r y l a t e dS T A T1p r o l o n g s t h ee x p r e s s i o n o fi n t e rf e r o n-i n d u c e di m m u n e r eg u l a t o r y g e n e s[J].P N A S,2009,106(23):9373-9378.[22] K h o d a r e vN N,R o i z m a nB,W e i c h s e l b a u m RR.M o l e c u l a r p a-t h w a y s:i n t e r f e r o n/s t a t1p a t h w a y:r o l e i n t h e t u m o r r e s i s t a n c e t o g e n o t o x i cs t r e s sa n da g g r e s s i v e g r o w t h[J].C l i n C a n c e rR e s,2012,18(11):3015-3021.[23] C h e o n H,Y a n g J,S t a r kG R.T h e f u n c t i o n so f s i g n a l t r a n s-d u ce r s a n d a c t i v a t o r s of t r a n s c r i p t i o n s1a n d3a s c y t o k i n e-i n-d u c i b le p r o t e i n s[J].J I n t e r fC y t o kR e s,2011,31(1):33-40.[24] L i u H,G o l j i J,B r o d e u rL K,e ta l.T u m o r-d e r i v e dI F Nt r i g-g e r sc h r o n i c p a t h w a y a g o n i s ma n ds e n s i t i v i t y t oA D A Rl o s s[J].N a tM e d,2018,25(1):95-102.[25] B h a t N,F i t z g e r a l d K A.R e c o g n i t i o no fc y t o s o l i c D N A b yc G A Sa n do t h e r S T I N G-de p e n d e n t s e n s o r s[J].E u r J I m m u-n o l,2014,44(3):634-640.[26] B r o n t eV.T u m o r sS T I N Ga d a p t i v ea n t i t u m o r i m m u n i t y[J].I m m u n i t y,2014,41(5):679-681.[27]S u nN K,H u a n g SL,C h a n g TC,e t a l.T L R4a n dN F k a p p a Bs i g n a l i n g i sc r i t i c a l f o rt a x o l r e s i s t a n c e i no v a r i a nc a r c i n o m ac e l l s[J].JC e l l P h y s i o l,2018,233(3):2489-2501.[28] D e s a i SD,R e e dRE,B u r k s J,e t a l.I S G15d i s r u p t s c y t o s k e l e-t a l a r c h i t e c t u r e a n d p r o m o t e sm o t i l i t y i nh u m a nb r e a s t c a n c e rc e l l s[J].E x p B i o lM e d(M a y w o o d),2012,237(1):38-49.[29] K i e s s l i n g A,H o g r e f eC,E r bS,e ta l.E x p r e s s i o n,r e g u l a t i o na n d f u n c t i o no f t h e I S G y l a t i o ns y s t e mi n p r o s t a t e c a n c e r[J].O n c o g e n e,2009,28(28):2606-2620.[30]A g a r w a lS,M c g o w e n K,J a n s s o n K,e ta l.A b s t r a c t2896:S T A T1d e p e n d e n t i n t e r f e r o n-r e l a t e dD N Ad a m a g e r e s i s t a n c es i g n a t u r e(I R D S)a s a s u r v i v a lm e c h a n i s mi nc a s t r a t e r e s i s t a n tp r o s t a t e c a n c e r(C R P C)[J].C a n c e rR e s,2017,77(13S u p p l): 2896.(2020-10-27收稿)㊃151㊃刘郴郴等.肾透明细胞癌舒尼替尼耐药的分子标志物筛选及相关生物学功能分析。
基于网络药理学探讨三子养亲汤治疗慢性阻塞性肺疾病的作用机制

基于网络药理学探讨三子养亲汤治疗慢性阻塞性肺疾病的作用机制作者:马馨蕊吕晓东庞立建刘勇明梁元钰来源:《世界中医药》2022年第11期摘要目的:運用网络药理学的研究方法探讨三子养亲汤治疗慢性阻塞性肺疾病(COPD)的作用机制。
方法:利用中药系统药理学数据库与分析平台(TCMSP)对三子养亲汤中药物的化学成分、作用靶标进行检索与筛选;利用GeneCards数据库对COPD的相关靶标进行检索与筛选;取药物与疾病的交集靶点,将交集靶点输入到String数据库进行分析,构建多层次网络和蛋白质-蛋白质相互作用(PPI)网络,并对其进行基因本体(GO)富集分析和京都基因和基因组百科全书(KEGG)富集分析。
结果:筛选后得出22个三子养亲汤有效成分和231个作用靶点。
COPD相关基因中Relevance score大于3的基因有5 737个,药物与疾病的共有靶点有115个。
PPI网络分析得出,三子养亲汤治疗COPD与AKT1、MAPK1、IL6、JUN、CASP3等基因有关。
GO功能富集分析和KEGG通路富集分析得到GO条目170个、KEGG通路162条。
KEGG通路富集分析得出三子养亲汤治疗COPD主要通过包括Lipid and atherosclerosis和PI3K-AKT signaling pathway等通路进行调控。
结论:本研究从网络药理学的方向,系统地阐述了三子养亲汤通过药物的多成分、多靶点特点对慢性阻塞性肺疾病产生作用机制,为三子养亲汤治疗慢性阻塞性肺疾病的进一步研究提供了基础。
关键词三子养亲汤;慢性阻塞性肺疾病;网络药理学;蛋白质-蛋白质相互作用网络;基因本体功能富集分析;京都基因和基因组百科全书通路富集分析Mechanism of Sanzi Yangqin Decoction in Treatment of Chronic Obstructive Pulmonary Disease Based on Network PharmacologyMA Xinrui1,LYU Xiaodong1,PANG Lijian2,LIU Yongming1,LANG Yuanyu1(1 Liaoning University of Traditional Chinese Medicine,Shenyang 110847,China; 2 The Affiliated Hospital of Liaoning University of Traditional Chinese Medicine,Shenyang 110032,China)Abstract Objective:To explore the mechanism of Sanzi Yangqin Decoction in the treatment of chronic obstructive pulmonary disease(COPD) based on network pharmacology.Methods:The chemical components and targets of the herbal medicines in the Sanzi Yingqin Decoction were searched via Traditional Chinese Medicine Systems Pharmacology Database and Analysis Platform (TCMSP).GeneCards was searched for the targets of COPD.The common targets shared by the herbal medicines and COPD were identified and input to String for the construction of a multi-level network and a protein-protein interaction(PPI) network.Gene ontology(GO) annotation and Kyoto Encyclopedia of Genes and Genomes(KEGG) pathway enrichment were carried out for the common targets.Results:After screening,22 effective components and 231 targets of Sanzi Yangqin Decoction were obtained.There were 5 737 COPD-related genes with relevance score greater than 3.The decoction and COPD shared 115 common targets.The PPI network revealed that the treatment of COPD by Sanzi Yangqin Decoction was associated with AKT1,MAPK1,IL6,JUN,CASP3,and other genes.GO annotation and KEGG pathway enrichment predicted 170 GO terms and 162 KEGG pathways,which indicated that Sanzi Yangqin Decoction treated COPD mainly through lipid and atherosclerosis signaling pathway and PI3K-Akt signalingpathway.Conclusion:Sanzi Yangqin Decoction treats COPD through multiple components and targets.This study provides a basis for further research on the treatment of COPD by Sanzi Yangqin Decoction.Keywords Sanzi Yangqin Decoction; Chronic obstructive pulmonary disease; Network pharmacology; Protein-protein interaction(PPI) network; Gene ontology(GO) annotation; Kyoto Encyclopedia of Genes and Genomes(KEGG) pathway enrichment中图分类号:R285;R289.5文献标识码:Adoi:10.3969/j.issn.1673-7202.2022.17.012慢性阻塞性肺疾病(Chronic Obstructive Pulmonary Disease,COPD)是一种常见,可预防和可治疗的疾病,其特征在于持续的呼吸道症状和气流受限,这是由于气道和(或)肺泡异常所致,通常是由于长期暴露于有害颗粒或气体所引起,最常见的呼吸系统症状包括呼吸困难、咳嗽和(或)咳痰[1]。
单细胞转录组鉴定炎症细胞因子

单细胞转录组鉴定炎症细胞因子英文回答:Single-cell transcriptomics has revolutionized our understanding of cellular heterogeneity and has provided valuable insights into various biological processes, including inflammation. In order to identify inflammatory cytokines at the single-cell level, a combination of experimental and computational approaches can be employed.Experimental approaches involve isolating and sequencing the transcriptomes of individual cells. This can be done using methods such as single-cell RNA sequencing (scRNA-seq), which allows for the measurement of gene expression in individual cells. By profiling the transcriptomes of cells before and after exposure to an inflammatory stimulus, differentially expressed genes can be identified. These genes are likely to be involved in the production of inflammatory cytokines.Once the differentially expressed genes are identified, computational methods can be used to further analyze the data and identify the specific inflammatory cytokines produced by different cell types. This can involve clustering analysis to group cells with similar gene expression patterns, as well as gene set enrichment analysis to identify enriched pathways or functions. By comparing the gene expression profiles of different cell clusters, it is possible to infer the cytokines produced by each cluster.For example, let's say we are interested in identifying the inflammatory cytokines produced by macrophages in response to lipopolysaccharide (LPS) stimulation. Weisolate and sequence the transcriptomes of individual macrophages before and after LPS treatment. We identify a set of differentially expressed genes that are upregulated in response to LPS. Using computational methods, we cluster the macrophages based on their gene expression profiles and perform gene set enrichment analysis. We find that acluster of macrophages highly expresses genes associated with the production of pro-inflammatory cytokines such astumor necrosis factor alpha (TNF-α) and interleukin-1 beta (IL-1β). This suggests that t hese macrophages are likelyto be responsible for the production of these cytokines in response to LPS stimulation.中文回答:单细胞转录组学已经彻底改变了我们对细胞异质性的理解,并为各种生物学过程提供了宝贵的见解,包括炎症反应。
基因通路富集分析方法大总结
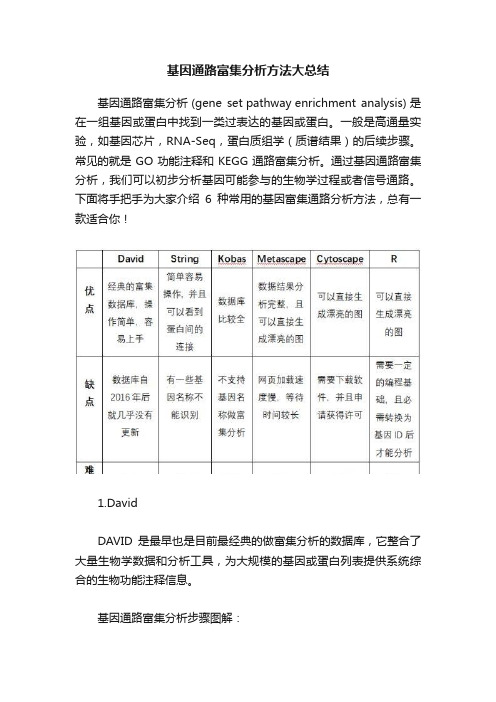
基因通路富集分析方法大总结基因通路富集分析 (gene set pathway enrichment analysis) 是在一组基因或蛋白中找到一类过表达的基因或蛋白。
一般是高通量实验,如基因芯片,RNA-Seq,蛋白质组学(质谱结果)的后续步骤。
常见的就是GO功能注释和KEGG通路富集分析。
通过基因通路富集分析,我们可以初步分析基因可能参与的生物学过程或者信号通路。
下面将手把手为大家介绍6种常用的基因富集通路分析方法,总有一款适合你!1.DavidDAVID是最早也是目前最经典的做富集分析的数据库,它整合了大量生物学数据和分析工具,为大规模的基因或蛋白列表提供系统综合的生物功能注释信息。
基因通路富集分析步骤图解:第一步:打开网址,点击Functional Annotation。
第二步:输入基因集,选择输入类型第三步:选择物种,查看结果(包括GO和KEGG通路结果)2.StringString数据库是瑞士苏黎世大学构建的一个搜寻蛋白质之间相互作用的数据库。
既包括蛋白质之间的直接物理相互作用,也包括蛋白质之间的间接功能相关性。
它除了包含有实验数据、从PubMed摘要中挖掘的结果和综合其他数据库数据外,还有利用生物信息学的方法预测的结果。
基因通路富集分析步骤图解:第一步:打开网站,输入基因列表和选择物种;第二步:选择数据库内对应基因名称;第三步:结果下载-包括Go和KEGG通路。
(如有需要还可以下载蛋白连接的结果)4.KobasKobas是北京大学开发的用于注释和鉴定富集途径和疾病的数据库基因通路富集分析步骤图解:第一步:打开网站,选择Gene-list Enrichment第二步:选择输入类型,物种,输入基因列表,选择数据库,后可分析下载数据。
5.MetascapeMetascape是近年来新兴的富集分析数据,数据不仅更新快,其覆盖面也相当广泛。
从数据库种类来说,Metascape整合了GO、KEGG、UniProt和DrugBank等多个权威的数据资源,使其不仅能完成通路富集和生物过程注释,还能做基因相关的蛋白质网络分析和涉及到的药物分析,致力于为科研工作者提供每个基因全面而详细的信息。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Random samples of array genes
Module 2
bioinformatics.ca
DAVID demo
/tools.jsp
Module 2
bioinformatics.ca
Step 1: Define your gene list
1. 2. Gene list: e.g. RRP6, MRD1, RRP7, RRP43, RRP42 (yeast) Gene sets or annotations: e.g. Gene ontology, transcription factor binding sites in promoter
Enrichment Test
Enrichment Table
Spindle Apoptosis 0.00001 0.00025
ENRICHMENT TEST
Gene-set Databases
Module 2
bioinformatics.ca
Enrichment Analysis
• Given:
Example list is from the Human U95A array, select it here.
Module 2
bioinformatics.ca
Step 3: Check list & background
Module 2
bioinformatics.ca
Step 4: Run Enrichment Analysis
Canadian Bioinformatics Workshops
www.bioinformatics.ca
Module #: Title of Module
2
Module 2 Finding over-represented pathways in gene lists
Quaid Morris
Module 2
bioinformatics.ca
Step 8a: What can happen if no right-click
Module 2
bioinformatics.ca
Step 8b: How it looks in a spreadsheet
Module 2
bioinformatics.ca
http://morrislab.med.utoronto.ca
Outline
• Introduction to enrichment analysis • Demo of DAVID, a web tool for doing enrichment analysis • Theory of enrichment analysis:
– Fisher’s Exact Test – Multiple test corrections – Other enrichment tests (GSEA)
• DAVID Lab
Module 2
bioinformatics.ca
Microarray Experiment (gene expression table)
• Either
– (a) Copy and paste your list in – (b) Upload a gene list file – (c) Choose an example gene list (so, click “demolist1” on next slide)
Module 2
Module 2
bioinformatics.ca
Enrichment Test
The output of an enrichment test is a P-value
The P-value assesses the probability that the overlap is at least as large as observed by random sampling the array genes.
• •
Question: Are any of the gene annotations surprisingly enriched in the gene list? Details:
– – – Where do the gene lists come from? How to assess “surprisingly” (statistics) How to correct for repeating the tests
6 Module 2
bioinformatics.ca
Two-class Design
Expression Matrix
Genes Ranked by Differential Statistic UP
Selection by Threshold
UP
DOWN Class-1 Class-2
DOWN
t t t … 1 2 3
t n
Module 2
bioinformatics.ca
Enrichment Test
Microarray Experiment (gene expression table)
Gene list (e.g UP-regulated)
Gene-set Databases
Background (all genes on the array)
a.k.a., the hypergeometric test
Gene list
Null hypothesis: List is a random sample from population Alternative hypothesis: More black genes than expected
E.g.: - Fold change - Log (ratio) - t-test -Significance analysis of microarrays
Module 2
bioinformatics.ca
Time-course Gene Design
Expression Matrix
E.g.: - K-means Clusters - K-medoids - SOM
Module 2
bioinformatics.ca
Step 6: View enrichment
Gene set name
EASE P-value 4.0E-4 means: 4.0 x 10-4
# of genes in set with annotation
Module 2
bioinformatics.ca
Module 2
bioinformatics.ca
Enrichment Test
Microarray Experiment (gene expression table)
Gene list (e.g UP-regulated)
Gene-set
Gene-set Databases
Background (all genes on the array)
Gene list
Null distribution
RRP6 MRD1 RRP7 RRP43 RRP42
P-value Answer = 4.6 x 10-4
Background population: 500 black genes, 4500 red genes
26 Module 2
bioinformatics.ca
RRP6 MRD1 RRP7 RRP43 RRP42
Background population: 500 black genes, 4500 red genes
25 Module 2
bioinformatics.ca
Fisher’s exact test
a.k.a., the hypergeometric test
bioinformatics.ca
Step 8: Download results
Download spreadsheet (in tabdelimited text format). Beware: if you click you may get text in a browser window, if this happens, “right-click” to save as a file.
– Bonferroni – Benjamini-Hochberg FDR
• Other enrichment tests
– GSEA for ranked lists (with a short intro)
Module 2
bioinformatics.ca
Fisher’s exact test
Module 2
bioinformatics.ca
Step 2: Choose background
You can either upload a background list (in the upload tab, see previous step) or choose one of the background sets (shown here)
Step 7: Change parameters if desired
Set “count” to 1 and “EASE” to 1 if you want maximum # of categories.
Beware, only corrects within category.
Module 2
Background population: 500 black genes, 4500 red genes
