序列拼接工具使用指南
利用SeqMan进行序列拼接

利用SeqMan进行序列拼接利用SeqMan进行序列拼接Step1:打开Seqman软件Step2:加入你要拼接的序列点击Add sequences查找并选中要拼接的序列(可按住control键进行多选)点击Add按钮填加选择的序列填加完后点击done注:最好用测序的图谱尽量不要直接用测序得到的序列Step3:去除末端序列主要是去除序列末端测序质量差或是载体序列有两种方法可以用来去除这类末端序列其一:利用Seqman自带的去除工具自动去除(利用Trim ends 按钮进行)其二:手工去除个人感觉手工去除方法最有效,因此下边我们以后工去除为例进行演示手工去除侧翼序列双击要去除侧翼序列的目标序列将鼠标放到测序图谱左边的一个黑色的竖线上,此时鼠标会变成一个有两个箭头的水平线按住左键拖动黑竖线,那么你就会发现侧翼序列的颜色变浅,这部分变浅的序列则就被去除,不再参加后面的拼接此步请将测序不准确或认为是载体的序列用这种方法去除。
测序准确的峰形图峰形规则,一般在序列的中部,如下图所示测序不准确的峰形图峰形较乱,很难判断是哪个碱基,一般位于序列两端,如下图所示Step4:进行序列拼接点击Assemble按钮在新出现窗口处点击拼接好的contig1在出现的Alignment of contig1 窗口中点击左三角显示序列的测序图谱点击菜单contig->strategy view可以观察序列拼接的宏观图Step5:查找拼接错误find conflict 点击菜单Edit点击Find Previous或Find Next查找接接中出现的错误还可以通过Seqman左下角的快捷按钮查找错误的拼接查找错误的拼接错误的拼接的类型类型1:两条序列的测序结果不一致并明显一条测序质量好而另一条质量差处理:直接将该处修改为正确的碱基类型2:两条序列的测序结果不一致并两条测序质量都比较差处理:重新测序或用新的合适引物重新测定类型3:两条序列的测序结果不一致并明显两条测序质量都好处理:测序过程出现问题,重新测定Step6:导出拼接的序列可选择合适的格式,导出拼接好的序列通过以上几步我们就能很快将几个测序片段进行拼接,大家可以拿着自己的序列试试!当然如果两个测序片段的拼接片段太短可能利用默认的参数不能完成拼接,大家可以试着修改一下拼接参数试试!如降低Match size及Minimum Match Percentage的值!修改参数命令。
高级剪辑技巧 在Adobe Premiere Pro中合并多个序列

高级剪辑技巧:在Adobe Premiere Pro中合并多个序列在视频编辑领域中,Adobe Premiere Pro被广泛使用,它提供了许多高级剪辑技巧,帮助用户更好地完成他们的项目。
在本教程中,我们将重点介绍如何在Adobe Premiere Pro中合并多个序列,以提高工作效率和视频质量。
首先,打开Adobe Premiere Pro软件并加载您的项目。
确保您已经创建了多个序列,每个序列都有不同的内容或场景。
您可以通过单击菜单栏中的“文件”按钮,然后选择“新建序列”来创建新的序列。
您还可以使用键盘快捷键Ctrl + N来快速创建一个新的序列。
接下来,选择您想要合并的第一个序列。
在项目窗口中选中该序列,并将其拖放到时间轴窗口中。
确保它与您的当前序列对齐。
然后,选择您要合并的第二个序列并重复上述操作。
将其拖放到时间轴窗口中,并确保它与第一个序列紧密对齐。
现在,您将在时间轴窗口中看到两个序列并排放置。
为了将它们合并成一个序列,您可以使用一个简单但强大的功能 - 剪辑合并工具。
这个工具可以帮助您将两个或多个序列合并成一个更大的序列,而不会改变原始序列的内容。
要使用剪辑合并工具,首先选择您要合并的第一个序列。
然后,在工具栏中选择剪辑合并工具(通常显示为两个Overlapping箭头)。
将剪辑合并工具拖放到第一个序列的末尾,并释放鼠标。
现在,您将看到一个新的合并序列出现在时间轴窗口中。
继续这个过程,选择您要合并的第二个序列,将剪辑合并工具拖到它的末尾并释放鼠标。
再次,您将看到一个新的合并序列在时间轴窗口中出现在前一个合并序列的末尾。
重复这个步骤,直到您将所有需要合并的序列都添加到时间轴窗口中。
最后,您将获得一个包含所有序列内容的大型合并序列。
通过合并序列,您可以将多个独立的序列组合成一个更大的序列,并且可以更轻松地在不同场景之间进行切换和编辑。
这对于制作长篇视频或复杂的项目非常有用。
另一个有用的技巧是在合并序列后,您可以对它们进行重新排序和调整。
基因序列拼接器软件 使用说明书(软件操作文档)

基因序列拼接器软件使用说明书(软件操作文档)1. 引言基因序列拼接器软件(MergeSeq)基于研究者测序获得或从核酸数5据库(如GenBank等)中批量下载的fasta格式存储的基因片段,按照使用者指定顺序,将同一物种不同基因片段拼接成由多基因组成的长序列,用于不同物种间的分子系统发育分析。
1.1编写目的本说明书为在Linux/UNIX环境下使用MergeSeq软件的用户编写。
10它将指引使用者按步骤搭建该软件的运行环境,明确输入文件的录入格式,熟悉运行参数的配置规则,理解软件运行时出现的状态信息并掌握获取输出fasta格式文件的方法。
1.2项目背景非模式生物(如蜘蛛等)构建分子系统发育树一般选取低速进化15(Slow-Evolving)且有种属特异性的基因片段进行分析。
为提高结果的置信度,一般采取多基因组合分析的方法(Dimitrov et al., 2016; Wheeler et al., 2016)。
但在实际操作中,将同一物种不同基因片段拼接在一起是一件十分费事且容易出错的重复性操作。
尤其当某一物种某个基因数据缺失时,20必须在拼接后的长序列中填充与同一基因其他对齐序列等长的占位符以保证结果为对齐。
本项目基于测序或下载获得的fasta格式基因序列片段,按照研究者指定的基因排列顺序,自动将同一物种不同基因片段拼接成多基因长序列,并保证结果对齐,可直接用于不同物种间的分子系统发育分析。
251.3 定义(专门术语的定义和缩写词的原意)fasta格式(fasta format):fasta格式是一种基于文本用于表示核酸序列或多肽序列的格式。
其中核酸或氨基酸均以单个字母来表示,且允许在序列前添加序列名及注释。
该格式已成为生物信息学领域常用的30标准文件格式。
2. 软件性能2.1.数据精确度本软件不涉及数字的计算和处理,输入、输出数据的格式均为35UTF-8编码的文本类型文件。
2.2.时间特性本软件对输入数据、输出数据的处理时间由基因片段长度和运行软件主机性能决定。
DNA star Seqman 使用说明 DNA序列拼接
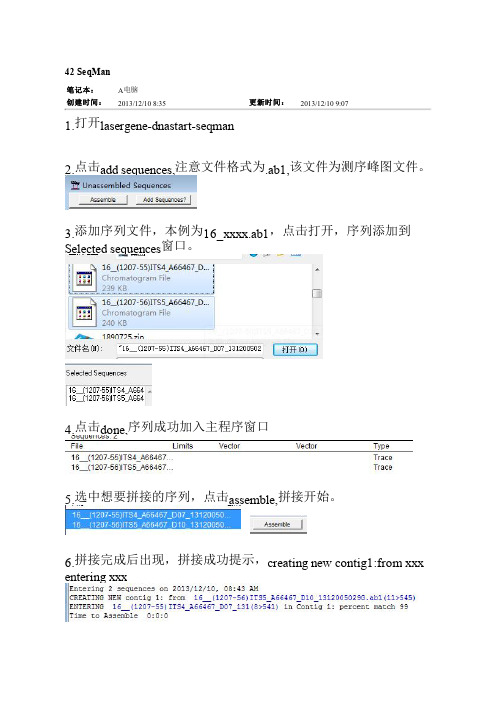
42 SeqMan笔记本:A电脑创建时间:2013/12/10 8:35更新时间:2013/12/10 9:071.打开lasergene-dnastart-seqman2.点击add sequences,注意文件格式为.ab1,该文件为测序峰图文件。
3.添加序列文件,本例为16_xxxx.ab1,点击打开,序列添加到Selected sequences窗口。
4.点击done,序列成功加入主程序窗口5.选中想要拼接的序列,点击assemble,拼接开始。
6.拼接完成后出现,拼接成功提示,creating new contig1:from xxx entering xxx7.点击窗口右上角,“-”最小化,将拼接提示最小化,回到主窗口。
8. 此时主窗口上方出现拼接好的contig1的信息,574bp,来源于两条序列。
9.双击contig1出现具体的拼接过程窗口。
10.点击16前的黑色三角符号,可以看到序列峰图(注意峰图非常重要,不同颜色代表不同碱基,峰型表示测序可信度)。
11.详细讲一下峰图:测序反应开始时和结束时的序列是读不准的(测序的原理决定)。
一个测序反应最多能测定500-800个碱基,且测序反应开始和结束的碱基读不准。
ITS45的长度在500bp左右,意味着单向测序末端会读不准。
采用双向测序,在R向峰分辨率极度降低时,F向正好处在分辨率最高的测序区域,所以这段序列程序会以F向测序结果为准。
seqman在序列拼接的同时,让测序峰图可见,让我们可以判断测序结果的可靠性。
12.接着说拼接完成后如何拷贝拼接好的序列,其实非常简单,选中顶上的consensus中的序列,全选,ctrl+C,拼接好的序列就复制到剪切板中了,可以粘贴到txt中使用。
seqman使用说明

修改参数命令
关于Seqman的简单介绍就到这里,另外由 于资料准备仓促,如有不完善或不准确的 地方,请大家指出,谢谢! 下期讲座预告 “轻松学用生物软件(3)”,具 体时间及内容将在螺旋课堂上做出预告, 欢迎大家参加!
手工去除侧翼序列
测序准确的峰形图
峰形规则,一般在序列的中部,如下图所示
测序不准确的峰形图
峰形较乱,很难判断是哪个碱基,一般位于序列两端, 如下图所示
轻松学用生物软件(2) 利用SeqMan进行序列拼接
主讲人:huaxing 2008年4月11日
螺旋课堂系列讲座
主要内容
大家好,首先感谢大家关注、支持螺旋网!今天 我们一起一步一步的学习利用Dnastar 6.0中的子 程序Seqman进行序列拼接,希望能对大家有所帮 助。 注意:Dnastar 6.0的安装程序及破解补丁大家可 以到螺旋网的“生物信息学及生物软件交流”版块 免费下载!
手工去除侧翼序列
1. 双击要去除侧翼序列的目标序列 2. 将鼠标放到测序图谱左边的一个黑色的竖线上,此时鼠标 会变成一个有两个箭头的水平线 3. 按住左键拖动黑竖线,那么你就会发现侧翼序列的颜色变 浅,这部分变浅的序列则就被去除,不再参加后面的拼接 4. 此步请将测序不准确或认为是载体的序列用这种方法去 除。
错误的拼接的类型
1. 两条序列的测序结果不一致并明显一条测序质量好而另 一条质量差 处理:直接将该处修改为正确的碱基 2. 3. 两条序列的测序结果不一致并两条测序质量都比较差 处理:重新测序或用新的合适引物重新测定 两条序列的测序结果不一致并明显两条测序质量都好 处理:测序过程出现问题,重新测定
利用Final Cut Pro实现视频序列镜头拼接

利用Final Cut Pro实现视频序列镜头拼接Final Cut Pro是一款功能强大的视频编辑软件,能够帮助用户实现视频序列镜头的拼接。
在本文中,我们将介绍一些使用Final Cut Pro进行视频序列镜头拼接的技巧和步骤。
首先,打开Final Cut Pro软件,并导入你想要拼接的视频序列镜头文件。
你可以通过点击菜单栏中的“文件”选项,然后选择“导入”来导入视频文件。
或者,你也可以直接拖拽文件到软件的项目库面板中。
当视频序列镜头文件成功导入后,你需要将这些镜头按照你想要的顺序进行排列。
你可以在项目库面板中选择一个镜头文件,然后拖动到时间轴面板中的合适位置。
依此类推,再将其他镜头文件按照顺序拖动到时间轴面板中。
在拖拽镜头文件到时间轴面板时,你可以通过放大或缩小时间轴面板来调整视频序列的时间轴。
这样做可以更精确地控制每个镜头的长度和位置。
如果你想要调整视频序列中单个镜头的持续时间,你可以将鼠标悬停在时间轴上的该镜头位置,然后用鼠标拖动调整该镜头的起始和结束点。
这样,你可以灵活地控制每个镜头的播放时间。
此外,Final Cut Pro还提供了一些过渡效果选项,可以让你在不同镜头之间添加流畅的过渡。
在时间轴面板中,你可以选择两个相邻的镜头,并使用菜单栏中的“编辑”选项来选择合适的过渡效果。
只需简单点击一下,过渡效果就会被添加到两个镜头之间。
当你完成了视频序列镜头的拼接后,你可以在导出之前对视频进行一些调整和编辑。
Final Cut Pro提供了许多强大的编辑工具,例如剪切、修剪、添加文本等,可以让你对视频进行更多个性化的处理。
最后,完成编辑后,你可以通过点击菜单栏中的“文件”选项,然后选择“导出”来导出你的视频。
Final Cut Pro为用户提供了多种导出选项,你可以根据需要选择合适的导出格式和参数。
综上所述,Final Cut Pro是一款功能强大的视频编辑软件,能够帮助用户实现视频序列镜头的拼接。
PCR序列拼接-CExpress使用说明

PCR序列拼接软件Contig Express使用简介由于一般测序方法的限制,测续结果一般只能到800bp左右,所以对于较长片段只能用不同引物测序后根据结果拼接,ContigExpress正式是一款非常实用的序列拼接软件。
Contig Express是著名的软件Vector NTI的组件之一。
经过一些简单的处理该组件可以被剥离出来独立运行,经我们试用效果良好。
Contig Express将每个PCR测序片断视为一个Contig,当您输入多个Contig后,它会自动寻找其中的公共序列,然后将他们拼接好后的结果已图形方式呈现给您。
完全免除您手工逐个片断Blast,然后肉眼找共同序列的苦恼。
Contig Express 9.1可以从论坛中下载。
2 - 向Contig Express中加入需要拼接的序列目前PCR测序的结果多以ABI和Fasta两种格式提供。
一般来说ABI结果是原始结果最为可信,因此我们最好向Contig Express中直接倒入ABI格式的原始数据。
倒入的方法是"Project---Add Fragments---From ABI file..."。
在接下来弹出的"Import Sequence From"对话框中选择测序公司发给您的ABI文件。
需要注意的是目前公司发给您的原始ABI文件多是"AB1"文件名。
Contig Express无法直接识别,因此有必要在文件类型中选择“All Files(*.*)"。
由于要拼接多个序列这时您可以配合Ctrl键进行多选。
3 - 用Contig Express进行序列拼接现在我们已经选好了要进行拼接的序列了,接下来的步骤就交给Contig Express让它为我们自动拼接了。
用鼠标配合Ctrl键选择要进行拼接的序列。
然后点击"Assemble---Assemble Selected Fragments"。
生物学软件seqman序列拼接步骤

测序后的序列为两种形式:abi,seq
abi:波峰图seq:atcg序列
seqman→file→new→skip→add sequences→选择一对引物的.seq和.abi 格式的文件双击(或者选中文件→add)→done→assemble→双击右侧的conting→看峰的好坏进行裁剪→contig→save consensus→single file→命名保存为.fas格式→file→close
mega比对
用mega打开所需比对的文件,如果要添加序列可以选中最下面一个基因序列的一个碱基,右键copy。
选中一个碱基→W→align DNA→OK→OK
→alignment→align by clustalw→OK→OK
将比对完的数据另存为mega格式
用mega打开该文件
点击TA
C:保守位点V:变异位点Pi:简约信息位点S:单个位点0 :0倍退化位点 2 :2倍退化位点 4 :4倍退化位点Statistics→nucleotide composition 核苷酸组成
Distance→compute pairmise distance…→OK→compute两两遗传距离。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
序列拼接工具使用指南金唯智
A. 序列拼接工具在哪
B. 如何使用序列拼接工具-使用测序订单号拼接-上传.ab1文件拼接
序列拼接工具在哪?
登录金唯智订单系统 工具箱 序列拼接
如何使用序列拼接工具?
A.通过测序订单号拼接(推荐)
B.通过上传.ab1文件拼接
A. 使用测序订单号
①输入所需拼接的订单号(如有多份订单用“;”隔开)
②点击拼接就会出现拼接好的结果,根据需要下载对应格式的文件
B.通过上传ab1文件(测序结果)进行拼接
①点击上传ab1文件
②选中需拼接的样品,点击打开,系统即会自动进行拼接,结果同A中方法一致,下载操作。
