生物信息学_高通量测序技术及数据分析
高通量测序技术的生物信息学分析

高通量测序技术的生物信息学分析引言:高通量测序技术作为一种新型基因测序技术,已经被广泛应用于现代生物研究中。
利用高通量测序技术,科学家们可以快速解码基因组序列、转录组序列以及蛋白质组序列。
然而,高通量测序技术不仅仅是一种实验技术,它也需要强大的生物信息学工具来支持数据分析和解读。
本文将介绍高通量测序技术的生物信息学分析,包括原始数据质量控制、序列比对和注释、基因表达分析、基因变异分析等等。
一、原始数据质量控制原始数据质量控制是高通量测序数据分析的第一步,其目的是剔除低质量序列以及包含污染序列的读段,确保下游的数据分析结果准确可靠。
利用FASTQC等软件对原始的FASTQ格式数据进行质量评估,可以得到关于以下几个质量指标的信息:1. GC含量2. Q20和Q30的比例3. 碱基分布的均匀性4. 过度重叠序列的比例基于以上质量指标,可以对数据进行质量控制处理,包括碱基修剪、低质量序列过滤、去除污染序列等等。
二、序列比对和注释序列比对指的是将高通量测序数据基因组参考序列进行比对,得到参考序列上的SNP、InDel的信息,从而对样品进行全面的基因变异检测、注释和分析。
在序列比对和注释的过程中,需要正确选择合适的比对软件和参考序列。
目前较为流行的比对软件包括BWA、Bowtie、STAR等等。
针对RNA-seq数据的注释工具包括Cufflinks、StringTie、Transcriptome Assembly等等。
基于参考序列的比对结果,还可以利用Variant Effect Predictor (VEP)等工具对候选变异位点进行注释。
注释信息包括dbSNP、ClinVar、ExAC等公共数据库的信息,帮助生物学家了解该变异的生物学特性,并识别其潜在的影响。
三、基因表达分析高通量测序技术还可以用于RNA表达谱的分析,以揭示不同组织和不同发育阶段的基因表达差异。
在基因表达分析中,首先将RNA-seq数据进行质量控制和过滤,然后对序列进行比对和注释,获得基因的计数信息。
高通量基因表达数据分析与生物信息学方法综述

高通量基因表达数据分析与生物信息学方法综述概述随着高通量测序技术的不断发展,基因表达数据的产生速度和规模大幅增加。
这些数据的分析对于深入理解生物体内基因调控网络和相关疾病的发生机制具有重要意义。
生物信息学方法的应用为高通量基因表达数据的解读提供了强大的工具。
本文将综述高通量基因表达数据分析的方法,包括预处理、差异表达分析、功能注释以及数据可视化方法。
1. 高通量测序数据的预处理高通量测序数据包括了RNA测序、小RNA测序、DNA甲基化测序等。
在进行数据分析之前,需要进行一系列的预处理步骤以确保数据的质量和准确性。
预处理包括了去除低质量序列、去除接头污染、去除PCR扩增产物、去除宿主污染等。
一般使用的预处理工具包括Trim Galore、Fastx-toolkit、Cutadapt等。
2. 差异表达分析差异表达分析是高通量基因表达数据分析的重要步骤,通过对实验组和对照组进行比较,筛选出在不同生物条件下显著表达变化的基因。
在差异表达分析中,常使用的方法包括DESeq、edgeR、limma等。
这些方法可以考虑到数据的离散性和复现性,并对差异表达结果进行统计显著性检验。
3. 功能注释功能注释是对差异表达基因的功能进行解读和理解的过程。
通过将差异表达基因与公共数据库进行比对,可以确定基因的功能和相关代谢通路。
常用的功能注释工具包括DAVID、KOBAS、GOseq、KEGG等。
这些工具可以对差异表达基因进行富集分析、通路分析和功能注释。
4. 数据可视化方法数据可视化是高通量基因表达数据分析的重要环节,能够直观地展示数据分布、差异表达基因的表达模式和特征。
常用的数据可视化工具包括ggplot2、heatmap、PCA、Venn图等。
这些工具可以绘制柱状图、热图、散点图、气泡图等多种图形,帮助研究人员深入理解基因表达数据。
结论高通量基因表达数据分析需要经过预处理、差异表达分析、功能注释和数据可视化等多个步骤。
生物信息学中的高通量测序数据分析研究

生物信息学中的高通量测序数据分析研究在生物学领域中,随着人们对生物系统的研究日益深入,高通量测序技术的出现为基因和蛋白质序列的鉴定和分析提供了非常有效的手段。
高通量测序数据分析研究作为生物信息学领域中的一个重要分支,以其高效、快速、准确的特性,不断地吸引着研究者的关注。
高通量测序技术是指一种能够同时测定大量DNA序列的方法,它不仅能够用于基因组组装和注释,还可以通过RNA测序技术来分析基因表达。
这种技术极大地加快了生物学研究的速度和效率,为生物学家提供了大量的测序数据。
然而,这些数据的处理和分析却是非常复杂的。
首先,高通量测序数据的预处理是数据分析的第一步。
当测序数据被生成后,必须对其进行质量控制、过滤低质量序列、去除接头序列等处理,才能得到较为准确的数据。
此外,还有一些其他的预处理过程,例如去重、去序列污染等。
接着,对于各类高通量测序数据进行生物信息学分析非常关键。
生物信息学分析的主要目的是确定基因或蛋白质序列,并了解它们在不同生理状态下的表达和功能。
对于RNA测序数据,其主要方法是将原始数据经过拼接或比对成转录本,然后对得到的转录本进行表达量分析,从而确定不同表达和差异表达基因的信息。
而对于DNA测序数据,则可通过基因组拼装、变异分析、基因预测和功能注释等方法进行深入研究。
近年来,生物信息学领域发展迅速,高通量测序数据分析也成为了该领域中的一个热门研究方向。
许多学者正在研究开发更加准确、更加高效、更加专业的分析工具和方法。
例如,结合机器学习技术的表达量分析方法和差异表达基因分析,能够更加准确地发现差异表达的基因;metagenomics(环境微生物组学)领域,则还需要研究多样性分析、代谢通路分析等更为复杂的问题。
总之,高通量测序数据分析是一个关键的研究领域,它为更深入、全面的生物学研究提供了非常重要的工具和方法。
未来,生物信息学领域需要进一步发展,带来更多的高精度、高效率的数据处理和分析技术,以满足更严格的生物学研究需求。
生物信息学中的高通量基因测序数据处理与分析

生物信息学中的高通量基因测序数据处理与分析随着高通量基因测序技术的发展,大量的基因测序数据得以产生。
这些数据对于生物信息学的研究和应用具有重要意义,因此高通量基因测序数据的处理与分析成为了生物信息学领域的重点之一。
高通量基因测序数据处理是指对原始的测序数据进行加工、清洗和预处理的过程。
首先,需要将测序数据从测序仪中读取出来,得到序列文件。
接着,需要对序列文件进行质量控制,去除低质量序列,过滤掉可能的污染和重复序列。
其次,对于RNA测序数据,需要对序列进行去除adaptor序列、多态性核苷酸等预处理步骤。
最后,对于基因组测序数据,还需要进行比对到参考基因组的工作。
高通量基因测序数据的处理过程中,还需要注意到错误和偏倚的存在。
测序错误可以来源于测序仪的误差,也可以来源于PCR扩增的偏差。
针对这些问题,研究人员可以利用错误纠正算法和统计模型来识别和修复错误的测序数据。
同时,也可以通过样本间和实验间的重复测序来评估和控制测序的偏差。
处理完高通量基因测序数据之后,研究人员就可以进一步进行数据分析。
高通量基因测序数据的分析主要包括基因定量和差异表达分析、基因组注释和变异分析、及关联分析和机器学习等。
基因定量分析可以通过计算读数或转录本的丰度来研究基因的表达模式;差异表达分析可以用于比较不同条件或组织中基因的表达差异,从而找出与特定生物过程或疾病相关的基因。
基因组注释分析可以将基因定位到基因组中的特定位置,并评估基因功能和调控元件的存在。
变异分析可以用于检测和注释基因组中的突变和多态性,研究其与疾病相关性,以及对个体差异的贡献。
关联分析和机器学习可以挖掘大规模测序数据中的相关性和模式,为生物学研究提供新的理解和预测。
为了更好地处理和分析高通量基因测序数据,研究人员还需要掌握一些常用的生物信息学工具和算法。
例如,常用的序列比对算法包括BLAST、Bowtie、BWA等;基因定量和差异表达分析可使用DESeq2、edgeR、limma 等;基因组注释可利用Ensembl、NCBI、UCSC等数据库和工具。
高通量测序的生物信息学分析
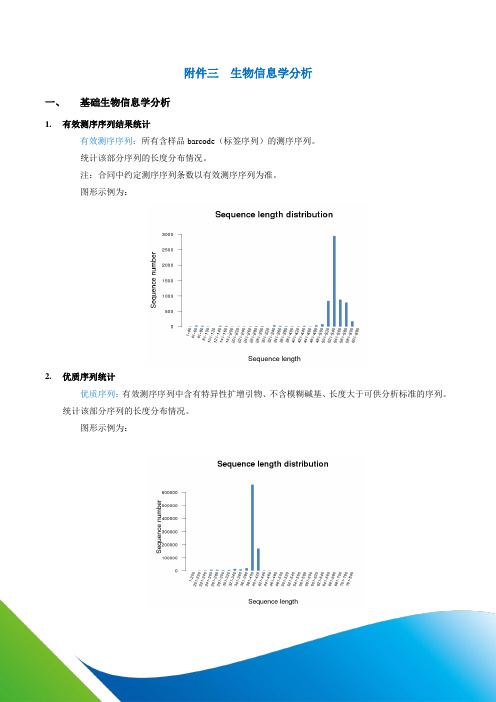
附件三生物信息学分析一、基础生物信息学分析1.有效测序序列结果统计有效测序序列:所有含样品barcode(标签序列)的测序序列。
统计该部分序列的长度分布情况。
注:合同中约定测序序列条数以有效测序序列为准。
图形示例为:2.优质序列统计优质序列:有效测序序列中含有特异性扩增引物、不含模糊碱基、长度大于可供分析标准的序列。
统计该部分序列的长度分布情况。
图形示例为:3.各样本序列数目统计:统计各个样本所含有效测序序列和优质序列数目。
结果示例为:样品有效序列优质序列AB4.OTU生成:根据序列的相似性,将序列归为多个OTU(操作分类单元),以便后续分析。
OTU name A B C D E F G HOTU1 149 410 27 252 45 124 136 101OTU2 0 0 0 0 0 0 0 0OTU3 2 3 14 23 1 5 17 29OTU4 0 47 0 11 0 5 1 7OTU5 19 28 82 9 57 45 303 9OTU6 0 0 0 0 0 0 0 0OTU7 0 182 94 24 14 5 12 60OTU8 0 0 0 0 0 0 0 0...... …………………………………………5.稀释曲线(rarefaction 分析)根据第4条中获得的OTU数据,做出每个样品的Rarefaction曲线。
本合同默认生成OTU相似水平为0.03的rarefaction曲线。
rarefaction曲线结果示例:6.指数分析计算各个样品的相关分析指数,包括:•丰度指数:ace\chao•多样性指数:shannon\simpson•本合同默认生成OTU相似水平为0.03的上述指数值。
多样性指数分析结果示例:注:默认分析以上所列指数,如有特殊需要请说明。
7.Shannon-Wiener曲线利用各样品的测序量在不同测序深度时的微生物多样性指数构建曲线,反映各样本在不同测序数量时的微生物多样性。
生物信息学与高通量数据分析

生物信息学与高通量数据分析生物信息学是一门综合性学科,通过计算机科学和统计学的原理与方法,来研究生物学中的基因组学、转录组学、蛋白质组学等领域。
而高通量数据分析则是生物信息学中的一个重要分支,其涉及大规模的数据收集、处理和解析,以揭示生物系统的运作方式以及恶性疾病的形成机制。
本文将简要介绍生物信息学和高通量数据分析的基本概念、方法和应用。
一、生物信息学的基本概念与研究方法生物信息学是将计算机科学和生物学相结合的学科,旨在利用计算机和统计学的方法来解析与理解生物学中的大量数据。
它包括生物数据库的构建与管理、序列比对与注释、基因表达数据的挖掘与分析等内容。
生物信息学通过整合和分析这些数据,揭示宏观和微观层面上的生物学规律,为科学家们提供深入研究生物系统的便捷工具。
在生物信息学中,常用的研究方法包括序列比对、蛋白质结构预测、进化分析和基因表达分析等。
序列比对是通过将待研究的序列与已知序列进行比对,以找出其相似性和功能区域。
蛋白质结构预测则是根据已知的蛋白质结构,通过计算机模拟和算法预测待研究蛋白质的二级、三级结构。
进化分析通过比较不同生物种群的基因组、蛋白质或DNA序列,推断它们的亲缘关系和进化历程。
基因表达分析则是通过测定不同条件下基因的表达水平,来研究基因调控与功能。
二、高通量数据分析的意义与挑战高通量数据分析是生物信息学中的重要组成部分,其包括了基因组学、转录组学、蛋白质组学等大规模数据的采集、处理和解析过程。
高通量数据来源于一系列高效的实验技术,如基因芯片、测序技术和质谱技术等。
这些技术的发展使得大量生物数据的快速获取成为可能,提供了解析生物系统和研究疾病机制的宝贵资源。
然而,高通量数据分析也面临着许多挑战。
首先,数据量庞大,需要进行高效的存储和处理;其次,数据质量不稳定,可能存在实验误差和测量偏差,需要进行数据清洗和预处理;此外,数据分析方法和工具多样,选择合适的分析策略对于结果的准确性和可靠性至关重要。
高通量基因测序数据分析及生物信息学算法评估

高通量基因测序数据分析及生物信息学算法评估近年来,随着测序技术的发展和普及,高通量基因测序已成为研究生物学和医学领域的重要工具。
高通量基因测序技术能够迅速、准确地获得大量的DNA或RNA序列数据,为研究人员提供了大量的数据资源,但同时也带来了数据分析和算法评估的挑战。
高通量基因测序数据分析是对产生的海量序列进行处理和解析的过程,其目的是从混合的DNA或RNA样本中准确地识别和描述基因组中的序列变异和表达变化。
这一过程通常包括质量控制、序列比对、变异检测、基因表达分析等步骤。
质量控制是高通量基因测序数据分析的起点,它主要用于检测和过滤掉低质量的序列数据。
低质量的序列数据可能由于测序仪器或实验操作等原因导致错误或偏倚,因此需要通过质量控制筛选出高质量的数据,以保证后续的分析结果准确可靠。
序列比对是高通量基因测序数据分析中的核心步骤之一,它将测序得到的短序列与已知的参考基因组或转录组序列进行比对。
目前常用的比对算法包括BWA、Bowtie等。
比对的目的是确定每个测序片段与参考序列的匹配位置,从而获得关于基因组中变异和表达的信息。
变异检测是高通量基因测序数据分析的重要任务之一。
通过比对结果,可以识别出基因组中的SNP(单核苷酸多态性)、Indel(插入缺失)等变异。
变异检测的方法包括基于规则的变异挖掘、基于统计学的变异检验等,这些方法能够帮助研究人员分析和理解基因组的个体差异。
基因表达分析是高通量基因测序数据分析的另一个重要任务。
通过比对并计算每个基因的表达水平,可以研究基因的功能和调控机制。
常用的基因表达分析方法包括RPKM(Reads Per Kilobase of transcript per Million mapped reads)、DESeq2(Differential Expression analysis based on the Negative Binomial Distribution)等。
生物信息学中的高通量数据处理与分析

生物信息学中的高通量数据处理与分析随着生物技术的不断发展,生物研究过程中产生的数据量越来越大,从基因测序到蛋白质组学,都需要处理和分析海量的数据。
这些数据需要高效地存储、管理、分析和可视化,这就需要生物信息学中的高通量数据处理与分析。
I. 生物信息学中的高通量数据高通量数据是指能够同时分析多个样品或者检测多个分子的数据。
在生物实验中,高通量数据主要来自基因测序、轮廓芯片、蛋白质组学等领域。
这些数据的产生量及其复杂性已经远远超过了传统的实验方法。
1. 基因测序数据基因测序是现代遗传学和生物学的基础工具。
通过对基因组、转录组或者蛋白质组的测序,可以帮助我们深入理解生物体的结构和功能。
基因测序数据量巨大,每一次基因测序都会产生几百万到几十亿条碱基序列。
这些数据需要经过预处理、比对、拼接、注释等复杂的处理之后才能生成可靠的结果。
2. 蛋白质组学数据蛋白质组学是研究生物体内蛋白质的种类、数量、结构和功能的学科。
蛋白质组学数据包括蛋白质组的鉴定、定量和结构分析。
鉴定蛋白质需要将复杂的混合物分离出来,通过蛋白质质谱技术进行鉴定。
这个过程中需要对大量的数据进行分析和解释,通过与数据库的比对,找到与之匹配的蛋白质。
II. 高通量数据处理与分析的挑战处理和分析高通量数据需要面对许多挑战。
首先,这些数据量非常庞大,处理过程需要大量的计算资源和存储空间。
其次,处理的过程非常复杂,需要运用许多不同的算法和工具。
最后,数据的质量也是一个重要的因素。
高通量数据中存在许多误差和噪声,因此需要有效的数据预处理和质量控制方法。
1. 数据预处理数据预处理是数据分析的第一步。
基因测序数据需要进行序列质量评估、序列去重、序列比对、序列拼接等处理,才能得到有效的结果。
蛋白质组学数据需要进行数据清洗、信噪比估计、谱酶定量、定量值筛选、差异分析等处理。
2. 数据分析对于高通量数据的分析一般分为两种:有监督的分析和无监督的分析。
有监督的分析是基于已有的信息对数据进行分析和解释,例如对基因测序数据进行差异分析、基因富集分析等。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
– 参考文献
• Metzker, M.L. (2010). Sequencing technologies - the next generation. Nat Rev Genet 11, 31-46.
/nrg/journal/v11/n1/full/nrg2626.html
背景介绍
背景介绍
• 第一代测序技术
– Sanger测序法
• 链终止法 • 双脱氧终止法 • 1975年
Frederick Sanger 弗雷德里克· 桑格 1918年8月13日-2013年11月19日 1958年 诺贝尔化学奖 1980年 诺贝尔化学奖
Transcription
/s/blog_7110867f0100zi09.html
基因芯片与高通量 测序的比较
芯片与测序比较
• 基因芯片
– 约20年的历史,技术比较成熟,成本相对较低 – 原理
• 探针,互补配对的原则 • 靶序列用荧光标记 • 通过荧光强度间接反映靶序列的数量
生物信息学
高通量测序技术及数据分析介绍
高通量测序技术及数据分析介绍
• 背景介绍
– 第一代测序技术 – 第二代(高通量)测序技术
• 基因芯片与高通量测序的比较 • 高通量测序技术的应用
– – – – – – 高通量测序数据分析概览 高通量测序数据质量评估与过滤 基因组测序 RNA-seq ChIP-seq UCSC Genome Bioinformatics
背景介绍
• 以Illumina为例简单介绍测序原理
cBot
Illumina HiSeq 2500
背景介绍
• 高通量测序数据格式
– fasta
• 序列文件的第一行是由大于符号(>)打头的任意文 字说明,主要为标记序列用。从第二行开始是序列 本身,标准核苷酸符号,通常核苷酸符号大小写均 可
– fastq
背景介绍
• 第二代测序技术
– 边合成边测序
• 2005年左右 • Sequencing by synthesis
Illumina HiSeq 2500
– 代表性测序技术
• • • • • Illumina/Solexa Roche/454 ABI/SOLiD Polonator HeliScope
NCBI/Sanger or Illumina 1.8 and later. Using a Phred scale encoded using ASCII 33 to 93. This is the standard for fastq formats except for the early Illumina data formats (this changed with version 1.8 of the Illumina Pipeline). Illumina Pipeline 1.2 and earlier. Using a Solexa/Illumina scale (-5 to 40) using ASCII 59 to 104. The Workbench automatically converts these quality scores to the Phred scale on import in order to ensure a common scale for analyses across data sets from different platforms (see details on the conversion next to the sample below). Illumina Pipeline 1.3 and 1.4. Using a Phred scale using ASCII 64 to 104. Illumina Pipeline 1.5 to 1.7. Using a Phred scale using ASCII 64 to 104. Values 0 (@) and 1 (A) are not used anymore. Value 2 (B) has special meaning and is used as a trim clipping. This means that when selecting Illumina Pipeline 1.5 and later, the reads are trimmed when a B is encountered in the input file if the Trim reads option is checked. 36 39 39 39 39 39 39 39 39 39 38 39 39 36 36 34 34 29 31 2 20 20 19 19 19 38 38 38 36 36 36 36 36 36 30 32 35 3-end
• 首先将DNA样本进行片段化处理形成200-500bp的片 段,引物序列连接到DNA片段的一端,然后末端加 上接头,将片段固定在flow cell上生成DNA簇,上机 测序单端读取序列。
– 双末端测序,pair,在第一轮测序完成后,去除第一轮 测序的模板链,引导互补链在原位置再生和扩增, 以达到第二轮测序所用的模板量,进行第二轮互补 链的合成测序。
• 第一行由‘@’开始,后面跟着序列的描述信息,这点 跟fasta格式是一样的;第二行是序列;第三行由‘+’ 开始,后面也可以跟着序列的描述信息;第四行是 第二行序列的质量评价(quality values),字符数跟 第二行的序列是相等的。背介绍• 高通量测序数据格式
– fastq
Q =-10 log10(p) OR Q =-10 log10[p/(1-p)] (p:碱基错误率) 字符的ASCII值 - 64 = 质量值 OR 字符的ASCII值 - 33 = 质量值
