dnaman比对序列结果解读
DNAman使用说明

查看文章DNAMAN使用说明书(中文)2008年04月16日星期三下午10:50DNAMAN 是一种常用的核酸序列分析软件。
由于它功能强大,使用方便,已成为一种普遍使用的DNA 序列分析工具。
本文以DNAMAN 5.2.9 Demo version 为例,简单介绍其使用方法。
打开DNAMAN,可以看到如下界面:第一栏为主菜单栏。
除了帮助菜单外,有十个常用主菜单,第二栏为工具栏:第三栏为浏览器栏:在浏览器栏下方的工作区左侧,可见Channel 工具条,DNAMAN 提供20 个Channel,(如左所示:)点击Channel 工具条上相应的数字,即可击活相应的Channel。
每个Channel 可以装入一个序列。
将要分析的序列(DNA 序列或氨基酸序列)放入Channel 中可以节约存取序列时间,加快分析速度。
此版本DNAMAN 提供自动载入功能,用户只需激活某个Channel,然后打开一个序列文件,则打开的序列自动载入被激活的Channel 中。
本文以具体使用DNAMAN 的过程为例来说明如何使用DNAMAN 分析序列。
1.将待分析序列装入Channel(1)通过File Open 命令打开待分析序列文件,则打开的序列自动装入默认Channel。
(初始为channel1)可以通过激活不同的channel (例如:channel5)来改变序列装入的Channel。
(2)通过Sequence/Load Sequence 菜单的子菜单打开文件或将选定的部分序列装入Channel 。
通过Sequence/Current Sequence/Analysis Defination 命令打开一个对话框,通过此对话框可以设定序列的性质(DNA 或蛋白质),名称,要分析的片段等参数。
2.以不同形式显示序列通过Sequence//Display Sequence 命令打开对话框,如下图所示:根据不同的需要,可以选择显示不同的序列转换形式。
DNA序列分析与结构比对

DNA序列分析与结构比对DNA(脱氧核糖核酸)是构成遗传物质的分子,它指导所有生命的形成和发展。
DNA序列是由不同的碱基对组成的排列顺序,而这些碱基对的不同排列顺序,决定了不同的生物体的基因特征。
因此,DNA序列的分析和比对,对于理解生命的机理、诊断和治疗疾病都具有非常重要的意义。
一、DNA序列的分析DNA序列的分析是指对DNA序列进行测序、注释、分类、比对等过程。
DNA测序是一项基础的实验前提,通过它我们可以获取到DNA序列的数据。
DNA注释是将测序数据进行翻译、比对和分类,并以一定的方式存储。
在分类的过程中,我们可以将DNA序列根据不同的类型进行分类,如线粒体DNA、叶绿体DNA和核DNA等等。
我们可以通过对DNA序列的分析,来研究基因,从而探索生命的本质和各种生物体的演化过程。
二、DNA序列的比对DNA序列的比对是将两个DNA序列进行对比,确定其相同和不同之处的过程。
DNA序列的比对可以用于基因检测、病理诊断、动物进化研究等领域。
在DNA序列的比对当中,比对技术是非常核心的一部分。
目前,主要有以下两种DNA序列比对的算法:1、全局比对:通过比对整个DNA序列来确定差异。
全局比对的优点在于比对的结果非常准确,但是由于比对的长度过长,所以计算速度相对较慢。
2、局部比对:主要是针对两个DNA序列中长度较短的区域,进行匹配和比对。
局部比对的优点在于比对速度非常快,但是比对的结果可能仅限于某一段序列,因此需要进行针对性的分析。
三、DNA序列的结构比对DNA序列的结构比对指的是查找DNA序列中的一些结构特征,例如基础对序列、序列的二级结构以及序列的三级结构等。
DNA序列的结构比对可以通过计算序列的折叠情况、组合情况来求出序列的结构差异。
通过比对不同的序列结构,我们可以获得更精确的结构信息,这些信息在疾病预测、治疗和药物设计上具有重要的价值。
在DNA序列的分析和比对中,遗传多样性是非常重要的一部分。
DNA序列的遗传多样性涵盖了种类、强度、频率等多个方面。
DNAman使用方法教学课件ppt

基因组浏览器
总结词
DnaMan具备可视化基因组浏览器,方便用户查看基因组结构和基因注释。
详细描述
DnaMan的基因组浏览器支持多种基因组数据可视化展示,包括基因注释、基因 转录本、SNP、重复序列等,可帮助用户深入分析基因组结构及功能。
分子进化分析
总结词
DnaMan支持分子进化分析,可探究物种间的亲缘关系和演 化历程。
详细描述
DnaMan的分子进化分析功能包括多种进化树构建方法,如 NJ树、MP树、ML树等,并提供了丰富的分子进化分析工具 ,如PAML、CODEML等。
04
DnaMan使用中的常见问题及解决 办法
序列导入问题
总结词
在导入序列时遇到的各种问题,如格式不正确、文件名限制等。
详细描述
DnaMan支持多种格式导入,如FASTA、GenBank等,但常因格式问题导致 导入失败;另外,序列文件名中不能含有特殊字符,否则无法导入。
DnaMan具备强大的序列编辑、可视化和注释功能,同时提 供了大量内置的分析工具和数据库,方便用户进行各种生物 信息学研究。
DnaMan的起源与发展
1
DnaMan起源于20世纪90年代末期,由美国一 家生物技术公司开发。
2
经过多年的发展和不断更新,DnaMan已经成 为了生物信息学领域中广受欢迎的工具之一。
插入视频
添加注释
在课件中插入DnaMan软件操作视频,以 便学生更好地了解软件操作流程和功能使用 方法。
对于一些复杂的操作和功能,可以添加注释 来帮助学生更好地理解和记忆。
演示教学课件的技巧
熟悉课件
在演示教学课件前,需要熟悉课 件的内容和排版,以便更好地讲 解。
互动教学
DNA测序结果中常见的几个问题

1 、为什么开始一段序列的信号很杂乱,几乎难以辨别?这主要是因为残存的染料单体造成的干扰峰所致,该干扰峰和正常序列峰重叠在一起;另外,测序电泳开始阶段电压有一个稳定期,所以经常有20-50 bp 的紧接着引物的片段读不清楚,有时甚至更长。
2 、为什么在序列的末端容易产生N 值,峰图较杂?由于测序反应的信号是逐渐减弱的,所以序列末端的信号会很弱,峰图自然就会杂乱,加上测序胶的分辨率问题,如果碱基分不开,就会产生N 值,正常情况下ABI377测序仪能正确读出500个碱基的有效序列。
3 、测序结果怎么找不到我的引物序列?如果找不到测序所用的引物序列。
这是正常的,因为引物本身是不被标记的,所以在测序报告中是找不到的;如果找不到克隆片段中的扩增引物,可能是您克隆的酶切位点距离您的测序引物太近,开始一段序列很杂,几乎难以辨别,有可能看不清或看不到扩增引物;另外插入片段的插入方向如果是反的,此时需找引物的互补序列。
4 、测序结果怎么看不到我克隆的酶切位点?可能的原因同上,您克隆的酶切位点距离您的测序引物太近,开始一段序列很杂,几乎难以辨别,有可能看不清或看不到酶切位点。
通常我们会尽量选择距离酶切位点远点的引物,当然,若是样品出现意外原因,如空载、载体自连等,克隆的酶切位点也是看不到的。
5 、你测出的结果与我预想的不一致,给我的结果与我需要的序列有差距,这是怎么回事? 首先,我们会核实给您的测序结果是否对应您的样品编号,如果对应的是您的样品,由于不知您的实验背景,测得的序列是否与您预想的结果一致我们无法判断,我们能做到的是检查发送给您的测序结果和您提供来的样品是否一致。
6 、序列图为什么会有背景噪音(杂带)?是否会影响测序结果?序列图的背景杂带是由荧光染料引起,如果太强会影响测序结果,要看信噪比,我们给的结果信噪比大都在98%以上。
7 、测序结果为什么与标准序列有差别?原因可能有:样品个体之间的差别、测序准确率的问题,自动测序仪分析序列的准确并非100%,建议至少测一次双向,通过双向测序可以最大限度减少测序的错误。
序列分析软件DNAMAN的使用方法

DNAMAN 是一种常用的核酸序列分析 软件。由于它功能强大,使用方便,已 成为一种普遍使用的DNA 序列分析工具。
打开DNAMAN,可以看到如下界面:
第一栏为主菜单栏。除了帮助菜单外,有十个 常用主菜单, 第二栏为工具栏: 第三栏为浏览器栏: 在浏览器栏下方的工作 区左侧,可见Channel 工具条,DNAMAN 提 供20 个Channel,点击Channel 工具条上相 应的数字,即可击活相应的Channel。 每个Channel 可以装入一个序列。将要分析 的序列(DNA 序列或氨基酸序列)放入 Channel 中可以节约存取序列时间,加快分 析速度。
Annotations 是否显示注释 Comparision 比对参数, 其中Window 代表Window size(单位比对长度), Mismatch 代表Mismatch size(单位比对长度中许 可的错配值)要快速比对,需将此项设为0。 Both stran 代表Both strand(双链比对)选择此项, 是指用Sequence 2 中的序列的正链和负链分别和 Sequence 1 比较。 Sequence 2 正链与Sequence 1 比较结果用黑色点 表示,Sequence 2 负链比对结果用红色点表示。
ห้องสมุดไป่ตู้
选择所需的项目,然后按提示操作点击按扭,出现下 列对话框: 参数说明如下: Enzyme 代表(enzyme data file),点击旁边的下拉按钮, 出现两个默认选项,restrict.enz 和dnamane.enz, 如果添加过自制的酶列表,则附加显示自制酶列表文 件名。 其中restrict.enz 数据文件包含180 种限制酶, dnamane.enz 数据文件包含2524 种限制酶。 选择其中一个数据文件,相应的酶在左边的显示框中 列出(按酶名称字母表顺序),鼠标双击酶名称,则 对应的酶被选中,在右边空白框中列出。
序列分析软件DNAMAN_的使用方法中文

饶志明
博士/教授/ 博士生导师
江南大学生物工程学院工业微生物中心 江南大学工业生物技术教育部重点实验室
E-mail: raozhm@
DNAMAN 是一种常用的核酸序列分析 软件。由于它功能强大,使用方便,已 成为一种普遍使用的DNA 序列分析工具。
oligoprimerprimergeneratordnaman如何使用primerpremier50引物设计多重pcr引物设计探针设计引物评析引物同源性分析用blastn软件进行同源性比较选择两个引物3端与同一非目的基因不同源的序列作为引物设计题目一xx微生物xx酶基因的克隆及其在大肠杆菌中的表达目的基因编码如耐高温淀粉酶生淀粉酶普鲁兰酶植酸酶碱性甘露聚糖酶脂肪酶碱性蛋白酶谷氨酰胺转移酶木聚糖酶纤维素降解酶甘油脱氢酶甘油脱水酶荧光素酶葡萄糖苷酶过氧化物酶过氧化氢酶已糖激酶葡萄6磷酸脱氢酶乳酸脱氢酶苹果酸脱氢酶尿酸酶胆红素氧化酶吡喃葡萄糖激酶单宁酶表达载体
(2)多序列同源性分析
通过打开Sequence/Multiple Sequence Alignment 命令打开对话框, 参数说明如下: File 从文件中选择参加比对的序列 Folder 从文件夹中选择参加比对的序列 Channel 从channel 中选择参加比对的序列 Dbase 从数据库中选择参加比对的序列 Remove 清除选择的序列(鼠标点击左边显示框中的 序列名选择) Clear 清除全部序列
2. 以不同形式显示序列
通过Sequence//Display Sequence 命令打开对话框, 根据不同的需要,可以选择显示不同的 序列转换形式。 对话框选项说明如下: Sequence &Composition 显示序列和 成分
01_dnaman_介绍(1)

快速启动
DNAMAN使用Channel(通道)保存用于分析的活动序列
启动DNAMAN程序 1.点按序列控制中的“File”标签 2.双击Example1.seq 3.点按序列控制中的“Channel ”标签 示例1序列载入通速启动
1 3
2
示例1序列载入通道1 1. 点击Restriction Analysis 工具, 或 2. 点击Translation 工具, 或
DNAMAN 序列分析软件
生物信息学平台
DNAMAN 特征
综合序列分析系统 序列编辑 限制性分析-电子克隆 多序列比对 系统发育分析 点图分析 顺序装配 PCR引物选择 蛋白质序列分析 数据库 – 序列管理
DNAMAN 用户界面
DNAMAN 用户界面
快速工具栏 序列控制 浏览器栏
功能区工具栏 文档栏 文档
3. 点击Search 工具
Restriction Analysis
1 2
点击Restriction Analysis 工具 1. 选中Show Summary 2. 选中Draw Restriction Map 3. 点击Next
3
Restriction Analysis
2
Restriction Analysis 1. 点击3’ Overhang 2. 按下Select All Button 3. 点击Finish
1 3
Restriction Analysis
3 12
Restriction Analysis 1. 选择Annotations 2. 点击OK 按钮 3. 分析结果
序列分析软件DNAMAN的使用方法简介
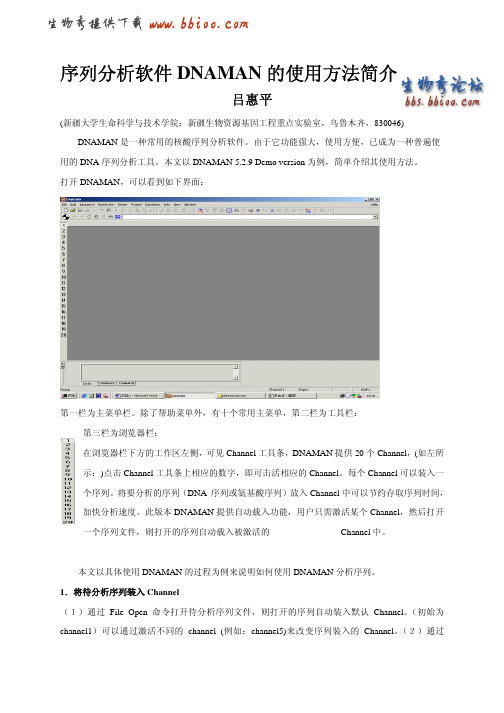
序列分析软件DNAMAN的使用方法简介吕惠平(新疆大学生命科学与技术学院;新疆生物资源基因工程重点实验室,乌鲁木齐,830046) DNAMAN是一种常用的核酸序列分析软件。
由于它功能强大,使用方便,已成为一种普遍使用的DNA序列分析工具。
本文以DNAMAN 5.2.9 Demo version为例,简单介绍其使用方法。
打开DNAMAN,可以看到如下界面:第一栏为主菜单栏。
除了帮助菜单外,有十个常用主菜单,第二栏为工具栏:第三栏为浏览器栏:在浏览器栏下方的工作区左侧,可见Channel工具条,DNAMAN提供20个Channel,(如左所示:)点击Channel工具条上相应的数字,即可击活相应的Channel。
每个Channel可以装入一个序列。
将要分析的序列(DNA 序列或氨基酸序列)放入Channel中可以节约存取序列时间,加快分析速度。
此版本DNAMAN提供自动载入功能,用户只需激活某个Channel,然后打开一个序列文件,则打开的序列自动载入被激活的 Channel中。
本文以具体使用DNAMAN的过程为例来说明如何使用DNAMAN分析序列。
1.将待分析序列装入Channel(1)通过File Open命令打开待分析序列文件,则打开的序列自动装入默认Channel。
(初始为channel1)可以通过激活不同的channel (例如:channel5)来改变序列装入的Channel。
(2)通过Sequence/Load Sequence菜单的子菜单打开文件或将选定的部分序列装入Channel。
通过Sequence/Current Sequence/Analysis Defination命令打开一个对话框,通过此对话框可以设定序列的性质(DNA 或蛋白质),名称,要分析的片段等参数。
2.以不同形式显示序列通过Sequence//Display Sequence命令打开对话框,如下图所示:根据不同的需要,可以选择显示不同的序列转换形式。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
dnaman比对序列结果解读
摘要:
1.Dnaman 比对序列结果
2.结果解读
3.结论
正文:
在生物信息学领域,Dnaman 是一款广泛使用的DNA 比对工具,通过对比两个或多个DNA 序列,可以揭示它们之间的相似性和差异性。
本文将基于Dnaman 比对序列结果,对其进行解读。
首先,我们来了解Dnaman 比对序列结果。
Dnaman 将输入的DNA 序列进行比对,生成比对结果文件,其中包括了比对过程中所涉及到的所有参数。
这些参数包括比对算法、比对分数、插入、删除、替换等。
通过对这些参数的解读,可以了解序列之间的相似程度以及具体的差异所在。
接下来,我们对比对结果进行解读。
比对分数是衡量两个序列相似性的重要指标,分数越高,表示序列之间的相似性越高。
在Dnaman 的比对结果中,会列出每个位置的比对分数。
通过观察这些分数,可以发现序列间的相似性和差异性。
如果分数较低,说明该位置的序列差异较大,可能是由于插入、删除或替换等操作导致的。
而如果分数较高,则说明该位置序列高度保守,即两者间的序列相似。
在解读过程中,还需要注意插入、删除和替换等操作。
这些操作会导致序列间的差异,进而影响比对结果。
插入是指在参考序列中不存在的核苷酸在比对序列中出现,而删除则是指比对序列中的核苷酸在参考序列中不存在。
替换
则是指比对序列中的核苷酸与参考序列中的核苷酸不同。
在解读过程中,需要分析这些操作的分布和性质,以便更好地理解序列间的关系。
根据以上分析,我们可以得出结论:通过对Dnaman 比对序列结果的解读,可以揭示DNA 序列之间的相似性和差异性,以及序列间的关系。
这对于研究基因组结构、功能和进化等方面具有重要意义。
