Oligo引物设计软件使用方法
primer5和oligo使用技巧概况

一、Primer Premier 5.0 的使用技巧简介1. 功能“Premier”的主要功能分四大块,其中有三种功能比较常用,即引物设计、限制性内切酶位点分析和DNA 基元(motif查找。
“Premier”还具有同源性分析功能,但并非其特长,在此略过。
此外,该软件还有一些特殊功能,其中最重要的是设计简并引物,另外还有序列“朗读”、DNA 与蛋白序列的互换、语音提示键盘输入等等。
有时需要根据一段氨基酸序列反推到DNA 来设计引物,由于大多数氨基酸(20 种常见结构氨基酸中的18 种的遗传密码不只一种,因此,由氨基酸序列反推DNA 序列时,会遇到部分碱基的不确定性。
这样设计并合成的引物实际上是多个序列的混和物,它们的序列组成大部分相同,但在某些位点有所变化,称之为简并引物。
遗传密码规则因物种或细胞亚结构的不同而异,比如在线粒体内的遗传密码与细胞核是不一样的。
“Premier”可以针对模板DNA 的来源以相应的遗传密码规则转换DNA 和氨基酸序列。
软件共给出八种生物亚结构的不同遗传密码规则供用户选择,有纤毛虫大核(Ciliate Macronuclear、无脊椎动物线粒体(Invertebrate Mitochon drion、支原体(Mycoplasma、植物线粒体(Plant Mitochondrion、原生动物线粒体(P rotozoan Mitochondrion、一般标准(Standard、脊椎动物线粒体(Vertebrate Mitochondr ion和酵母线粒体(Yeast Mitochondrion。
2. 使用步骤及技巧“Premier”软件启动界面如下:其主要功能在主界面上一目了然(按钮功能如上述。
限制性酶切点分析及基元查找功能比较简单,点击该功能按钮后,选择相应的限制性内切酶或基元(如-10 序列,-35 序列等,按确定即可。
常见的限制性内切酶和基元一般都可以找到。
你还可以编辑或者添加新限制性内切酶或基元。
oligo7官方说明 中文版

稳定性较差的引物与产物相差30°Tm。一组警告并不会取消它的资格,而只是表明它可能不是最优的选择。 当警告是“前向起爆药的终端稳定性太高”时,并不意味着反应会受到任何损害。这意味着当使用复杂的 底物时,如基因组DNA或逆转录的总mRNA,你可能会有错误的产物形成问题。至少有必要仔细检查预测产品 的大小。
The next slide shows the calculation graphically (data for temperature of 37° ).
DG 计算示例
Total DG = 1+1+0.3+0.5-1.3-1.3-2.2 = -2.0 kcal/mol
这是一种罕见情况。通常情况下,悬挂的末端正在稳定下来。这是在提醒你,有时添加一个基 地可能会降低双相熔化温度,后面展示了一个即使添加两个碱基也不会影响TM的例子
在搜索引物和探针之后,您可 以打开选定的寡核苷酸窗口。 在此示例中,在所选寡核苷酸 窗口底部列出的文件中执行了 对一致性引物的排序搜索(要以 此方式搜索,您需要单击 Search for Primer&Probe(搜 索引物和探针)窗口的 “SubSearches”(子搜索)选项 卡,然后单击“Consensus Primers”(一致性引物)检查 框中,选择文件并开始搜索。)。选中“选择共识寡核 苷酸”框时,如右图所示,当您双击显示有关给定引 物信息的行时,Oligo 7会选择共识引物。
引物位置的列表可以通过点击(或按住Option键单击)窗口的标题来按分数、寡头位置或/和窗 口的任何其他列进行排序。左上角的下拉菜单允许您选择正向或反向引物显示。在搜索T aqMan集合或嵌套引物之后,您将有更多的选择。
寡核苷酸设置窗口
oligo使用说明

oligo使用说明一、介绍oligo是一款功能强大的软件,用于处理DNA或RNA序列的设计和分析。
本文档提供了oligo的使用说明,包括软件安装、功能介绍和操作指南等内容。
二、安装1、软件包在oligo官方网站()上oligo软件的安装包。
2、安装软件双击安装包,按照安装向导的指示完成软件的安装过程。
三、功能介绍oligo拥有以下主要功能:1、序列设计- DNA或RNA序列的快速设计和合成。
- 引物和探针的设计。
- 引物二聚体和杂交结构的模拟。
2、序列分析- 序列的一致性和变异性分析。
- 序列的限制酶切位点分析。
- 序列的物理性质和二级结构预测。
四、操作指南1、序列设计a:创建新项目:在菜单栏中选择“文件”,然后选择“新建项目”。
b:输入序列:在新建项目中,输入待设计的序列。
c:设计引物:选择“设计引物”功能,根据需要设置引物的参数,“开始设计”按钮。
d:查看设计结果:设计完成后,查看引物设计的结果,根据结果选择合适的引物。
2、序列分析a:导入序列:在菜单栏中选择“文件”,然后选择“导入序列”。
b:分析序列:选择“分析序列”功能,根据需要选择相应的分析方法,“开始分析”按钮。
c:查看分析结果:分析完成后,查看分析结果,进行相关的数据分析和解释。
五、附件本文档涉及的附件包括:oligo软件安装包、示例项目文件等。
六、法律名词及注释1、DNA(脱氧核糖核酸):一种存在于细胞中的生物大分子,携带遗传信息。
2、RNA(核糖核酸):一种通过转录过程从DNA中合成的分子,参与蛋白质的合成。
3、引物(primer):DNA或RNA序列,在PCR等实验中用于引导扩增或反向转录。
4、探针(probe):DNA或RNA序列,通过与目标序列特异性结合,进行检测或识别。
七、结束。
Oligo 7 使用教程 个人总结
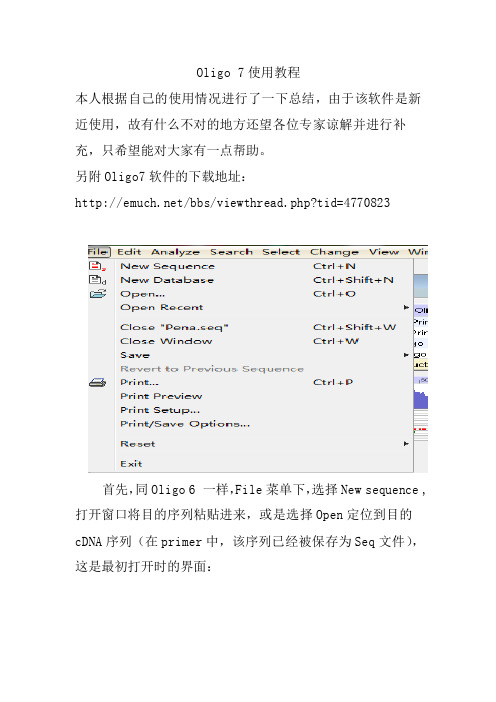
Oligo 7使用教程本人根据自己的使用情况进行了一下总结,由于该软件是新近使用,故有什么不对的地方还望各位专家谅解并进行补充,只希望能对大家有一点帮助。
另附Oligo7软件的下载地址:/bbs/viewthread.php?tid=4770823首先,同Oligo 6 一样,File菜单下,选择New sequence ,打开窗口将目的序列粘贴进来,或是选择Open定位到目的cDNA序列(在primer中,该序列已经被保存为Seq文件),这是最初打开时的界面:然后就是进行引物的设计了。
Search 菜单下,选择for primes &probes ,即出现引物搜寻窗口:根据自己的实际情况选择Parameters 或Ranges设置引物的相关参数和范围。
然后选择Search即开始进行引物的搜索,之后会出现软件所列出的依据得分(Score)高低排列设计的引物。
双击每一行所列出的引物会弹出该对引物的具体信息,以及软件对该对引物的相关评价。
双击之后在最初的Sequence 窗口中就会出现下面的窗口:点击绿色方形图标前面的i标志可了解对应的具体信息。
之后便是对该引物的具体分析了,这部分的分析同Oligo 6基本上是一样的。
选择Analyze菜单,如下图:(1)Analyze中,第一项为Key info,点击Selected primers,会给出两条引物的概括性信息,其中包括引物的Tm值,此值Oligo是采用nearest neighbor method计算,会比Primer5中引物的Tm值略高,此窗口中还给出引物的Delta G和3’端的Delta G.3’端的Delta G过高,会在错配位点形成双链结构并引起DNA聚合反应,因此此项绝对值应该小一些,最好不要超过9。
(2)Analyze中第二项为Duplex Formation,即二聚体形成分析,可以选择上游引物或下游引物,分析上游引物间二聚体形成情况和下游引物间的二聚体情况,还可以选择Upper/Lower ,即上下游引物之间的二聚体形成情况。
Oligo6使用说明

Oligo使用方法介绍作为目前最好、最专业的引物设计软件,Oligo的功能很强,在这里我们介绍它的一些主要功能:如:普通引物对的搜索、测序引物的设计、杂交探针的设计以及评估引物对质量等等。
在正式进行引物设计前,我们首先面临的一个任务就是向Oligo程序导入模板序列,根据不同的实验情况,导入模板有三种方法:1,直接用键盘输入:a,点击file菜单中的New Sequence 浮动命令,或直接点击工具栏中的New Sequence命令,进入序列展示窗口;b,此时即可键入DNA序列;c,如果需要的话,Oligo提供碱基回放功能,在边键入时边读出碱基,防止输入错误。
点击Edit菜单中的“Readback on”即可。
2,利用复制和粘贴:当我们序列已经作为TXT文件存在或其它oligo不能直接open的文件格式,如word 文件.html格式,这个功能就显得很有用了。
在相应文件中复制序列后在序列展示窗口粘贴,oligo会自动去除非碱基字符。
当序列输入或粘贴完成后,点击Accept/Discard菜单中的Accept浮动命令,即可进入引物设计模式。
3,如果序列已经保存为Seq格式或者FASTA,GenBank格式时,oligo就可以直接打开序列文件。
点击File菜单中的“Open”浮动命令,找到所需文件,打开即可。
进入引物设计模式后,oligo一般会弹出三个窗口,分别是6-碱基频率窗口,碱基退火温度窗口以及序列内部碱基稳定性窗口,其中的退火温度窗口是我们引物设计的主窗口,其它的两个窗口则在设计过程中起辅助作用,比如6-碱基频率窗口可以使我们很直观地看到所设计引物在相应物种基因组中的出现频率,如果我们的模板是基因组DNA或混合DNA时,该信息就显得有用了,而内部稳定性窗口则可以显示引物的5’端稳定性是否稍高于3’端等。
一, 普通引物对的搜索:以Mouse 4E(cDNA序列)为例。
我们的目的是以Mouse 4E(2361 bp)为模板,设计一对引物来扩增出600-800bp长的PCR产物。
Oligo6.0软件使用说明

Oligo使用方法介绍作为目前最好、最专业的引物设计软件,Oligo的功能很强,在这里我们介绍它的一些主要功能:如:普通引物对的搜索、测序引物的设计、杂交探针的设计以及评估引物对质量等等。
在正式进行引物设计前,我们首先面临的一个任务就是向Oligo程序导入模板序列,根据不同的实验情况,导入模板有三种方法:1,直接用键盘输入:a,点击file菜单中的New Sequence 浮动命令,或直接点击工具栏中的New Sequence命令,进入序列展示窗口;b,此时即可键入DNA序列;c,如果需要的话,Oligo提供碱基回放功能,在边键入时边读出碱基,防止输入错误。
点击Edit菜单中的“Readback on”即可。
2,利用复制和粘贴:当我们序列已经作为TXT文件存在或其它oligo不能直接open的文件格式,如word 文件.html格式,这个功能就显得很有用了。
在相应文件中复制序列后在序列展示窗口粘贴,oligo会自动去除非碱基字符。
当序列输入或粘贴完成后,点击Accept/Discard菜单中的Accept浮动命令,即可进入引物设计模式。
3,如果序列已经保存为Seq格式或者FASTA,GenBank格式时,oligo就可以直接打开序列文件。
点击File菜单中的“Open”浮动命令,找到所需文件,打开即可。
进入引物设计模式后,oligo一般会弹出三个窗口,分别是6-碱基频率窗口,碱基退火温度窗口以及序列内部碱基稳定性窗口,其中的退火温度窗口是我们引物设计的主窗口,其它的两个窗口则在设计过程中起辅助作用,比如6-碱基频率窗口可以使我们很直观地看到所设计引物在相应物种基因组中的出现频率,如果我们的模板是基因组DNA或混合DNA时,该信息就显得有用了,而内部稳定性窗口则可以显示引物的5’端稳定性是否稍高于3’端等。
一,普通引物对的搜索:以Mouse 4E(cDNA序列)为例。
我们的目的是以Mouse 4E(2361 bp)为模板,设计一对引物来扩增出600-800bp长的PCR产物。
Oligo6.0软件教程

Oligo使用方法介绍作为目前最好、最专业的引物设计软件,Oligo的功能很强,在这里我们介绍它的一些主要功能:如:普通引物对的搜索、测序引物的设计、杂交探针的设计以及评估引物对质量等等。
在正式进行引物设计前,我们首先面临的一个任务就是向Oligo程序导入模板序列,根据不同的实验情况,导入模板有三种方法:1,直接用键盘输入:a,点击file菜单中的New Sequence 浮动命令,或直接点击工具栏中的New Sequence命令,进入序列展示窗口;b,此时即可键入DNA序列;c,如果需要的话,Oligo提供碱基回放功能,在边键入时边读出碱基,防止输入错误。
点击Edit菜单中的“Readback on”即可。
2,利用复制和粘贴:当我们序列已经作为TXT文件存在或其它oligo 不能直接open的文件格式,如word文件.html格式,这个功能就显得很有用了。
在相应文件中复制序列后在序列展示窗口粘贴,oligo 会自动去除非碱基字符。
当序列输入或粘贴完成后,点击Accept/Discard菜单中的Accept浮动命令,即可进入引物设计模式。
3,如果序列已经保存为Seq格式或者FASTA,GenBank格式时,oligo 就可以直接打开序列文件。
点击File菜单中的“Open”浮动命令,找到所需文件,打开即可。
进入引物设计模式后,oligo一般会弹出三个窗口,分别是6-碱基频率窗口,碱基退火温度窗口以及序列内部碱基稳定性窗口,其中的退火温度窗口是我们引物设计的主窗口,其它的两个窗口则在设计过程中起辅助作用,比如6-碱基频率窗口可以使我们很直观地看到所设计引物在相应物种基因组中的出现频率,如果我们的模板是基因组DNA或混合DNA时,该信息就显得有用了,而内部稳定性窗口则可以显示引物的5’端稳定性是否稍高于3’端等。
一,普通引物对的搜索:以Mouse 4E(cDNA序列)为例。
我们的目的是以Mouse 4E(2361 bp)为模板,设计一对引物来扩增出600-800bp长的PCR产物。
引物设计软件Oligo使用方法介绍荧光定量PCR仪

引物设计软件Oligo使用方法介绍荧光定量PCR仪|荧光定量PCR基因扩增仪作为目前最好、最专业的引物设计软件,Oligo的功能很强,在这里我们介绍它的一些主要功能,如:普通引物对的搜索、测序引物的设计、杂交探针的设计以及评估引物对质量等等。
在正式进行引物设计前,我们首先面临的一个任务就是向Oligo程序导入模板序列,根据不同的实验情况,导入模板有三种方法:1、直接用键盘输入:a、点击file菜单中的New Sequence 浮动命令,或直接点击工具栏中的New Sequence命令,进入序列展示窗口;b、此时即可键入DNA序列;c、如果需要的话,Oligo提供碱基回放功能,在边键入时边读出碱基,防止输入错误。
点击Edit菜单中的“Readback on”即可。
2、利用复制和粘贴:当我们序列已经作为TXT文件存在或其它oligo不能直接open的文件格式,如word文件。
html 格式,这个功能就显得很有用了。
在相应文件中复制序列后在序列展示窗口粘贴,oligo会自动去除非碱基字符。
当序列输入或粘贴完成后,点击Accept/Discard菜单中的Accept浮动命令,即可进入引物设计模式。
3、如果序列已经保存为Seq格式或者FASTA,GenBank格式时,oligo就可以直接打开序列文件。
点击File菜单中的“Open”浮动命令,找到所需文件,打开即可。
进入引物设计模式后,oligo一般会弹出三个窗口,分别是6-碱基频率窗口,碱基退火温度窗口以及序列内部碱基稳定性窗口,其中的退火温度窗口是我们引物设计的主窗口,其它的两个窗口则在设计过程中起辅助作用,比如6-碱基频率窗口可以使我们很直观地看到所设计引物在相应物种基因组中的出现频率,如果我们的模板是基因组DNA或混合DNA 时,该信息就显得有用了,而内部稳定性窗口则可以显示引物的5‘端稳定性是否稍高于3’端等。
一、普通引物对的搜索:以Mouse 4E(cDNA序列)为例。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
作为目前最好、最专业的引物设计软件,Oligo的功能很强,在这里我们介绍它的一些主要功能:如:普通引物对的搜索、测序引物的设计、杂交探针的设计以及评估引物对质量等等。
在正式进行引物设计前,我们首先面临的一个任务就是向Oligo程序导入模板序列,根据不同的实验情况,导入模板有三种方法:
1,直接用键盘输入:
a,点击file菜单中的New Sequence 浮动命令,或直接点击工具栏中的New Sequence 命令,进入序列展示窗口;
b,此时即可键入DNA序列;
c,如果需要的话,Oligo提供碱基回放功能,在边键入时边读出碱基,防止输入错误。
点击Edit菜单中的“Readback on”即可。
2,利用复制和粘贴:当我们序列已经作为TXT文件存在或其它oligo不能直接open的文件格式,如word文件.html格式,这个功能就显得很有用了。
在相应文件中复制序列后在序列展示窗口粘贴,oligo会自动去除非碱基字符。
当序列输入或粘贴完成后,点击Accept/Discard菜单中的Accept浮动命令,即可进入引物设计模式。
3,如果序列已经保存为Seq格式或者FASTA,GenBank格式时,oligo就可以直接打开序列文件。
点击File菜单中的“Open”浮动命令,找到所需文件,打开即可。
进入引物设计模式后,oligo一般会弹出三个窗口,分别是6-碱基频率窗口,碱基退火温度窗口以及序列内部碱基稳定性窗口,其中的退火温度窗口是我们引物设计的主窗口,其它的两个窗口则在设计过程中起辅助作用,比如6-碱基频率窗口可以使我们很直观地看到所设计引物在相应物种基因组中的出现频率,如果我们的模板是基因组DNA或混合DNA时,
该信息就显得有用了,而内部稳定性窗口则可以显示引物的5’端稳定性是否稍高于3’端等。
一,普通引物对的搜索:
以Mouse 4E(cDNA序列)为例。
我们的目的是以Mouse 4E(2361 bp)为模板,设计一对引物来扩增出600-800bp长的pcr产物。
1,点击“Search“菜单中的”For Primers and Probes“命令,进入引物搜索对话框;2,由于我们要设计的是一对PCR引物,因此正、负链的复选框都要选上,同时选上Compatible pairs。
在Oligo默认的状态下,对此引物对的要求有:a,无二聚体;b,3’端高度特异,GC含量有限定,d,去除错误引发引物等。
3,剩下的工作是确定上、下游引物的位置及PCR产物的长度以及引物设计参数。
①单击:“search Ranges”按钮,弹出“Search Ranges”对话框。
输入上游引物的范围:1-2000,下游引物的位置:100-2300;PCR产物的长度600-800bp。
②单击“Paramaters”按钮进入“Search Parameters”对话框,对话框种分三个活页,分别是:不同设定,参数以及更多参数。
③在“普通设定”窗口,为我们提供了对引物非常直观的设定方法,从高到低分六个等级,最后还有一个用户定制选项。
④当我们对引物的各种参数的含义及应该设定多大值并不是特别清楚时,就可以直接设定Very high/High等来完成对引物设计参数的设定。
⑤当我们选中“Automatically Change String”后,Oligo会在引物搜索过程中:如果在高等级设定中无法找到引物对时自动降低一个定级来进行搜索,知道找到引物对。
在设计反向PCR引物对时,就选中“Inverse PCR”复选框。
⑥我们还可以让引物的长度可以改变,以适应设定的Tm值或PE?(Prime Effitions,引发效率)。
也可以限定所选引物对的最大数目。
⑦在“Parameters”窗口中,实际上需要我们改动的只有引物的长度,根据试验的要求作相应的改变,如23nt。
其他参数就使用Oligo的默认值,一般无需改变。
在“More Parameters“窗口中,一般也无需作任何改变,直接用Oligo的默认值。
4,点击“确认”-“Ok”进入搜索窗口,再次单击“OK”后,Oligo自动完成引物对的搜索,并出现一个搜索结果窗口。
显示出得到的引物对数目。
5,点击“Ok”,出现“PrimerPairs”窗口,在窗口中列出了7对引物的简单信息,引物位置,产物长度,最佳退火温度及GC含量。
单击“Sort”按钮,可以按产物长度,最佳退火温度及GC含量从小到大或从大到小的顺序排列。
6,点击任意一对引物,在旁边马上弹出一个叫“PCR”的窗口,在窗口中我们可以看到这对引物在模板中的大概位置,最佳退火温度(该温度可以直接应用于我们实际PCR中的第二步退火)。
另外还有引物的Tm值,GC含量,引发效率,同时还计算出了产物的Tm值于引物Tm值的差异以及引物间Tm值的差异。
一般我们尽量保持前者在20度以内,后者在5-6度以内。
点击不同的引物对时,PCR窗口内容同时作相应的改变。
7,要想了解引物的详细信息,如二聚体,发卡结构形成情况、组成、Tm值和错误引发位点等,这时只需点击“Analyze”菜单中的相应命令,就可以分别得到相关信息。
例如点击“Duplex Formation”,我们就可以得到上游引物间,下游引物间及上下游引物间形成二聚体的情况。
同理,“Hairpin Formation”,“Composition and Tm”,“False Priming Sites”等命令就可以显示出相关信息。
所有这些分析的结果都有助于我们从Oligo搜索得到的多对引物中选择最佳的引物对。
需要说明的是,1,如果一次搜索得到的引物对太多时,我们可以适当提高选定的条件和规
定更合适的搜索范围来减少引物对数;2,引物一般尽量不要在3’端或是离3’端太近的位置形成二聚体,同时二聚体的自有能绝对值尽量小于10;3,对于错误引发,在普通PCR 中,如果引发效率在160 points以上时,就可能出现杂带,在测序反应中,错误引发效率则应该严格控制在120 points以下。
8,Oligo中每对引物的详细信息可以直接导出为一个文本文件:
首先点击“File”菜单中的“Print/Save Options”,在出现的对话框中的普通设定选中“Selected”;在“Analyze I”中选择需要保存的信息,在“Analyze II”中选中PCR。
单击确定按钮退出,这时再次点击“File”菜单,Save浮动命令中的“Data Save as”,选择路径及文件名即可。
二,测序引物的设计:
在oligo中,测序引物设计过程与普通引物设计大部分都很相似。
还是以Mouse 4E为例,假如我们要在600-800bp的位置设计一条测序引物。
Open-Search
在“Search”窗口中,选中“Sequece Primer”,同时去除负链Search的选中
在“Search Range”窗口,输入正链的600-800bp
在“Parameters”中选定very high ,引物长度改为18bp
结果得到11条测序引物,与普通引物同理,根据其详细信息就可以挑选到一条最佳的测序引物。
三,探针的设计:
探针的设计与测序引物设计基本相同,只需使“Search”窗口的探针设计选中,改变探针的长度就可以。
四,评估引物对:
我们在各种文献中查到的引物,如果直接进行PCR,往往很难重复别人的实验结果。
这时就想到,是不是引物的质量有问题?能不能用软件来对这些问题引物进行一些分析呢?答案时肯定的。
1,点击File菜单中的New命令;
2,在“Edit New Sequence”的窗口中用键盘输入上游引物;
3,如果该引物的首位置不是1的话,可以在“Edit”窗口中输入新的5’端位置数字,如20;
4,点击Accept/Discard菜单的Accept命令;
5,如果引物序列长度不同于当前的引物的话,可以从“Change”菜单中改变当前的引物长度;
6,选取当前序列为上游引物(点击“upper”按钮);
7,从Edit菜单中选取“Lower Primer”命令,在Edit Lower窗口中输入下游引物的序列;8,在Edit窗口的上角处,输入相应的5’位置;
9,选取“Accept and Quit”命令;
如果想让程序给出最佳退火温度,在此时的对话框中输入PCR产物的长度以及GC含量所占百分比,一般哺乳动物的cDNA序列中GC大约占44%。
10,点击OK就可以在“Analyze”菜单中完成各种分析了。
