第七章序列特征分析分解
人大版时间序列分析基于R(第2版)习题答案
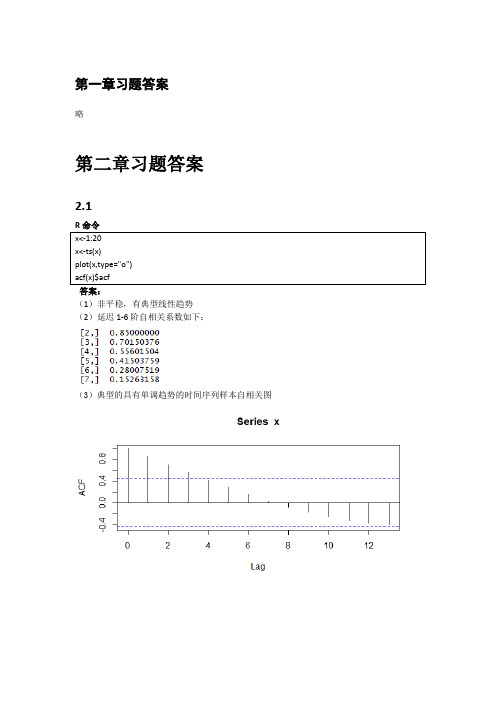
第一章习题答案略第二章习题答案2.1答案:(1)非平稳,有典型线性趋势(2)延迟1-6阶自相关系数如下:(3)典型的具有单调趋势的时间序列样本自相关图2.2(1)非平稳,时序图如下(2)1-24阶自相关系数如下(3)自相关图呈现典型的长期趋势与周期并存的特征2.3R命令答案(1)1-24阶自相关系数(2)平稳序列(3)非白噪声序列Box-Pierce testdata: rainX-squared = 0.2709, df = 3, p-value = 0.9654X-squared = 7.7505, df = 6, p-value = 0.257X-squared = 8.4681, df = 9, p-value = 0.4877X-squared = 19.914, df = 12, p-value = 0.06873X-squared = 21.803, df = 15, p-value = 0.1131X-squared = 29.445, df = 18, p-value = 0.04322.4答案:我们自定义函数,计算该序列各阶延迟的Q统计量及相应P值。
由于延迟1-12阶Q统计量的P值均显著大于0.05,所以该序列为纯随机序列。
2.5答案(1)绘制时序图与自相关图(2)序列时序图显示出典型的周期特征,该序列非平稳(3)该序列为非白噪声序列Box-Pierce testdata: xX-squared = 36.592, df = 3, p-value = 5.612e-08X-squared = 84.84, df = 6, p-value = 3.331e-162.6答案(1)如果是进行平稳性图识别,该序列自相关图呈现一定的趋势序列特征,可以视为非平稳非白噪声序列。
如果通过adf检验进行序列平稳性识别,该序列带漂移项的0阶滞后P值小于0.05,可以视为平稳非白噪声序列Box-Pierce testdata: xX-squared = 47.99, df = 3, p-value = 2.14e-10X-squared = 60.084, df = 6, p-value = 4.327e-11(2)差分序列平稳,非白噪声序列Box-Pierce testdata: yX-squared = 22.412, df = 3, p-value = 5.355e-05X-squared = 27.755, df = 6, p-value = 0.00010452.7答案(1)时序图和自相关图显示该序列有趋势特征,所以图识别为非平稳序列。
第七章时间序列分析答案

第七章时间数列分析一、填空题1、时间指标数值2、逐期增长量累计增长量3、增长水平(或增长量)发展速度4、本期水平去年同期水平5、年距发展速度 1(或100%)6、几何平均法方程法7、同季(月)平均法趋势与季节模型法8、平均季节比重法平均季节比率法9、报告期水平基期水平10、序时平均数(或动态平均数)平均数11、和差12、季节变动长期趋势13、逐期增长量环比增长速度14、长明显1-5 A C C A D 6-10 A B A D B三、多选题1、CDE2、ABDE3、ABCE4、ACDE5、BDE6、BD7、ABCD8、ACE9、AE 10、ACE四、简答题1、序时平均数与一般平均数的异同。
答:(1)相同之处。
二者都是将具体数值抽象化,用一个代表性的数指来代表总体的一般水平。
(2)不同之处。
①计算的依据不同。
一般平均数是根据变量数列计算的,而序时平均数则是根据时间数列计算的;②对比的指标不同。
一般平均数是总体标志总量与总体单位总量对比的结果,而序时平均数则是时间数列各期发展水平的总和与时期项数对比的结果;③说明的问题不同。
一般平均数说明现象在同一时间、不同空间上所达到的一般水平,而序时平均数则说明现象在同一空间、不同时间上所达到的一般水平。
2、时期数列与时点数列的区别。
答:①时期数列中的指标值为时期数,时点数列中的指标值为时点数;②时期数列中的指标值具有可加性,而时点数列中的指标值则不具有可加性;③时期数列中指标值的大小与时间间隔的长短有直接关系,而时点数列中指标值的大小与时间间隔的长短则没有直接关系;④时期数列中的指标值是通过连续调查取得的,而时点数列中的指标值则是通过一次性调查取得的。
3、时间数列的编制原则。
答:(1)基本原则:保持数列中的各项指标数值具有可比性。
(2)具体原则:①时间长短统一;②总体范围统一;③指标口径统一;④计算方法统一;⑤计量单位统一。
4、计算和应用平均速度应注意的问题。
第七章.时间序列(平均发展速度)

128.9 128.9 28.9 28.9
114.9 148.1 14.9 48.1
112.5 166.6 12.5 66.6
108.1 180.2
8.1 80.2
108.1 194.8
8.1 94.8
三、平均发展速度和平均增长速度
1.平均发展速度是现象环比发展速度的序时平 均数。
2.平均增长速度是现象环比增长速度的序时平 均数,可以根据以下公式计算:
解:已知a0 15, a1 a2 a3 60, n 3,
则X 3 X 2 X n ai a0 0,即 i 1
3
X
2
X
X
4 0,解得X
1.151
平均发展速度的计算
两种方法的比较:
几何平均法:
an
n
a0 X G
方程法:X n X n1 X 2 X n ai a0 i 1
繁荣 116
115ቤተ መጻሕፍቲ ባይዱ
拐点 114
113 112 111 110 109 108 107 106 105
104 103 102 101 100
衰退 拐点
萧条 拐点
繁荣 拐点
复苏 拐点
经济周期:循环性变动 年份
时间数列的组合模型
(1)加法模型:Y=T+S+C+I
计量单位相同 的总量指标
对长期趋势 产生的或正 或负的偏差
定基增长速度=定基发展速度-1 环比增长速度=环比发展速度-1 年距增长速度=年距发展速度-1
环比增长速度 定基增长速度 年距增长速度
ai ai1 ai 100﹪
ai 1
ai 1
ai a0 ai 100﹪
a0
时间序列分析课件-07-ARIMA模型、疏系数模型、季节模型

xt 0 1t at
• 考察一阶差分后序列和二阶差分序列 的平稳性与方差
比较
• 一阶差分
– 平稳
xt xt xt1
1 at at1 – 方差小
• 二阶差分(过差分)
– 平稳
2 xt xt xt1 at 2at1 at2
– 方差大
Var(xt ) Var(at at1)
• 参数估计
(1 0.44746 B 0.28132 B4 )(1 B)(1 B4 )xt t
模型检验
残差白噪声检验
参数显著性检验
延迟 阶数
2统 计量
P值
待估 t 统
参数 计量
P值
6
2.09 0.7191 1
12 10.99 0.3584 4
5.48 <0.0001 -3.41 <0.0001
2 2
Var(2xt ) Var(at 2at1 at2 )
6 2
ARIMA模型
• ARIMA模型结构 • ARIMA模型性质 • ARIMA模型建模 • ARIMA模型预测 • 疏系数模型 • 季节模型
ARIMA模型结构
• 使用场合
– 差分平稳序列拟合
• 模型结构
( B) d
E( t )
Tt 0 1 xtm l xtlm
• 简单/复杂季节模型 • X-11 • etc
• AR • MA • ARMA • WN • etc
3.考虑残差
获 得 观 察 值 序
Y
Y
平稳性 检验
白噪声 检验
分 析
结
N
束 N
列
差分 运算
拟合
ARMA 模型
序列数据的特征提取方法及在基因组学研究方面的应用分析

序列数据的特征提取方法及在基因组学研究方面的应用分析引言:基因组学是研究生物体基因组结构、功能和调控的学科,其中序列数据的处理与分析是关键的一环。
随着高通量测序技术的不断发展,获取到的序列数据呈现急剧增加的趋势。
如何从庞大的序列数据中提取有用的特征信息并进行深入的分析成为了基因组学研究领域中的重要课题。
本文将介绍序列数据的特征提取方法,并重点探讨其在基因组学研究方面的应用和意义。
一、序列数据的特征提取方法1.1 k-mer特征:k-mer是指序列中连续k个碱基的组合。
k-mer特征提取是一种广泛应用于基因组学研究的方法。
通过统计序列中所有可能的k-mer的出现频率,可以得到一个特定长度的特征向量。
这些特征向量可以用于比较和分类不同的生物组织、物种或环境。
k-mer特征提取方法简单高效,可应用于多种序列数据类型,如基因序列、转录组数据、代谢组数据等。
1.2 Motif特征:Motif是指在DNA或蛋白质序列中的重复模式或保守序列。
Motif特征提取是一种常用于分析基因组和蛋白质序列的方法。
通过使用计算机算法和模式识别技术,可以从序列数据中提取出具有生物学意义的Motif。
Motif特征在识别转录因子结合位点、预测启动子和剪接位点等方面起着重要作用。
1.3 突变特征:突变是指基因组中发生的DNA序列的变化。
突变特征提取是一种用于鉴定和分析基因组变异的方法。
通过比较多个个体或物种的序列数据,可以发现其中存在的突变。
突变特征对于研究个体之间的差异以及相关疾病的遗传基础具有重要的意义。
二、序列数据特征提取方法在基因组学研究中的应用2.1 基因表达谱的分析:基因表达谱是指在特定条件下基因表达的水平。
通过对转录组数据的特征提取,可以得到不同基因的表达模式,从而揭示基因在不同生理和病理过程中的功能。
例如,通过对肿瘤组织和正常组织的转录组数据进行特征提取和比较,可以发现与癌症相关的基因。
2.2 DNA甲基化的分析:DNA甲基化是指DNA分子上的甲基基团添加或拆除的过程,对基因的转录和表达有重要影响。
时间序列的分解分析

时间序列的分解分析一、时间序列分解分析的原理时间序列分解分析的原理是基于时间序列数据的两个基本特征:长期趋势和短期季节变动。
长期趋势是指时间序列数据在长期内呈现的整体上升或下降趋势,而短期季节变动则是指时间序列数据在每个季节内的周期性变动。
时间序列分解分析将时间序列数据分解成长期趋势、季节性、循环和随机成分,以便更好地理解和分析时间序列数据。
二、时间序列分解分析的步骤时间序列分解分析的步骤通常包括以下几个步骤:数据获取、数据处理、分解分析、模型建立和预测。
1. 数据获取:从相应的数据源获取需要分析的时间序列数据。
对于涉及的时间序列数据,通常需要有一定的历史数据,以便进行分析和建模。
2. 数据处理:对获取的时间序列数据进行数据处理,例如数据清洗、缺失值填补、异常值处理等。
这一步骤的目的是确保数据的准确性和完整性。
3. 分解分析:对经过数据处理的时间序列数据进行分解分析。
通常使用的方法有移动平均法、指数平滑法和加法模型等。
这些方法可以将时间序列数据分解成长期趋势、季节性、循环和随机成分。
4. 模型建立:基于分解分析的结果,建立合适的模型。
常用的模型有ARIMA模型、指数平滑模型、回归分析等。
模型的选择需要根据具体的时间序列数据和分析目的来确定。
5. 预测:利用建立的模型对未来的时间序列数据进行预测。
根据建立的模型,可以得到未来一段时间内的长期趋势、季节性、循环和随机成分的预测值,从而提供决策参考。
三、实例分析为了更好地理解时间序列分解分析的步骤和应用,我们以某公司销售额数据为例进行分析。
假设该公司的销售额数据具有长期增长趋势和季节性变动。
1. 数据获取:从公司的销售系统中获取过去几年的销售额数据,包括每个月的销售额。
2. 数据处理:对获取的销售额数据进行数据清洗,排除异常值和缺失值。
3. 分解分析:利用加法模型对销售额数据进行分解分析。
加法模型将销售额数据分解成长期趋势、季节性、循环和随机成分。
通过分析过去几年的销售额数据,可以得到相应的分解结果。
特征分解和奇异值分解的真正意义
特征分解和奇异值分解的真正意义特征分解和奇异值分解在机器学习的应用中经常出现,在学习线性代数的时候也学习过。
线性代数学完之后,之后去按照步骤去求解特征值和特征向量,也没搞明白特征值和特征向量究竟有什么作用。
这篇文章的主要内容包括:1、什么是特征分解2、什么是奇异值分解3、如何求解特征值和特征向量4、特征值和特征向量有什么意义一、特征分解特征分解(eigendecomposition):是使用最广的矩阵分解之一,通过特征分解可以将矩阵分解成一组特征值和特征向量。
方阵A的特征向量(eigenvector)是指与A相乘后相当于对该向量进行缩放的非零向量v其中v就是方阵A的特征向量,λ就是A的特征值。
如果v是A 的特征向量,那么任何缩放后的向量s*v(s为任意实数,且不为0)也是A的特征向量。
同时sv和v具有相同的特征值。
所以,通常情况下我们都只考虑单位特征向量。
通过将矩阵分解成为特征值和特征向量,来帮助我们分析矩阵。
二、奇异值分解奇异值分解(singular value decomposition,SVD):是将矩阵分解成为特征值和特征向量的另一种方法,通过奇异值分解,可以将矩阵分解为奇异向量(singular vector)和奇异值(singular value)。
通过奇异值分解,我们可以得到一些与特征分解相同类型的信息。
而且,奇异值分解的应用非常广泛,如推荐系统、图片压缩等。
每一个实数矩阵都有一个奇异值分解,但不一定有特征分解。
非方阵的矩阵没有特征分解,此时我们只能使用奇异值分解。
奇异值分解,可以将矩阵A分成三个矩阵的乘积:假设A是一个m×n的矩阵,那么U是一个m×m的矩阵,D是一个m×n的矩阵,V是一个n×n的矩阵。
其中,矩阵U和V都是正交矩阵,而矩阵D是对角矩阵。
矩阵D不一定是方阵。
对角矩阵D对角线上的元素就是矩阵A的奇异值(singular value)。
第七章-盈余特征和盈余持续性
稳健性原则和盈余的持续性
稳健性原则对研发支出的影响:
研发支出的特性; 稳健性原则要求企业把本期研发支出直接费用
化,不能记为资产供以后期间分摊。
36
稳健性原则和盈余的持续性
稳健性原则对研发支出的影响:
影响结果: 研发支出当期的盈余被低报,产生盈余储备, 研发支出以后期间的盈余则被高报。
32
稳健性原则和盈余的持续性
稳健性原则对盈余的时间序列特征影响显 著的例子:
福建高速 峨眉山 美国礼来公司
33
稳健性原则和盈余的持续性
稳健性原则影响的主要项目:
存货; 研发支出; 广告支出;
34
稳健性原则和盈余的持续性
稳健性原则对存货的影响:
存货计价: 后进先出法: 2007年新会计准则已取消该法。 先进先出法: 通货膨胀时期和通货紧缩时期有什么不同 的影响?
我国上市公司的情况:
比如:
TCL 万科
25
应计盈余、现金盈余与盈余持续性
思考:
在运用这一原则判断企业盈余的持续性时,是 否需要注意哪些前提条件?
… …
26
应计盈余、现金盈余与盈余持续性
在大多数时候,对于大多数企业而言,其 盈利模式、盈利能力是平稳持续发展的。 也只有在这种情况下,我们能有较大把握 利用过去的数据来预测未来盈利状况。
依据应计制方法计算出来的盈余包含:
现金盈余:已经收到了现金的盈余; 应计盈余:企业有权利在未来收到现金的盈余。
14
应计盈余、现金盈余与盈余持续性
应计盈余的计算:
应计盈余是已确认的盈余中未收到现金的部分。 有现金流量表:
应计盈余 净利润 经营活动现金流
15
应计盈余、现金盈余与盈余持续性
核酸序列特征分析
核酸序列特征分析核酸序列特征分析是生物信息学研究中重要的一个方面。
它可以帮助我们更深入地理解基因组及基因表达研究。
本文旨在介绍核酸序列特征分析,其中包括核酸序列分析、核酸序列特征抽取和质粒抽取等内容。
首先,介绍核酸序列分析,其中包括特征分类、序列特征检测、序列分类和序列比对等。
核酸特征分类是将核酸序列分为有用的和无用的,从而排除噪声。
核酸序列特征检测包括对不同类型的基因、基因组表达、基因功能和结构等特征的检测,以及比较不同物种序列或不同基因组结构的检测。
核酸序列分类是用特征抽取技术分析序列长度,以确定序列的分类及特征。
序列比对是比较两个或多个序列的相似性,以发现可能的相似性或共同特征。
其次,介绍核酸序列特征抽取。
它分为特征抽取和质粒抽取两大类。
特征抽取的主要目的是抽取出序列的非特定特征,比如k-mer特征,基于序列单位的反向字典学习(RLD)等方法。
质粒抽取的目的是抽取出序列以及其表达周围的特定特征,比如突变、位点突变、基因连接等。
特征抽取是对序列的概括,抽取出重要的特征,而质粒抽取是对序列表达的概括,可以捕捉到序列的精细结构信息。
最后,介绍核酸序列特征分析的一些应用。
一方面,核酸序列特征分析可以用于揭示基因组结构和功能特征。
例如,可以利用序列比对技术对不同物种序列进行对比,揭示出不同物种的关键基因。
另一方面,核酸序列特征分析也可以用于揭示表达调控机制。
例如,可以用特征分类和序列特征抽取技术,结合表达评价结果,探索基因表达调控的内在机制。
综上所述,核酸序列特征分析是生物信息学研究中重要的一个方面。
它可以用来探索基因组结构和功能特征,揭示表达调控机制,改进基因调控机制,为临床实验提供分析指导,并帮助我们更加深入地了解基因组研究和基因表达研究。
因此,核酸序列特征分析的研究将给生物信息学领域带来许多新的机会。
生物信息学讲义——序列特征分析
生物信息学讲义——序列特征分析生物信息学是一门应用生物学、计算机科学和统计学等多学科知识的交叉学科。
其中,序列特征分析是生物信息学中的一个重要研究领域。
它涉及到对生物学序列的各类特征进行提取、分析和解释的过程,可以用于从序列数据中推断生物功能、结构和进化等信息。
序列特征分析的首要任务是对生物学序列进行特征提取。
常见的生物学序列包括DNA序列、RNA序列和蛋白质序列等。
这些序列通常以一串字符的形式保存,比如以“A”、“T”、“G”、“C”表示DNA序列中的碱基。
通过使用序列分析工具,可以将这些字符转化为序列特征的数值表示,以方便后续的计算和分析。
在序列特征分析中,常用的特征包括序列长度、碱基或氨基酸组成、序列重复性、序列保守性、二级结构等。
其中,序列长度是最基本的特征,可以直接从序列中读取得到。
碱基或氨基酸组成是指序列中各类碱基或氨基酸的相对含量。
序列重复性是指序列中出现的重复单元的种类和数量。
序列保守性是指序列在不同物种或不同基因中的保守程度,用于推断序列的功能和进化关系。
二级结构是指蛋白质序列中各个氨基酸的空间排列方式,用于推断蛋白质的结构和功能。
在实际应用中,序列特征分析可以帮助研究人员理解生物系统的结构和功能。
例如,通过分析DNA序列中的启动子、编码区和调控元件等特征,可以推断基因的结构和转录调控机制。
通过分析蛋白质序列中的保守模体和功能域等特征,可以推断蛋白质的功能和进化关系。
通过分析RNA序列的二级结构和稳定性等特征,可以推断RNA的折叠方式和功能。
为了完成序列特征分析的任务,研究人员通常会借助各种生物信息学工具和算法。
比如,BLAST(Basic Local Alignment Search Tool)是一种常用的序列比对工具,可以通过比对已知序列库中的序列,从而推断未知序列的一些特征和功能。
HMM(Hidden Markov Model)是一种常用的序列模型,可以用于推断未知蛋白质序列的二级结构和功能。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
起始外显子
终止外显子
二、CpG岛— —CpG islands
CpG岛是指DNA序列上的一个区域,此区域含有大 量相联的胞嘧啶(C)、鸟嘌呤(G),以及使两者相连 的磷酸酯键(p)。CpG岛的概念是Gardiner-garden和 Fromner于1987年提出的,基因中平均每100 Kb即可出现。
及基因结构信息的开放式在线资源,尤其适用于脊 椎动物、拟南芥和玉米等真核生物。 GENSCAN的网址为:
http:///GENSCAN.html
GENSCAN在线操作页面
用GENSCAN预测AC002390序列的基因/外显子
用GENSCAN预测AC002390序列的基因/外显子的位置图
核苷酸总是GT,并且其3'端的最后两个核苷酸总是AG,即:
5'-GT ……AG-3',这个规律有助于真核生物开放阅读框的识 别。
利用GENSCAN识别基因开放阅读框
GENSCAN是美国麻省理工学院的Chris Burge 于1997年开发成功的人类(或脊椎动物)基因预测
软件,它是根据基因组DNA序列来预测开放阅读框
操纵子模型结构
原核生物大多数基因表达调控是通过操纵子机制实现的。
所谓操纵子通常由调节基因、启动子、操纵基因以及2个以 上的编码序列(结构基因)在原核生物基因组中成簇串联组 成。其中结构基因的表达受到操纵基因的调控。调节基因能 产生作用于操纵基因的阻遏物(一种蛋白质),操纵基因靠 近它所控制的结构基因,阻遏物与操纵基因的结合能阻止结 构基因的转录。
一、开放阅读框ORF—open reading frame
开放阅读框指的是从5'端开始翻译起始密码子(ATG)
到终止密码子(TTA、TAG、TGA)的蛋白质编码碱基序列。 每个序列都有6个可能的开放阅读框,其中3个开始于第1、2、 3个碱基位点并沿着给定序列的5'→ 3'的方向进行延伸,而另 外的3个开始于第1、2、3个碱基位点但沿着互补序列的5'→ 3'的方向进行延伸。在开始这项工作之前,我们并不知道 DNA双链中哪一条单链是编码链,也不知道准确的翻译起始 点在何处,由于每条链都有3种可能的开发阅读框,2条链共 计6种可能的开放读框,我们的目的就是从这6个可能的开放 阅读框中找出一个正确的开放阅读框。根据这个开放阅读框 翻译得到的氨基酸序列才是真正表达的蛋白质产物。
真核生物的开放阅读框
真核生物的开放阅读框不仅含有编码蛋白的外显子 (exon),而且还有内含子(intron),并且内含子将开放 阅读框分割为若干个小片段。开放阅读框的长度变化范围非 常大,因此真核生物的基因预测远比原核生物困难。但是, 在真核生物的开放阅读框中,外显子与内含子之间的连接绝 大部分情况下满足GT-AG规律:内含子序列 5' 端的起始两个
第七章
序列特征分析
Analysis of Sequence Characterristics
第一节 引言
Section 1 Introduction
一、基因结构
基因的概念是随着遗传学、分子生物学、 生物化学等领域的发展不断完善的。从分子 生物学角度来看,基因是负载特定生物遗传 信息的DNA分子片段,在一定的条件下能够 表达这种遗传信息,产生特定的生理功能。
蛋白质的二级结构
H表示螺旋 G表示3-螺旋 S代表转向
E表示折叠 I表示π螺旋
B表示β桥 T表示氢键转角
蛋白质空间结构
蛋白质的生物学功能在很大 程度上取决于蛋白质的空间结构, 但蛋白质的空间结构又取决于蛋白 质一级结构中的氨基酸组成和排列 顺序,蛋白质结构构象多样性导致 了不同的生物学功能。蛋白质分子 只有处于它自己特定的空间结构情况下,才能获得它特定的生 物活性,空间结构稍有破坏,就很可能会导致蛋白质生物活性 的降低甚至丧失,因为它们的特定的结构允许它们结合特定的 配体分子。知道了基因密码,科学家们可以推演出组成某种蛋 白质的氨基酸序列,却无法绘制蛋白质空间结构。因而,揭示 人类每一种蛋白质的空间结构,已成为后基因组时代的制高点, 这也是结构基因组学的基本任务。
对DNA序列和蛋白质序列进行序列特征分析, 能够使我们从分子层次上了解基因的结构特点,
了解与基因表达调控相关的信息,了解 DNA序列与
蛋白质序列之间的Βιβλιοθήκη 码,了解蛋白质序列与蛋白质空间结构之间的关系和规律,为进一步研究了
解蛋白质功能与蛋白质结构之间的关系提供理论
依据。
第二节 DNA序列特征分析
Section 2 Analysis of DNA Sequence Characteristics 分析DNA序列,除了进行序列比对之外,更重要的工作 是从序列中找到基因及其表达调控信息。寻找基因的工作有 两个:一是识别与基因相关的特殊序列信号,如启动子、起 始密码子,通过信号识别大致确定基因所在的区域;二是预 测基因的编码区域,或预测外显子所在的区域。在此基础上, 结合两个方面的结果确定基因的位置和结构。绝大部分基因 表达调控信息隐藏在基因序列的上游区域,在组成上具有一 定的特征,可以通过序列分析识别这些特征。
真核生物基因结构:
一个完整的真核生物基因,不但包括编码区域,还包括 5'端和3'端两侧长度不等的特异性序列,虽然这些序列不编
码氨基酸,却在基因表达的过程中起着重要的作用。所以, 严格的“基因”这一术语的分子生物学定义是:产生一条多 肽链或功能RNA所必须的全部核苷酸序列。
二、蛋白质结构
蛋白质是一种生物大分子,蛋白质中相邻的氨基 酸通过肽键形成一条伸展的肽链,这条链称为蛋白质
原核生物基因结构:
一个完整的原核基因结构是从基因的5'端启动子区域 开始,到3'端终止区域结束。基因的转录开始位置由转录 起始位点确定,转录过程直至遇到转录终止位点结束,转 录的内容包括5'端非翻译区、开放阅读框及3'端非翻译区。 基因翻译的准确起止位置由起始密码子和终止密码子决定, 翻译的对象即为介于这两者之间的开放阅读框ORF。
的一级结构,不同蛋白质其肽链的长度不同,肽链中
不同氨基酸的组成和排列顺序也各不相同。肽链上的
氨基酸残基形成局部的二级结构,各种二级结构在空
间卷曲折叠形成特定的三维空间结构。有的蛋白质由 多条肽链组成,每条肽链称为亚基,亚基之间又有特
定的空间关系,称为蛋白质的四级结构。
蛋白质的一级结构
蛋白质的一级结构决定二级结构 蛋白质的二级结构决定三级结构
