核酸标记方法
常用核酸探针标记方法
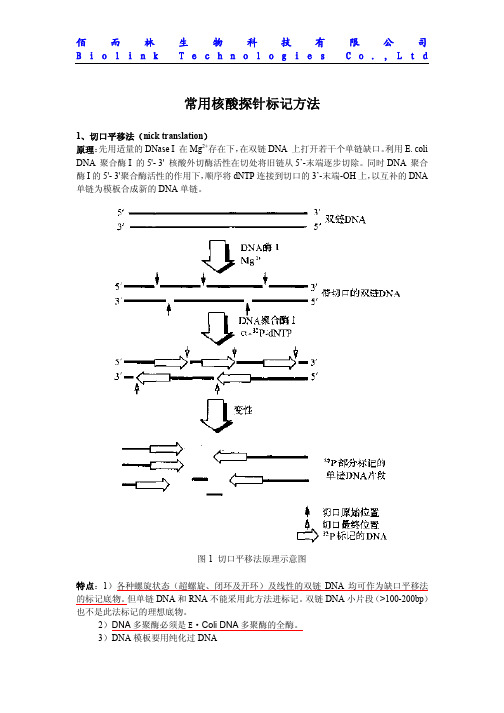
常用核酸探针标记方法1、切口平移法(nick translation)原理:先用适量的DNase I 在Mg2+存在下,在双链DNA 上打开若干个单链缺口。
利用E. coli DNA 聚合酶I 的5'- 3' 核酸外切酶活性在切处将旧链从5’-末端逐步切除。
同时DNA 聚合酶I的5'- 3'聚合酶活性的作用下,顺序将dNTP连接到切口的3’-末端-OH上,以互补的DNA 单链为模板合成新的DNA单链。
图1 切口平移法原理示意图特点:1)各种螺旋状态(超螺旋、闭环及开环)及线性的双链DNA均可作为缺口平移法的标记底物。
但单链DNA和RNA不能采用此方法进标记。
双链DNA小片段(>100-200bp)也不是此法标记的理想底物。
2)DNA多聚酶必须是E·Coli DNA多聚酶的全酶。
3)DNA模板要用纯化过DNA2、随机引物法(random priming)原理:将待标记的DNA探针片段变性后与随机引物一起杂交,然后以此杂交的寡核苷酸为引物,大肠杆菌DNA聚合酶I大片段(E·Coli DNA polymerase I Klenow Fragment)的催化下,合成与探针DNA互补的DNA链。
当反应液中含有标记的dNTP时,即形成标记的探针。
图2 随机引物标记法原理示意图特点:1)除了能进行双链DNA标记外,也可用于单链DNA和RNA探针的标记。
2)所得到的标记产物是新合成的DNA单链,而所加入的DNA片段本身并不能被标记。
3)新形成的标记DNA单链的长度与加入寡核苷酸引物的量成反比,因为加入的寡核苷酸数量越多,合成起点也越多,得到的片段的长度也越短。
按标准方法得到的标记产物长度一般为200-400bp。
3、末端标记与切口平移法和随机引物法不同,DNA末端标记法并不将DNA片段的全长进行标记,而是只将其一端(5’或3’端)进行部分标记。
其特点是可得到全长DNA片段,DNA片段并非均匀标记,标记活性不高。
核酸测试方法

核酸测试方法《核酸测试方法》一、概述核酸检测是利用核酸的特异抗原性,通过形成特异的抗体结合反应来识别有害微生物,从而实现定性和定量检测目的的方法。
它是目前最新的一种确诊微生物存在的方法,用于检测微生物的存在。
它可用于直接诊断和监测可能潜伏在病原体中的微生物,可以确定微生物的种类,鉴定微生物的分布和感染有害微生物的个体。
二、基本原理核酸检测的基本原理是核酸(DNA或RNA)检测。
它是以检测体外核酸,如病毒核酸或细胞核酸,或检测体内有害细胞的核酸,作为被检物,运用形成特异的特异抗体-抗原反应来识别有害微生物的一种理论和技术。
核酸检测的特点是生物特异性强,可以使用到不同的抗体,可以以定性或定量的方式检测出有害微生物。
核酸检测可以检测出病原体的基因序列,并可以分析和判断病原体的类型,使检测效率大大提高。
三、实施方法1. 核酸提取:核酸提取是核酸检测的一个非常重要的环节。
从检测样本中提取病原体的DNA或RNA,这是核酸检测的前提。
根据检测目的,可以采用不同的提取方法,选择最佳的提取方法,为核酸检测提供有效的核酸样品。
2. 核酸标记:如果要实现核酸检测,需要在完成核酸提取后,对检测样本中病毒核酸进行标记,以便对其进行定性或定量检测。
标记有两种方式:一种是利用特异的核苷酸引物,利用PCR法进行放大,另一种是利用放射性核素或酶标记,将病毒核酸与特定的抗体或受体特异性结合。
3. 核酸技术:核酸检测技术有多种,最常用的有Southern hybridization(南方杂交)、 Polymerase Chain Reaction(聚合酶链反应)及RNA逆转录聚合酶链反应技术。
4. 数据分析:核酸检测完成后,还需要对检测结果进行分析和评价。
根据检测结果,可以判断是否有有害微生物的存在,以及病原微生物的种类。
四、安全预防1. 核酸检测应遵循和遵守相关安全技术标准,如防止病原体污染、规范操作流程、保证实验室空气流通、密封保存核酸等。
地高辛标记核酸探针的标记方法

地高辛标记核酸探针的标记方法Last revision on 21 December 2020地高辛标记核酸探针的标记方法核酸探针已被广泛用于筛选重组克隆、基因多样性的种性检测和真菌种群内及种群之间的系统发育关系评价。
最早使用的放射性同位素标记核酸探针具有敏感性高、特异性好、分辨力强的特点,但放射性同位素标记也存在着一系列令人困扰的问题,如成本高、探针半衰期短、放射性物质危害人体健康等。
而且在进行放射性同位素标记实验时,需要有专门的实验室及相应的实验保护设施,还需要由经过培训的专业人员来操作,因而限制了在普通实验室进行分子生物学实验。
近几年发展起来的非放射性核酸探针大多通过酶促、光化学和化学手段掺入一种报道基团,这种报道基团可通过高灵敏度的冷光、荧光或金属沉淀等检测系统检测。
另外,应用pH电极或感应器技术的电化学检测系统也有报道。
在这些检测系统中,灵敏度最高的是生物素- 亲合素检测系统和半抗原-抗半抗原地高辛检测系统。
由于生物样品中常含有内源性生物素及生物结合蛋白,生物素标记的核酸探针会发生一些非特异性结合,从而影响实验效果。
与生物素-亲合素系统同样具有高灵敏度,却减少了非特异性结合的地高辛检测系统,已为人们所接受,并得到广泛的应用。
地高辛(Digoxigenin ,DIG) 又称异羟基洋地黄毒甙元,是一种类固醇半抗原分子。
其化学结构如图1 所示。
采用人工方法可以将地高辛的线型间隔臂与dUTP 连接起来,形成DIG-11-dUTP,通过随机引物法或PCR法将其掺入到DNA探针中。
RNA探针的标记是使用噬菌体信息编码的RNA聚合酶,通过体外转录将DIG-11-dUTP掺入到RNA探针中。
寡核苷酸探针的标记则是通过末端转移酶催化,在3'末端加上DIG-11-dUTP/dATP 或DIG-11-ddUTP 尾巴。
对于目的DNA 或RNA 来说,分子杂交后,杂交部分可通过ELISA 实验程序加以检测,即加入一种结合有碱性磷酸酶的地高辛-特异性抗体,它与地高辛半抗原分子形成酶联抗体-半抗原(DIG) 复合物,再加入相应的显色底物,使杂交部分得以显示。
生物素标记核酸方法

生物素标记核酸方法引言:生物素标记核酸方法是一种常用的实验技术,用于检测、定位和研究核酸的功能和分布情况。
生物素(Biotin)是一种小分子化合物,具有较强的亲和力和特异性,可以与酶和抗体等蛋白质结合,从而实现对核酸的标记和检测。
本文将介绍生物素标记核酸的原理、方法和应用。
一、生物素标记核酸的原理生物素标记核酸的原理基于生物素与亲和剂的结合。
生物素能够与亲和剂如酶、抗体等特异性结合,形成稳定的复合物。
通过将亲和剂标记在生物素上,就可以实现对核酸的标记。
常用的生物素标记方法包括生物素标记末端法、生物素标记引物法和生物素标记缺口法等。
二、生物素标记核酸的方法1. 生物素标记末端法生物素标记末端法是一种将生物素标记在核酸的末端的方法。
首先,将核酸末端修复,以便连接生物素分子。
然后,在核酸末端上引入生物素分子,通过特定的酶或化学试剂进行连接。
最后,通过适当的检测方法,如酶标测定法或荧光探针法,可以检测到生物素标记的核酸。
2. 生物素标记引物法生物素标记引物法是一种将生物素标记在核酸引物上的方法。
在引物的适当位置引入生物素分子,并通过特定的酶或化学试剂进行连接。
接下来,使用生物素标记的引物与待检测的核酸进行反应,形成生物素标记的核酸引物-目标核酸复合物。
最后,通过适当的检测方法,如PCR、电泳或原位杂交等,可以检测到生物素标记的核酸。
3. 生物素标记缺口法生物素标记缺口法是一种通过特定酶切割核酸,形成生物素标记的缺口的方法。
首先,在核酸的适当位置引入生物素分子,并通过特定的酶或化学试剂进行连接。
然后,使用特定酶切割核酸,使核酸链断裂形成缺口,并在断裂的位置上引入生物素标记。
最后,通过适当的检测方法,如原位杂交或荧光显微镜观察等,可以检测到生物素标记的核酸。
三、生物素标记核酸的应用1. 分子生物学研究生物素标记核酸方法在分子生物学研究中广泛应用。
例如,通过生物素标记的引物进行PCR扩增,可以检测和定量目标核酸的存在和表达水平。
生物素-亲和素标记技术完整讲解

4 .4 在分离纯化中的应用(亲和层析)
1.平衡
2.上样
3.洗杂 4.洗脱
五 反应特点
1. BAS具有高灵敏度、高特异性、高稳定性和适用性等特点。 2.生物素易与蛋白质和核酸类等生物大分子结合,再和生物素 衍生物结合,将信号多级放大,能保持大分子物质的原有生 物活性。 3.亲和素与生物素间的结合具有极高的亲和力,其反应呈高度 专一性,不增加非特异性干扰,也不会因反应试剂浓度高低 受影响。 4.酸、碱、变性剂及有机溶剂均不会影响亲和素与生物素的结 合力。裴仁军等试验结果表明,链霉亲和素与生物素能够在 结合表面形成均一的多层膜维系紧密的结合。 5.BAS不仅能与酶、荧光素和放射性核素等各类标记技术结合, 还可制成亲和介质,用于分离提纯。 6.BAS操作方便,反应结果可用肉眼观察,实验成本低。
直接法:
+ B-E + A ABC
+ABC
Ab-Ag-Ab-B + ABC
Ab-Ag-Ab-B-ABC
直接法
间接法
(3)标记生物素-抗生物素法 标记亲和素-生物素法(labeled avidin-biotin, LAB)直接法是以标记亲和素(或链霉亲和素)直接 与免疫复合物中的生物素化一抗连接进行酶呈色 反应,间接法是采用生物素化的二抗和抗原结合, 由于加入了二抗,较直接法检测灵敏度要高,对 免疫细胞中免疫球蛋白的定位具有特异性。LAB 法需以生物素标记一抗,应用不如ABC法普遍, 与ABC法相比,LAB法操作较简单,但灵敏度较 低
四 应用
1
在免疫学中的应用
免疫荧光技术中的应用 免疫放射技术中的应用 胶体金技术中的应用 免疫酶技术中的应用 ELISA中的应用
2
3
4
氨基分子的nhs标记

氨基分子的nhs标记
NHS(N-hydroxysuccinimide)是一种用于标记生物分子氨基的活性染料,通常用于蛋白质、核酸、抗体等生物分子的标记。
NHS标记的基本步骤如下:
1. 制备生物分子:准备要标记的生物分子,确保其含有可反应的氨基官能团,并在适当的缓冲液中。
2. 标记反应:将适量的Cy3 NHS ester 加入到含有氨基官能团的生物分子溶液中,在适当的温度下孵育一段时间,使其充分反应。
3. 纯化:如果需要,可以通过透析或层析等方法对标记产物进行纯化。
需要注意的是,NHS 标记的反应条件和纯化方法需要根据具体情况进行调整。
同时,NHS 标记可能会影响生物分子的活性,需要在使用前进行评估。
分子生物学实验基础知识

分子生物学实验基础知识分子生物学是在生物化学基础上发展起来的,以研究核酸和蛋白质结构、功能等生命本质的学科,在核酸、蛋白质分子水平研究发病、诊断、治疗和预后的机制。
其中基因工程(基因技术,基因重组)是目前分子生物学研究热点,这些技术可以改造或扩增基因和基因产物,使微量的研究对象达到分析水平,是研究基因调控和表达的方法,也是分子水平研究疾病发生机制、基因诊断和基因治疗的方法。
转化(trans formation)、转染、转导、转位等是自然界基因重组存在的方式,也是人工基因重组常采用的手段。
基因重组的目的之一是基因克隆(gene clone),基因克隆可理解为以一分子基因为模板扩增得到的与模板分子结构完全相同的基因。
使需要分析研究的微量、混杂的目的基因易于纯化,得以增量,便于分析。
外来基因引起细胞生物性状改变的过程叫转化(transformation),以噬菌体把外源基因导入细菌的过程叫转染(transfection)。
利用载体(噬菌体或病毒)把遗传物质从一种宿主传给另一种宿主的过程叫转导(transduction)。
一个或一组基因从一处转移到基因组另一处的过程叫转位(transposition),这些游动的基因叫转位子。
一、基因工程的常用工具(一)载体载体(Vector)是把外源DNA(目的基因)导入宿主细胞,使之传代、扩增、表达的工具。
载体有质粒(plasmid)、噬菌体、单链丝状噬菌体和粘性末端质粒(粘粒)、病毒等。
载体具有能自我复制;有可选择的,便于筛选、鉴定的遗传标记;有供外源DNA插入的位点;本身体积小等特征。
质粒存在于多种细菌,是染色体(核)以外的独立遗传因子,由双链环状DNA组成,几乎完全裸露,很少有蛋白质结合。
质粒有严紧型和松弛型之分。
严紧型由DNA多聚酶Ⅲ复制,一个细胞可复制1-5个质粒。
而松弛型由DNA多聚酶Ⅰ复制,一个细胞可复制30-50个质粒,如果用氯霉素可阻止蛋白质合成,使质粒有效利用原料,复制更多的质粒。
核酸测序技术

核酸测序技术核酸测序技术是一种用于确定 DNA 或 RNA 分子序列的方法。
它是生物学和医学研究中的关键技术,被广泛应用于基因组学、医学诊断、药物开发等领域。
下面是核酸测序技术的基本原理和常见方法:1.Sanger 测序:•Sanger 测序是最早的一种测序方法,也被称为链终止法。
其基本原理是通过 DNA 聚合酶合成一条新的 DNA 链,但在加入一种称为二进制链特异性的二进制链特异性荧光标记的特殊核苷酸时,聚合过程会被中断。
通过多次反应,可以合成包含所有可能核苷酸的片段,然后通过电泳分离,根据终止点的不同确定序列。
2.Next Generation Sequencing (NGS):•NGS 技术是一组高通量测序技术,能够以高效率和低成本同时测序大量 DNA 或 RNA 样本。
NGS 方法包括 Illumina(碱基测序)、Ion Torrent(半导体测序)、PacBio(单分子实时测序)等,它们使用不同的原理和技术来实现测序。
但它们的共同之处在于将 DNA 或 RNA 样本分成小片段,并使用不同的方法对这些片段进行测序,最后通过计算机算法将这些片段重新组装成完整的序列。
3.第三代测序技术:•第三代测序技术是相对于传统 Sanger 测序和 NGS 技术而言的新一代测序技术。
它的代表性技术包括PacBio 和Oxford Nanopore。
这些技术能够直接测序单个 DNA 或 RNA 分子,避免了PCR 扩增和片段化等步骤,因此具有更快的速度和更低的假阳性率。
此外,第三代测序技术还能够实现长读长序列,有助于解决重复序列和结构变异等难题。
核酸测序技术的不断发展和创新,使得我们能够更加深入地理解生命的基本原理,同时也为医学诊断、个性化医疗、基因编辑等领域提供了重要的工具和支持。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
青岛农业大学
核酸化学结课论文
题目: 核酸的各种标记方法的优缺点比较学院: 生命科学学院
班级: 生物技术1003班
姓名: 陈冠旭
学号: 20105491
指导老师:高龄
2014/5/5
摘要
本文综述了几种常用的DNA/RNA遗传标记及其在遗传多样性研究中的应用。
几种标记方法各有其优缺点, 都能从某个角度反映生物的某些遗传特性, 在
科研中应根据研究目的和背景选用恰当的标记方法, 或不同的标记联合使用, 以
获得全面的信息。
关键词:DNA探针,RNA探针,优缺点
正文
核酸探针根据核酸的性质,可分为DNA和RNA探针;根据是否使用放射性
标记物的与否,可分为放射性标记探针和非放射性标记探针;根据是否存在互补链,可分为单链和双链探针;根据放射性标记物掺入情况,可分为均匀标记和末端标记探针。
下面将介绍各种类型的探针及标记方法。
一、双链DNA探针及其标记方法
分子生物研究中,最常用的探针即为双链DNA探针,它广泛应用于基因
的鉴定、临床诊断等方面。
双链DNA探针的合成方法主要有下列两种:切口平
移法和随机引物合成法。
1. 切口平移法(nick translation) 当双链DNA分子的一条链上产生切口时,E.coli DNA聚合酶Ⅰ就可将核苷酸连接到切口的3'羟基末端。
同时该酶具有从5'→3'
的核酸外切酶活性,能从切口的5'端除去核苷酸。
由于在切去核苷酸的同时又在
切口的3'端补上核苷酸,从而使切口沿着DNA链移动,用放射性核苷酸代替原先
无放射性的核苷酸,将放射性同位素掺入到合成新链中。
最合适的切口平移片
段一般为50-500个核苷酸。
切口平移反应受几种因素的影响: (a) 产物的比活性
取决于[α-32 P]dNTP的比活性和模板中核苷酸被置换的程度。
(b) DNA酶Ⅰ的
用量和E.coli DNA聚合酶的质量会影响产物片段的大小。
(c) DNA模板中的抑
制物如琼脂糖会抑制酶的活性, 故应使用仔细纯化后的DNA。
2. 随机引物合成法随机引物合成双链探针是使寡核苷酸引物与DNA模板结合,
在Klenow酶的作用下,合成DNA探针。
合成产物的大小、产量、比活性依赖于反应中模板、引物、dNTP和酶的量。
通常,产物平均长度为400-600个核苷酸。
利用随机引物进行反应的优点是:
(1)Klenow片段没有5'→3'外切酶活性,反应稳定,可以获得大量的有效探针。
(2)反应时对模板的要求不严格,用微量制备的质粒DNA模板也可进行反应。
(3)反应产物的比活性较高,可达4×109 cpm/μg探针。
(4)随机引物反应还可以在低熔点琼脂糖中直接进行。
二、单链DNA探针
用双链探针杂交检测另一个远缘DNA时,探针序列与被检测序列间有很多
错配。
而两条探针互补链之间的配对却十分稳定,即形成自身的无效杂交,结果使检测效率下降。
采用单链探针则可解决这一问题。
单链DNA探针的合成方法
主要有下列两种:
(1) 以M13载体衍生序列为模板,用Klenow片段合成单链探针;
(2) 以RNA为模板, 用反转录酶合成单链cDNA探针。
1. 从M13载体衍生序列合成单链DNA探针合成单链DNA探针可将模板序列
克隆到噬粒或M13噬菌体载体中,以此为模板,以特定的通用引物或以人工合成
的寡合苷酸为引物, 在[a-32P]-dNTP的存在下,由Klenow片段作用合成放射标记
探针,反应完毕后得到部分双链分子。
在克隆序列内或下游用限制性内切酶切
割这些长短不一的产物,然后通过变性凝胶电泳(如变性聚丙烯酰胺凝胶电泳)将
探针与模板分离开。
双链RF型M13 DNA也可用于单链DNA的制备,选用适当
的引物即可制备正链或负链单链探针。
2. 从RNA合成单链cDNA探针 cDNA单链探针主要用来分离cDNA文库中相
应的基因。
用RNA为模板合成cDNA探针所用的引物有两种:
(1)用寡聚dT为引物合成cDNA探针。
本方法只能用于带Poly(A)的mRNA,并
且产生的探针极大多数偏向于mRNA 3'末端序列。
(2) 可用随机引物合成cDNA探针。
该法可避免上述缺点,产生比活性较高的探针。
但由于模板RNA中通常含有多种不同的RNA分子,所得探针的序列往往比
以克隆DNA为模板所得的探针复杂得多, 应预先尽量富集mRNA中的目的序列。
反转录得到的产物RNA/DNA杂交双链经碱变性后,RNA单链可被迅速地降解成小片段,经Sephadex G-50柱层析即可得到单链探针。
三、末端标记DNA探针
1、利用本方法可对DNA分子量标准进行标记,利用它可定位因片段太小而无
法在凝胶中观察的DNA片段。
2、对DNA的纯度不很严格,少量制备的质粒也可进行末端标记合成探针。
3、末端标记还有其他的一些方法,如利用T4多核苷酸激酶标记脱磷的5'端突
出的DNA和平末端凹缺DNA分子,也可利用该酶进行交换反应标记5'末端。
四、寡核苷酸探针
利用寡核苷酸探针可检测到靶基因上单个核苷酸的点突变。
常用的寡核苷
酸探针主要有两种:单一已知序列的寡核苷酸探针和许多简并性寡核苷酸探针组
成的寡核苷酸探针库。
单一已知序列寡核苷酸探针能与它们的目的序列准确配
对,可以准确地设计杂交条件,以保证探针只与目的序列杂交而不与序列相近的非完全配对序列杂交,对于一些未知序列的目的片段则无效。
五、各种m iRNA标记方法的优缺点及适用条件
总结
每种遗传标记都能反映生物遗传特性的某些方面, 都各有其优缺点和适用范围。
在实际应用中相互结合、相互补充, 采用各种不同的遗传标记研究生物的遗传结构和控制机制, 才能从各个角度揭示生物的遗传本质以及遗传结构和功能的内在联系, 更深入全面地了解与我们的生存密切相关的生物世界, 更好地保护生物资源。
参考文献
[ 1] 李祥龙, 张亚平, 陈圣偶等.1997.山羊品种间线粒体DNA限制性片段长度多态性研究.动物学研究, 18(4):421 ~ 428
[ 2] 李祥龙, 张亚平, 陈圣偶等.1999.山羊mtDNA多态性及其起源分化研究.畜牧兽医学报, 30(4):313 ~ 319
[ 3] 赵兴波, 储明星, 李宁等.2001.绵羊线粒体DNA控制区5' 端序列PCR-SSCP 与序列分析.遗传学报, 28(3):225 ~ 228
[ 4] 张于光, 李迪强, 饶力群等.2003.东北虎微卫星DNA遗传标记的筛选及在亲子鉴定中的应用.动物学报, 49(1):118 ~ 123
[ 5] 张英杰、赵有璋, 刘月琴等.2004.3 个山羊群体中4 个微卫星DNA多态性及其与杂种优势的关系.遗传, 26(5):631 ~ 636
[ 6] 贾斌, 陈杰, 赵茹茜等.2003.新疆8 个绵羊品种遗传多样性和系统发生关系的微卫星分析.遗传学报, 30(9):847 ~ 854
[ 7] 王吉振, 储明星, 王爱国等.2004.用4个微卫星标记分析7 个绵羊群体之间的遗传关系.遗传, 26(5):637 ~ 643
[ 8] 梁永红, 邓彦昌, 熊远著.2000.DNA指纹技术用于湖北白猪群体遗传结构的研究.畜牧兽医学报, 31(2):106 ~ 112
[ 9] 王昕, 曹红鹤, 耿社民等.2002.利用微卫星标记对中国4种小型猪的遗传多样性研究.畜牧兽医学报, 33(6):530 ~ 532
[10] 方盛国, 冯文和, 张安居等.1994.大熊猫亲子鉴定———DNA指纹技术的应用.四川大学学报, 31(3):389 ~ 395
[11] 郭泽坤, 郭继彤, 安志兴等.2002.体细胞克隆山羊微卫星DNA分析.生物化学与生物物理进展, 29(4):655 ~ 658
[12] MISKAEA, ALVAREZ-SAAVEDRAE, TOWNSENDM, et al Microarray analysis of microRNA expression in the developingm amm alian brain [ J] G enom e Bio,l 2004, 5: R68.。
