16SrDNA鉴定菌株的标准操作规程
16S_rDNA鉴定细菌地方法

16S rDNA鉴定细菌的方法细菌16S rDNA鉴定主要分为7个局部:1.提取细菌基因组DNA,2.设计/选择引物进展PCR扩增,电泳检测纯度与大小。
5.目的片段测序。
6.BLAST比对获取相似片段。
试剂:培养基:通常选择组分简单且细菌生长良好的培养基〔培养基组分过于复杂会影响DNA的提取效果,也可以在裂解细菌前用TE缓冲液对菌体进展洗涤。
〕。
1.2 1M Tris-HCl (pH7.4, 7.6, 8.0)〔1L〕:121.1g Tris,加浓盐酸约〔70ml, 60ml, 42ml〕pH大约下降0.03个单位。
(Tris-HCl缓冲液〔0.05mol/L,25Tris〕溶液与x ml 0.1mol/L 盐酸混匀后,加水稀释至100ml 。
Tris缓冲液不仅被广泛用作核酸和蛋白质的溶剂,Tris也是蛋白质电泳缓冲液的主要成分之一) 0.5M EDTA〔pH8.0〕〔1L〕:186.1g Na2EDTA•2H2O,用NaOH调pH至8.0〔约20g〕,高温高压灭菌,室温保存。
(配置方法 1. 称取186.1g Na2EDTA•2H2O,置于1L烧杯中。
2. 参加约800mL的去离子水,充分搅拌。
3. 用NaOH调节pH值值8.0〔约20g NaOH〕。
注意:pH值至8.0时,EDTA才能溶解。
4. 加去离子水将溶液定容至1L。
5. 适量分成小份后,高温高压灭菌。
6. 室温保存。
〕1.4 10×TE Buffer(缓冲液〕(pH7.4,7.6,8.0)〔1L〕:组分:100 mM Tris-HCl,10 mM EDTA。
1M Tris-HCl〔pH7.4,7.6,8.0〕取100ml,0.5M EDTA〔pH8.0〕取20ml。
高温高压灭菌,室温保存。
1×TE Buffer用10×TE Buffer稀释10倍即可。
1.5 10%SDS〔W/V〕:称10gSDS,68℃加热溶解,用浓盐酸调pH至7.2。
[整理]16s_rdna菌种鉴定
![[整理]16s_rdna菌种鉴定](https://img.taocdn.com/s3/m/5171b59470fe910ef12d2af90242a8956becaa61.png)
16S rDNA方法鉴定细菌种属一.目的1. 掌握16S rDNA对细菌进行分类的原理及方法;2. 掌握DNA提取、PCR原理及方法、DNA片段回收等实验操作。
二、原理随着分子生物学的迅速发展,细菌的分类鉴定从传统的表型、生理生化分类进入到各种基因型分类水平,如(G+C)mol%、DNA杂交、rDNA指纹图、质粒图谱和16S rDNA序列分析等。
细菌中包括有三种核糖体RNA,分别为5S rRNA、16S rRNA、23S rRNA,rRNA基因由保守区和可变区组成。
16S rRNA对应于基因组DNA上的一段基因序列称为16S rDNA。
5S rRNA 虽易分析,但核苷酸太少,没有足够的遗传信息用于分类研究;23S rRNA含有的核苷酸数几乎是16S rRNA的两倍,分析较困难。
而16S rRNA相对分子量适中,又具有保守性和存在的普遍性等特点,序列变化与进化距离相适应,序列分析的重现性极高,因此,现在一般普遍采用16S rRNA作为序列分析对象对微生物进行测序分析。
在细菌的16SrDNA中有多个区段保守性,根据这些保守区可以设计出细菌通用物,可以扩增出所有细菌的16SrDNA片段,并且这些引物仅对细菌是特异性的,也就是说这些引物不会与非细菌的DNA互补,而细菌的16SrDNA可变区的差异可以用来区分不同的菌。
因此,16SrDNA可以作为细菌群落结构分析最常用的系统进化标记分子。
随着核酸测序技术的发展,越来越多的微生物的16SrDNA序列被测定并收入国际基因数据库中,这样用16SrDNA 作目的序列进行微生物群落结构分析更为快捷方便。
1、16S rRNA普遍存在于原核生物中。
rRNA参与生物蛋白质的合成过程,其功能是任何生物都必不可少的,而且在生物进化的漫长历程中保持不变,可看作为生物演变的时间钟。
2、在16S rRNA分子中,既含有高度保守的序列区域,又有中度保守和高度变化的序列区域,因而它适用于进化距离不同的各类生物亲缘关系的研究。
16S rDNA鉴定细菌的方法

16S rDNA鉴定细菌的方法细菌16S rDNA鉴定主要分为7个部分:1.提取细菌基因组DNA,2.设计/选择引物进行PCR扩增,电泳检测纯度与大小。
3.琼脂糖凝胶电泳分离4.胶回收目的片段5.目的片段测序。
6.BLAST比对获取相似片段。
7.构建系统进化树试剂:1.1培养基:通常选择组分简单且细菌生长良好的培养基(培养基组分过于复杂会影响DNA 的提取效果,也可以在裂解细菌前用TE缓冲液对菌体进行洗涤。
)。
1.2 1M Tris-HCl (pH7.4, 7.6, 8.0)(1L):121.1g Tris,加浓盐酸约(70ml, 60ml, 42ml),高温高盐灭菌后,室温保存。
冷却到室温后调pH,每升高1℃,pH大约下降0.03个单位。
1.3 0.5M EDTA(pH8.0)(1L):186.1g Na2EDTA•2H2O,用NaOH调pH至8.0(约20g),高温高压灭菌,室温保存。
1.4 10×TE Buffer(pH7.4,7.6,8.0)(1L):组分:100 mM Tris-HCl,10 mM EDTA。
1M Tris-HCl (pH7.4,7.6,8.0)取100ml,0.5M EDTA(pH8.0)取20ml。
高温高压灭菌,室温保存。
1×TE Buffer用10×TE Buffer稀释10倍即可。
1.5 10%SDS(W/V):称10g,68℃加热溶解,用浓盐酸调pH至7.2。
室温保存。
用之前在65℃溶解。
配置时要戴口罩。
6、5M NaCl:称292.2gNaCl,高温高压灭菌,4℃保存。
7、CTAB/NaCl(10%CTAB,0.7M NaCl):溶解4.1g NaCl,加10g CTAB(十六烷基三甲基溴化铵),加热搅拌。
用之前在65℃溶解。
8、氯仿/异戊醇:按氯仿:异戊醇=24:1(V/V)的比例加入异戊醇。
9、酚/氯仿/异戊醇(25:24:1):按苯酚与氯仿/异戊醇=1:1的比例混合Tris-HCl平衡苯酚与氯仿/异戊醇。
16SrDNA鉴定菌株地实用标准操作规程

16SrDNA鉴定菌株的标准操作规程1.适用范围本标准规定了通过特定引物对细菌的16SrDNA片段进行PCR扩增,然后对扩增片段进行序列分析比对,快速获得细菌种属信息的操作规程。
本标准适用于未知细菌的快速种属分析,以及为细菌的生化鉴定提供指导信息。
2.方法和原理16SrDNA鉴定是指用利用细菌16SrDNA序列测序的方法对细菌进行种属鉴定。
包括细菌基因组DNA提取、16SrDNA特异引物PCR扩增、扩增产物纯化、DNA 测序、序列比对等步骤。
是一种快速获得细菌种属信息的方法。
细菌rRNA(核糖体RNA)按沉降系数分为3种,分别为5S、16S和23S rRNA。
16S rDNA是细菌染色体上编码16S rRNA相对应的DNA序列。
16S rDNA由于大小适中,约1.5Kb左右,既能体现不同菌属之间的差异,又能利用测序技术较容易地得到其序列,故被细菌学家和分类学家接受。
在16S rRNA 分子中,可变区序列因细菌不同而异,恒定区序列基本保守,所以可利用恒定区序列设计引物,将16S rDNA片段扩增出来,利用可变区序列的差异来对不同菌属、菌种的细菌进行分类鉴定。
16SrDNA序列的前500bp序列变化较大,包含有丰富的细菌种属的特异性信息,所以对于绝大多数菌株来说,只需要第一对引物测前500bp序列即可鉴别出细菌的菌属。
针对科学论文发表或是前500bp无法鉴别的情况,需要进行16SrDNA 的全序列扩增和测序,得到较为全面的16SrDNA的序列信息。
由于测序仪一次反应最多只能测出700bp的有效序列,为了结果的可靠性,3.设备和材料3.1器材移液器(1000μL、200μL 、100μL、10μL);涡旋振荡器;Eppendorf MixMate;离心机;水浴锅;电泳仪;制冰机;低温冰箱;PCR仪:Veriti 96 Well ThermalCycler ; 凝胶成像仪:VersaDoc MP 4000;基因分析仪:AB3500、AB3130 3.2 试剂DNA 快速提取试剂:PrepMan Ultra ;琼脂糖;PCR 试剂:Taq 酶,10×Taq Buffer(Mg 2+),dNTPs ,ddH 2O 等; ExoSAP-IT ;测序试剂:BigDye Terminator ,5×Sequencing Buffer ;BigDye XTerminator Purification Kit ; 3.3 耗材移液器吸头:1000μL 、200μL 、10μL ;离心管:1.5mL 、200μL ;Micro Amp TM Optical 96-Well Reaction Plate ;Micro Amp TM Optical Adhesive Film ; 3.4 引物16SrDNA 名称 序列扩增长度 第1部分正向引物27F 5'-AGA GTT TGA TCC TGG CTC AG-3' 500 bp 左右反向引物519R 5'- GWA TTA CCG CGG CKG CTG -3' 第2部分 正向引物357F 5'- CTC CTA CGG GAG GCA GCA G-3' 750bp 左右反向引物1115R 5'-AGG GTT GCG CTC GTT GC-3' 第3部分 正向引物926F 5'- AAA CTY AAA KGA ATT GAC GG-3' 560 bp 左右反向引物1492R5'-TAC GGC TAC CTT GTT ACG ACT T-3'4. 操作流程5. 实验方法5.1 核酸提取:挑取单菌落,然后置于装有100μLPrepMan Ultra 的离心管中,涡旋震荡混匀30s 左右,然后100℃水浴10min 后,以离心机最大转速离心3min ,取10μL 上清液与490μL ddH 2O (即稀释50倍),混匀作为下步PCR 的模板DNA 。
16SrDNA鉴定菌株的标准操作规程

16SrDNA鉴定菌株的标准操作规程1.适用范围本标准规定了通过特定引物对细菌的16SrDNA片段进行PCR扩增,然后对扩增片段进行序列分析比对,快速获得细菌种属信息的操作规程。
本标准适用于未知细菌的快速种属分析,以及为细菌的生化鉴定提供指导信息。
2.方法和原理16SrDNA鉴定是指用利用细菌16SrDNA序列测序的方法对细菌进行种属鉴定。
包括细菌基因组DNA提取、16SrDNA特异引物PCR扩增、扩增产物纯化、DNA测序、序列比对等步骤。
是一种快速获得细菌种属信息的方法。
细菌rRNA(核糖体RNA)按沉降系数分为3种,分别为5S、16S和23S rRNA。
16S rDNA是细菌染色体上编码16S rRNA相对应的DNA序列。
16S rDNA由于大小适中,约1.5Kb左右,既能体现不同菌属之间的差异,又能利用测序技术较容易地得到其序列,故被细菌学家和分类学家接受。
在16S rRNA 分子中,可变区序列因细菌不同而异,恒定区序列基本保守,所以可利用恒定区序列设计引物,将16S rDNA片段扩增出来,利用可变区序列的差异来对不同菌属、菌种的细菌进行分类鉴定。
16SrDNA序列的前500bp序列变化较大,包含有丰富的细菌种属的特异性信息,所以对于绝大多数菌株来说,只需要第一对引物测前500bp序列即可鉴别出细菌的菌属。
针对科学论文发表或是前500bp无法鉴别的情况,需要进行16SrDNA的全序列扩增和测序,得到较为全面的16SrDNA的序列信息。
由于测序仪一次反应最多只能测出700bp的有效序列,为了结果的可靠性,通常将16SrDNA全长序列分成3部分进行测序。
3.设备和材料3.1器材移液器(1000μL、200μL 、100μL、10μL);涡旋振荡器;Eppendorf MixMate;离心机;水浴锅;电泳仪;制冰机;低温冰箱;PCR仪:Veriti 96 Well Thermal Cycler;凝胶成像仪:VersaDoc MP 4000;基因分析仪:AB3500、AB31303.2试剂DNA快速提取试剂:PrepMan Ultra;琼脂糖;PCR试剂:Taq酶,10×Taq Buffer(Mg2+),dNTPs,O等; ExoSAP-IT;测序试剂:BigDye Terminator,5×Sequencing Buffer;BigDye XTerminator ddH2Purification Kit;3.3耗材移液器吸头:1000μL、200μL、10μL;离心管:1.5mL、200μL;Micro Amp TM Optical 96-Well ReactionPlate;Micro Amp TM Optical Adhesive Film;3.4引物16SrDNA 名称序列扩增长度第1部分正向引物27F5'-AGA GTT TGA TCC TGG CTC AG-3'500 bp左右反向引物519R5'- GWA TTA CCG CGG CKG CTG -3'第2部分正向引物357F5'- CTC CTA CGG GAG GCA GCA G-3'750bp左右反向引物1115R5'-AGG GTT GCG CTC GTT GC-3'第3部分正向引物926F5'- AAA CTY AAA KGA ATT GAC GG-3'560 bp左右反向引物1492R5'-TAC GGC TAC CTT GTT ACG ACT T-3'其中M=C:A, Y=C:T. K=G:T, R=A:G, S=G:C. W=A:T; all 1:1 4.操作流程5.实验方法5.1 核酸提取:挑取单菌落,然后置于装有100μLPrepMan Ultra 的离心管中,涡旋震荡混匀30s 左右,然后100℃水浴10min 后,以离心机最大转速离心3min ,取10μL 上清液与490μL ddH 2O (即稀释50倍),混匀作为下步PCR 的模板DNA 。
16SrDNA鉴定细菌的方法

16S rDNA鉴定细菌的方法细菌16S rDNA鉴定主要分为7个部分:1.提取细菌基因组 DNA,2.设计/选择引物进行 PCR扩增,电泳检测纯度与大小。
3.琼脂糖凝胶电泳分离4.胶回收目的片段5.目的片段测序。
6.BLAST比对获取相似片段。
7.构建系统进化树试剂:1.1培养基:通常选择组分简单且细菌生长良好的培养基(培养基组分过于复杂会影响DNA 勺提取效果,也可以在裂解细菌前用TE缓冲液对菌体进行洗涤。
)。
1.21M Tris-HCl (pH7.4, 7.6, 8.0) (1L): 121.1g Tris,加浓盐酸约(70ml,60ml, 42ml ),高温高盐灭菌后,室温保存。
冷却到室温后调pH,每升高1C, pH大约下降0.03 个单位。
(Tris-HCl 缓冲液(0.05mol/L , 25C) 50ml 0.1mol/L 三羟甲基氨基甲烷(Tris)溶液与x ml 0.1mol/L 盐酸混匀后,加水稀释至 100ml。
Tris缓冲液不仅被广泛用作核酸和蛋白质的溶剂,Tris也是蛋白质电泳缓冲液的主要成分之一 )1.30.5M EDTA (pH8.0) (1L): 186.1g Na2EDTA?2H2O 用NaOH调pH 至8.0约20g ),高温高压灭菌,室温保存。
(配置方法1.称取186.1g Na2EDT?2H2O置于1L烧杯中。
2.加入约800mL的去离子水,充分搅拌。
3.用NaOH调节pH值值8.0 (约20g NaOH。
注意:pH值至8.0 时,EDTA才能溶解。
4.加去离子水将溶液定容至1L。
5.适量分成小份后,高温高压灭菌。
6.室温保存。
)1.410X TE Buffer (缓冲液)(pH7.4,7.6,8.0) (1L):组分:100 mMTris-HCl ,10 mM EDTA 1M Tris-HCl ( pH7.4,7.6,8.0 ) 取100ml,0.5M EDTA(pH8.0) 取20ml。
16SrDNA鉴定菌株的标准操作规程

16SrDNA鉴定菌株的标准操作规程1.适用范围本标准规定了通过特定引物对细菌的16SrDNA片段进行PCR扩增,然后对扩增片段进行序列分析比对,快速获得细菌种属信息的操作规程。
本标准适用于未知细菌的快速种属分析,以及为细菌的生化鉴定提供指导信息。
2.方法和原理16SrDNA鉴定是指用利用细菌16SrDNA序列测序的方法对细菌进行种属鉴定。
包括细菌基因组DNA提取、16SrDNA特异引物PCR扩增、扩增产物纯化、DNA测序、序列比对等步骤。
是一种快速获得细菌种属信息的方法。
细菌rRNA(核糖体RNA)按沉降系数分为3种,分别为5S、16S和23S rRNA。
16S rDNA是细菌染色体上编码16S rRNA相对应的DNA序列。
16S rDNA由于大小适中,约1.5Kb左右,既能体现不同菌属之间的差异,又能利用测序技术较容易地得到其序列,故被细菌学家和分类学家接受。
在 16S rRNA 分子中,可变区序列因细菌不同而异,恒定区序列基本保守,所以可利用恒定区序列设计引物,将16S rDNA片段扩增出来,利用可变区序列的差异来对不同菌属、菌种的细菌进行分类鉴定。
16SrDNA序列的前500bp序列变化较大,包含有丰富的细菌种属的特异性信息,所以对于绝大多数菌株来说,只需要第一对引物测前500bp序列即可鉴别出细菌的菌属。
针对科学论文发表或是前500bp无法鉴别的情况,需要进行16SrDNA的全序列扩增和测序,得到较为全面的16SrDNA的序列信息。
由于测序仪一次反应最多只能测出700bp的有效序列,为了结果的可靠性,通常将16SrDNA全长序列分成3部分进行测序。
3.设备和材料3.1器材移液器(1000μL、200μL 、100μL、10μL);涡旋振荡器;Eppendorf MixMate;离心机;水浴锅;电泳仪;制冰机;低温冰箱;PCR仪:Veriti 96 Well Thermal Cycler;凝胶成像仪:VersaDoc MP 4000;基因分析仪:AB3500、AB31303.2试剂DNA快速提取试剂:PrepMan Ultra;琼脂糖;PCR试剂:Taq酶,10×Taq Buffer(Mg2+),dNTPs,ddH2O等;ExoSAP-IT;测序试剂:BigDye Terminator,5×Sequencing Buffer;BigDye XTerminator Purification Kit;3.3耗材移液器吸头:1000μL、200μL、10μL;离心管:1.5mL、200μL;Micro Amp TM Optical 96-Well Reaction Plate;页脚内容2Micro Amp TM Optical Adhesive Film ;3.4 引物16SrDNA 名称 序列 扩增长度第1部分 正向引物27F 5'-AGA GTT TGA TCC TGG CTC AG-3'500 bp 左右反向引物519R 5'- GWA TTA CCG CGG CKG CTG -3'第2部分正向引物357F5'- CTC CTA CGG GAG GCA GCA G-3' 750bp 左右反向引物1115R 5'-AGG GTT GCG CTC GTT GC-3' 第3部分正向引物926F5'- AAA CTY AAA KGA ATT GAC GG-3'560 bp 左右反向引物1492R 5'-TAC GGC TAC CTT GTT ACG ACT T-3'其中M=C:A, Y=C:T. K=G:T, R=A:G, S=G:C. W=A:T; all 1:14. 操作流程5.实验方法5.1核酸提取:挑取单菌落,然后置于装有100μLPrepMan Ultra的离心管中,涡旋震荡混匀30s左右,然后100℃水浴10min后,以离心机最大转速离心3min,取10μL上清液与490μL ddH2O(即稀释50倍),混匀作为下步PCR的模板DNA。
用16S rDNA方法鉴定细菌种属
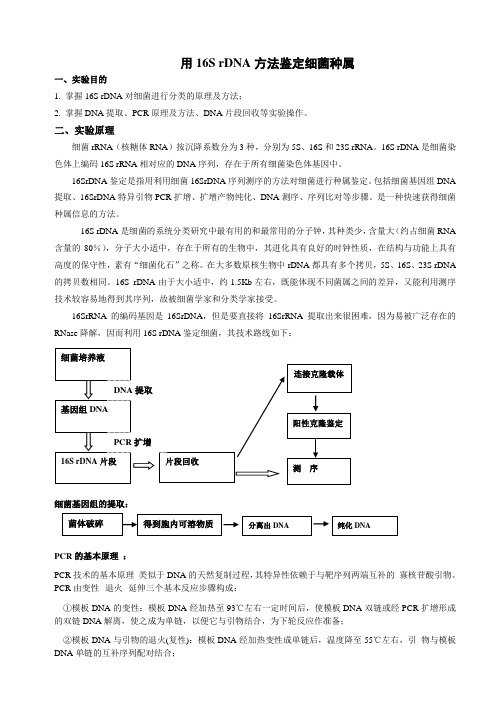
用16S rDNA 方法鉴定细菌种属一、实验目的1. 掌握16S rDNA 对细菌进行分类的原理及方法;2. 掌握DNA 提取、PCR 原理及方法、DNA 片段回收等实验操作。
二、实验原理细菌rRNA (核糖体RNA )按沉降系数分为3种,分别为5S 、16S 和23S rRNA 。
16S rDNA 是细菌染色体上编码16S rRNA 相对应的DNA 序列,存在于所有细菌染色体基因中。
16SrDNA 鉴定是指用利用细菌16SrDNA 序列测序的方法对细菌进行种属鉴定。
包括细菌基因组DNA 提取、16SrDNA 特异引物PCR 扩增、扩增产物纯化、DNA 测序、序列比对等步骤。
是一种快速获得细菌种属信息的方法。
16S rDNA 是细菌的系统分类研究中最有用的和最常用的分子钟,其种类少,含量大(约占细菌RNA 含量的80%),分子大小适中,存在于所有的生物中,其进化具有良好的时钟性质,在结构与功能上具有高度的保守性,素有“细菌化石”之称。
在大多数原核生物中rDNA 都具有多个拷贝,5S 、16S 、23S rDNA 的拷贝数相同。
16S rDNA 由于大小适中,约1.5Kb 左右,既能体现不同菌属之间的差异,又能利用测序技术较容易地得到其序列,故被细菌学家和分类学家接受。
16SrRNA 的编码基因是16SrDNA ,但是要直接将16SrRNA 提取出来很困难,因为易被广泛存在的RNase 降解,因而利用16S rDNA 鉴定细菌,其技术路线如下:细菌基因组的提取:PCR 的基本原理 :PCR 技术的基本原理 类似于DNA 的天然复制过程,其特异性依赖于与靶序列两端互补的 寡核苷酸引物。
PCR 由变性--退火--延伸三个基本反应步骤构成:①模板DNA 的变性:模板DNA 经加热至93℃左右一定时间后,使模板DNA 双链或经PCR 扩增形成的双链DNA 解离,使之成为单链,以便它与引物结合,为下轮反应作准备;②模板DNA 与引物的退火(复性):模板DNA 经加热变性成单链后,温度降至55℃左右,引 物与模板DNA 单链的互补序列配对结合;③引物的延伸:DNA模板--引物结合物在TaqDNA聚合酶的作用下,以dNTP为反应原料,靶序列为模板,按碱基配对与半保留复制原理,合成一条新的与模板DNA 链互补的半保留复制链重复循环变性--退火--延伸三过程,就可获得更多的“半保留复制链”,而且这种新链又可成为下次循环的模板。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
16SrDNA鉴定菌株的标准操作规程
1.适用范围
本标准规定了通过特定引物对细菌的16SrDNA片段进行PCR扩增,然后对扩增片段进行序列分析比对,快速获得细菌种属信息的操作规程。
本标准适用于未知细菌的快速种属分析,以及为细菌的生化鉴定提供指导信息。
2.方法和原理
16SrDNA鉴定是指用利用细菌16SrDNA序列测序的方法对细菌进行种属鉴定。
包括细菌基因组DNA提取、16SrDNA特异引物PCR扩增、扩增产物纯化、DNA测序、序列比对等步骤。
是一种快速获得细菌种属信息的方法。
细菌rRNA(核糖体RNA)按沉降系数分为3种,分别为5S、16S和23S rRNA。
16S rDNA是细菌染色体上编码16S rRNA相对应的DNA序列。
16S rDNA由于大小适中,约1.5Kb左右,既能体现不同菌属之间的差异,又能利用测序技术较容易地得到其序列,故被细菌学家和分类学家接受。
在16S rRNA分子中,可变区序列因细菌不同而异,恒定区序列基本保守,所以可利用恒定区序列设计引物,将16S rDNA片段扩增出来,利用可变区序列的差异来对不同菌属、菌种的细菌进行分类鉴定。
16SrDNA序列的前500bp序列变化较大,包含有丰富的细菌种属的特异性信息,所以对于绝大多数菌株来说,只需要第一对引物测前500bp序列即可鉴别出细菌的菌属。
针对科学论文发表或是前500bp无法鉴别的情况,需要进行16SrDNA的全序列扩增和测序,得到较为全面的16SrDNA的序列信息。
由于测序仪一次反应最多只能测出700bp的有效序列,为了结果的可靠性,通常将16SrDNA全长序列分成3部分进行测序。
3.设备和材料
3.1器材
移液器(1000μL、200μL 、100μL、10μL);涡旋振荡器;Eppendorf MixMate;离心机;水浴锅;电泳仪;制冰机;低温冰箱;PCR仪:Veriti 96 Well Thermal
Cycler ; 凝胶成像仪:VersaDoc MP 4000;基因分析仪:AB3500、AB3130 3.2 试剂
DNA 快速提取试剂:PrepMan Ultra ;琼脂糖;PCR 试剂:Taq 酶,10×Taq Buffer(Mg 2+),dNTPs ,ddH 2O 等; ExoSAP -IT ;测序试剂:BigDye Terminator ,5×Sequencing Buffer ;BigDye XTerminator Purification Kit ; 3.3 耗材
移液器吸头:1000μL 、200μL 、10μL ;离心管:1.5mL 、200μL ;Micro Amp TM Optical 96-Well Reaction Plate ;Micro Amp TM Optical Adhesive Film ; 3.4 引物
16SrDNA 名称 序列
扩增长度 第1部分
正向引物27F 5'-AGA GTT TGA TCC TGG CTC AG -3' 500 bp 左右
反向引物519R 5'- GWA TTA CCG CGG CKG CTG -3' 第2部分 正向引物357F 5'- CTC CTA CGG GAG GCA GCA G-3' 750bp 左右
反向引物1115R 5'-AGG GTT GCG CTC GTT GC-3' 第3部分 正向引物926F 5'- AAA CTY AAA KGA ATT GAC GG-3' 560 bp 左右
反向引物1492R
5'-TAC GGC TAC CTT GTT ACG ACT T-3'
其中M=C:A, Y=C:T. K=G:T, R=A:G, S=G:C. W=A:T; all 1:1
4. 操作流程
5. 实验方法
5.1 核酸提取:
挑取单菌落,然后置于装有100μLPrepMan Ultra 的离心管中,涡旋震荡混匀30s 左右,然后100℃水浴10min
后,以离心机最大转速离心3min ,取10μL 上清液与490μL ddH 2O (即稀释50倍),混匀作为下步PCR 的模板DNA 。
提取的DNA 于-20℃保存。
5.2 基因扩增 5.2.1 PCR 反应体系
备注:DNA模板量通常在100 ng以下,必要时可进行梯度稀释,确定最佳的DNA模
板使用量。
PCR反应体系应在冰中配置,然后置于冰箱中冷却3~5min,最后放于PCR仪上进行反应,这种冷启动法可增强PCR扩增的特异性。
5.2.2PCR反应条件
5.2.3电泳
称取1g琼脂糖置于100mL TAE电泳缓冲液中,加热融化,待温度降至60℃左右时,均匀铺板,制成1%的琼脂糖凝胶。
PCR反应结束后,加样,以100V 电压进行琼脂糖凝胶电泳。
电泳结束后,染色,用凝胶成像仪观察,拍照,记录实验结果。
5.3产物纯化
5.3.1每5μLPCR产物加入2μL ExoSAP-IT试剂,混匀。
5.3.2放入PCR仪中,37℃温育15min, 80℃温育15min。
5.3.3纯化后的PCR产物做为下一步测序反应的模板。
5.4测序反应
5.4.1测序反应体系
5.4.2测序反应条件
5.4.3测序反应纯化(BigDye XTerminator Purification Kit)
5.4.3.1每管加入27μL SAM Solution和6μL BigDye XTerminator Solution。
5.4.3.2放在Eppendorf MixMate 上2000rpm震荡30min。
5.4.3.3在离心机上以1000×g离心2min。
5.4.3.4每管吸取10μL上清液于96孔板中,放入测序仪中测序。
5.5序列比对
基因测序仪得到的测序结果,在MicroSEQ微生物鉴定系统中进行比对,得到菌种的种属信息。
6.关键说明
6.1提取细菌基因组DNA时,对于细胞壁比较薄的革兰氏阴性细菌,可挑
取一菌环菌株,置于100μL ddH2O 中,混匀,沸水热变性10min后,
离心3min分离,稀释50倍后做为PCR模板。
必要时做梯度PCR确认
最佳的模板量。
6.2PCR及测序反应时,为了保证酶的活性,整个体系应在冰中配置;然后
置于低温冰箱中冷却3~5min,最后放于PCR仪上进行反应,这种冷启
动法可增强PCR扩增的特异性。
6.316SrDNA序列的前500bp序列变化较大,包含有丰富的细菌种属的特异
性信息,所以对于绝大多数菌株来说,只需要第一对引物测前500bp
序列即可鉴别出细菌的菌属。
针对科学论文发表或是前500bp无法鉴
别的情况,需要进行16SrDNA的全序列扩增和测序,得到较为全面的
16SrDNA的序列信息。
具体参照附录。
PCR出现非特异性条带的原因:一是引物与靶序列不完全互补、或引物聚合形成二聚体。
二是Mg2+离子浓度过高、退火温度过低,及PCR循环次数过多有关。
其次是酶的质和量,往往一些来源的酶易出现非特异条带而另一来源的酶则不出现,酶量过多有时也会出现非特异性扩增。
其对策有:必要时重新设计引物。
减低酶量或调换另一来源的酶。
降低引物量,适当增加模板量,减少循环次数。
适当提高退火温度或采用二温度点法(93℃变性,65℃左右退火与延伸)。
PCR扩增有时出现涂抹带或片状带或地毯样带。
其原因往往由于酶量过多或酶的质量差,dNTP浓度过高,Mg2+浓度过高,退火温度过低,循环次数过多引起。
其对策有:减少酶量,或调换另一来源的酶。
②减少dNTP的浓度。
适当降低Mg2+浓度。
增加模板量,减少循环
次数。
用RT阴性对照检测是否被基因组DNA污染。
模板浓度过高
10ul体系100ngDNA
附录
(资料性附录)
1. 细菌16SrDNA 三部分的PCR 结果电泳图
M
750bp 1000bp 100bp
2000bp 1
2
3
500bp 250bp
2.细菌16SrDNA鉴定结果
2.116SrDNA前500bp即可反映细菌的种属信息:
菌株TL140924的鉴定结果
2.216SrDNA前500bp不足以反映细菌的种属信息:
菌株70528的鉴定结果
2.316SrDNA的全序列信息:
菌株70528的鉴定结果
如附录2.2、2.3所示,当待测菌株的16SrDNA前500bp序列不足以将待测菌株与库中菌株区分时,则需要测其16SrDNA全长序列以将其区分。
