生工生物miRNA测序项目生物信息分析报告
小RNA测序的一些可选的生物信息分析内容

小RNA测序的一些可选的生物信息分析内容一、Small RNA深度测序分析分子RNA(Small RNA)是生命活动的一类重要调控因子,长约10~40nt,广泛存在于从低等的病毒、线虫、植物到高等的动物机体中。
主要包括微RNA(miRNA)、小干扰RNA(siRNA)、与piwi相互作用的RNA(piRNA)三类,它们通过包括mRNA降解、翻译抑制、异染色质形成等多种途径,在生物体基因表达调控、生物个体发育、代谢及疾病的发生等过程中发挥着重要作用。
Small RNA结合Illumina HiSeq2500/MiSeq新一代测序平台,一次性可获得数百万条sRNA序列,通过生物信息方法,可以在全基因组水平快速鉴定和预测各种类型的Small RNA,得到其可能调控的基因的功能,为Small RNA功能及其对基因调控机制的研究提供了有力工具。
目前,提供1×50bp读长,HiSeq2500、MiSeq两种测序平台的Small RNA测序和信息分析服务。
技术优势精确度高:可以检测出sRNA单个碱基的差异;灵敏度高:可以检测出组织中几个到几万个拷贝的sRNA;价格低廉:一次可产出上百万条序列;样品起始量低:使用NEB原装small RNA建库试剂盒及配套试剂,样品起始量低至3ug;既可以鉴定已知的sRNA,又能够发现新的sRNA。
生物信息分析内容标准信息分析内容及流程:图1 Small RNA标准生物信息分析流程图此外,Small RNA还有如下可选分析内容:更多定制化分析内容,可以根据您的科研需求量身定制。
项目流程诺禾致源严格的样品检测流程,规范化的建库操作,高稳定的测序平台为您的项目获得高质量的数据提供了有力的保证。
HiSeq 2000项目周期45天,MiSeq测序项目周期14天。
图2 HiSeq/MiSeq Small RNA项目流程图建库测序流程诺禾致源使用NEB针对Illumina测序平台研发的Small RNA建库试剂盒(NEB Multiplex Small RNA Library Prep Set for Illumina)及配套的原装进口试剂,只需切胶一次,避免了反复切胶造成的Small RNA信息丢失,大大减少了所需的样品量。
生物信息学研究员基因组测序数据分析总结

生物信息学研究员基因组测序数据分析总结生物信息学研究员在基因组测序数据分析方面具有重要的角色和责任。
基因组测序数据是基因组学研究的核心资源,对于揭示基因功能、疾病机理等方面的研究具有重要的意义。
本文将就生物信息学研究员在基因组测序数据分析中所扮演的角色和开展的工作进行总结和讨论。
一、基因组测序数据质量控制为了确保基因组测序数据的准确性和可靠性,生物信息学研究员首先要对测序数据进行质量控制。
质量控制包括检查测序数据的碱基质量情况,检测是否存在低质量的碱基、接头序列等,并对测序数据进行修剪或过滤,以去除低质量的碱基或序列。
此外,生物信息学研究员还需检查是否存在接头污染、宿主序列等,并对其进行剔除或分离。
二、基因组测序数据比对与拼接基因组测序数据比对与拼接是生物信息学研究员在基因组测序数据分析中的重要环节。
基因组测序数据比对是将读取序列与参考基因组进行比对,以确定每个读取序列的来源和位置。
生物信息学研究员可以使用一系列的比对工具和算法进行比对分析,并通过评估比对质量来筛选可靠的比对结果。
基因组测序数据拼接是根据比对结果,将读取序列进行拼接,得到完整的基因组序列。
三、基因组测序数据变异检测基因组测序数据变异检测是生物信息学研究员在基因组测序数据分析中的重要任务。
通过对比对结果进行进一步分析,生物信息学研究员可以检测和鉴定基因组的各种变异信息,如单核苷酸多态性(SNP)、缺失、插入等。
生物信息学研究员可以使用一系列的工具和方法进行变异检测,并通过筛选和过滤得到高可靠性和高准确性的变异结果。
四、基因组测序数据功能注释和通路分析基因组测序数据功能注释和通路分析是生物信息学研究员在基因组测序数据分析中的重要内容。
生物信息学研究员可以利用一系列的数据库和工具,对基因组测序数据进行功能注释,如预测基因的功能、编码蛋白质的功能等。
此外,生物信息学研究员还可以进行通路分析,揭示基因组测序数据在生物学过程和信号通路中的作用和调控机制。
miRNA研究方法
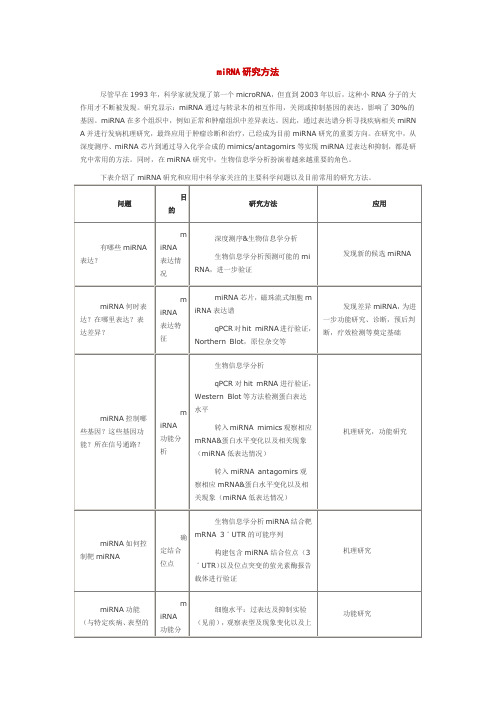
miRNA研究方法miRNA主要研究技术深度测序抽提分离小分子(例如18-30nt)RNA,通过RT-PCR扩增之后,利用solexa深度测序,并进行生物信息学分析,获得miRNA表达谱。
深度测序结合生物信息学分析手段,可以对海量数据进行分析,分别统计出已知的miRNA(miRNA-known)、新的miRNA(miRNA-new)以及可能的新miRNA(mi RNA-cadidate new),并对新发现的miRNA进行靶基因分析,功能预测等。
通过深度测序的方法,可以发现新的miRNA,为进一步的深入研究奠定基础。
miRNA芯片利用miRNA芯片(例如Agilent miRNA芯片),可以高通量分析miRNA表达的时空特异性、不同样本(例如癌组织和癌旁组织)中miRNA的差异表达,进而进行把基因分析,功能注释、通路分析,网络分析等,以了解miRNA在疾病发生中的作用。
与深度测序不同,miRNA芯片针对已知miRN A进行研究。
筛选到的差异miRNA可以利用q-pCR进行验证。
q-PCR q-PCR技术可以用来检测miRNA以及其靶mRNA和相关mRNA等的检测,主要方法有茎环法和加尾法等。
前者针对特定miRNA设计引物,特异性好,然而成本较高,周期长;后者采用通用引物,时间短,通量较大。
(欧易生物为您提供以上项目的优质服务,详情请关注:)Mimics以及Antogomirs 通常基因功能研究常常采用过表达或干涉(抑制、敲除)等方法,miRNA 研究也不例外。
通过导入化学合成的小分子miRNA mimics或拮抗miRNA 的antagomir,观察靶mRNA 及编码蛋白表达以及细胞、动物水平的表型变化,进行信号通路研究,是目前miRNA功能研究的常用方法。
萤光素酶报告系统在确定了靶mRNA之后,找到位于3ˊ-UTR区的miRNA结合位点是研究者下一步关心的内容。
通过生物信息学分析获得候选的miRNA结合区域,将野生型和结合位点突变的3ˊ-UTR序列克隆入商品化的萤光素酶报告载体(例如pMIR-REPORT? miRNA Expression Reporter Vector系统),通过观察miRNA对发光强度的影响对结合位点加以验证。
如何使用生物大数据技术分析miRNA表达谱

如何使用生物大数据技术分析miRNA表达谱miRNA(microRNA)是一类短小非编码RNA分子,起调控基因表达的重要作用。
miRNA的表达谱分析在生物学研究中具有重要意义,可以帮助我们理解miRNA的功能,发现miRNA与疾病之间的关联,并为疾病的诊断和治疗提供新思路。
生物大数据技术为miRNA表达谱的分析提供了巨大的支持和便利。
本文将介绍如何使用生物大数据技术分析miRNA表达谱。
首先,获取miRNA表达谱数据。
miRNA表达谱数据可以通过多种方法获得,包括实验室测序技术、公共数据库下载等。
实验室测序技术包括高通量测序技术(如RNA-seq)和芯片技术(如miRNA芯片)。
公共数据库,如The Cancer Genome Atlas(TCGA)、Gene Expression Omnibus(GEO)等,收集了大量的miRNA表达谱数据。
根据具体研究需求,选择合适的数据来源并下载相应的数据。
接下来,对miRNA表达谱数据进行预处理。
预处理的目标是去除噪声和干扰,保证数据的质量。
首先,进行数据清洗,去除低质量的读数和低表达miRNA。
其次,进行归一化操作,使不同样本之间的数据可比。
常用的归一化方法包括总读数归一化、RPKM归一化等。
最后,进行差异分析,找出在不同样本之间表达差异显著的miRNA。
差异分析方法包括t检验、方差分析、Wald检验等,根据实际情况选择合适的方法。
然后,进行miRNA的功能注释分析。
miRNA的功能注释可以帮助我们了解miRNA在调控基因表达和信号通路中的具体作用。
常用的功能注释方法包括GO (Gene Ontology)富集分析和KEGG(Kyoto Encyclopedia of Genes and Genomes)富集分析。
GO富集分析可以通过对miRNA靶基因的功能分类,揭示miRNA在生物学过程、分子功能和细胞组成等方面的参与情况。
KEGG富集分析可以帮助我们了解miRNA调控的代谢途径、信号通路以及相关的疾病。
生物信息学实习报告

一、实习背景随着生物科学的快速发展,生物信息学作为一门新兴交叉学科,日益受到广泛关注。
为了更好地将理论知识与实践相结合,提升自身综合素质,我于今年暑假期间,在XXX生物科技有限公司开展了为期一个月的生物信息学实习。
二、实习单位简介XXX生物科技有限公司是一家专注于生物信息学研究的科技型企业,主要从事基因组学、转录组学、蛋白质组学等领域的研究与开发。
公司拥有一支经验丰富的研发团队,为我国生物信息学领域的发展做出了积极贡献。
三、实习内容1. 基因组数据分析在实习期间,我主要参与了基因组数据分析项目。
具体工作如下:(1)学习并掌握了基因组数据分析的基本流程,包括数据预处理、比对、注释、统计等。
(2)熟练运用多种生物信息学软件,如SAMtools、BAMSurgeon、Picard等,对基因组数据进行处理和分析。
(3)通过分析基因组数据,发现基因变异、转录本结构变异等生物学特征,为后续研究提供依据。
2. 转录组数据分析除了基因组数据分析,我还参与了转录组数据分析项目。
具体工作如下:(1)学习并掌握了转录组数据分析的基本流程,包括数据预处理、比对、差异表达分析等。
(2)熟练运用多种生物信息学软件,如TopHat、Cufflinks、DESeq2等,对转录组数据进行处理和分析。
(3)通过分析转录组数据,发现差异表达基因、miRNA等生物学特征,为后续研究提供依据。
3. 蛋白质组数据分析此外,我还参与了蛋白质组数据分析项目。
具体工作如下:(1)学习并掌握了蛋白质组数据分析的基本流程,包括蛋白质提取、质谱分析、数据预处理等。
(2)熟练运用多种生物信息学软件,如Proteome Discoverer、Mascot等,对蛋白质组数据进行处理和分析。
(3)通过分析蛋白质组数据,发现蛋白质相互作用、信号通路等生物学特征,为后续研究提供依据。
四、实习收获1. 理论与实践相结合通过实习,我深刻体会到理论知识与实践操作的重要性。
miRNA测序基础及研究介绍

Publication: ( 1991 – May 1st, 2008) siRNA : 15,842 miRNA: 2,641
二、小RNA研究流程
Mapping Genome Total RNA Mapping known miRNA Build Library Clone & Sequencing Raw Data Clean Data Mapping ncRNAs Identify Repeat Identify Exon/Intron Get Unannotated
182 102 109
4.sRNA与重复序列的比较信息
5.sRNA与mRNA/exon/intron的比较信息
• Small RNAs derived from mRNAs/exons/introns (PCMVR-1)
Type Exon_sense Exon_antisense Intron_sense Intron_antisense # of unique 18735 2840 23510 12367 # of total 41496 10805 776249 177227
Bartel DP: MicroRNAs: genomics, biogenesis, mechanism, and function. Cell 2004 116: 281-297.
miRNA function
Lin He and Gregory J.Hannon. MicroRNAs: Small RNAs with a big role in gene regulation. Nature Reviews Genetics 5, 522–531 (2004)
*.table
miRNA_Id zma-MIR159a halibut)] zma-MIR159b halibut)] transcription_Id BM338067[ Pepsinogen A2 [Hippoglossus hippoglossus (Atlantic ],DN224978[ NADH dehydrogenase [Solanum tuberosum (Potato)] BM338067[ Pepsinogen A2 [Hippoglossus hippoglossus (Atlantic ],DN224978[ NADH dehydrogenase [Solanum tuberosum (Potato)]
miRNA分析介绍【高通量测序】
封面页(设计好之后可以删掉这个文本框哦)miRNA分析介绍袁力赟2015-8-28主要内容❝背景介绍❝实验流程❝分析流程、常用软件❝初级分析❝高级分析❝生物学意义分析Non-Coding RNANon-coding RNA的分类❝管家ncRNA:持续表达,是保持细胞正常和活力的所必须,rRNA, tRNA, snoRNA, snRNA, scRNA❝调控ncRNA:只在特定时期表达(比如物种发育,外界刺激)等调控其他基因的表达;micRNA, siRNA, piRNA等rRNA tRNAPiRNA :Gene silencingsnRNA :Pre-mRNA splicing snoRNA : rRNA modificationscRNA :protein synthesis and transport miRNA :gene expressionSmall RNARNA Length(nt)rRNA 120-tRNA 70-90snoRNA 60-400snRNA 100-300scRNA 100-300miRNA 18-25siRNA 18-21piRNA 22-33siRNA miRNAmiRNA的产生过程miRNA hairpinmicRNA❝在所有的small RNA中miRNA是最保守的,调控作用和范围比较广泛,目前研究的比较成熟的调控RNA。
❝1993年Victor Ambros等人在线虫中发现第一个miRNA LIN-4,调控lin-14蛋白质丰度。
❝2000 年发现第二个let-7, 抑制lin-41, lin-14, lin-28, lin-42, daf-12蛋白质的表达。
❝2014年 miRBase已经收录219个物种,28646个成熟miRNA序列。
micRNA权威数据库❝miRBase : miRNA序列数据、注释、预测靶基因等信息的全方位数据库,存储信息最全的公共数据库之一❝miRGene : miRNA基因组及调控数据库❝miRNAMap : miRNA 基因组图谱及miRNA靶基因❝PMRD:植物miRNA数据库/index.shtmlmiRBase❝miRBase 包含3个内容:❝miRBase Sequences所有发表的miRNA序列,基因组位置和相关的注释信息。
《2024年基于生物信息学发现肝细胞癌标志性miRNA及作用与机制研究》范文
《基于生物信息学发现肝细胞癌标志性miRNA及作用与机制研究》篇一一、引言肝细胞癌(Hepatocellular Carcinoma,HCC)是一种常见的恶性肿瘤,其发病率和死亡率均较高。
由于HCC的早期诊断困难,治疗手段有限,因此寻找有效的诊断标志物和治疗方法成为当前研究的重点。
近年来,随着生物信息学的发展,microRNA (miRNA)在肿瘤发生、发展及转移中的作用逐渐受到关注。
miRNA是一种非编码单链小分子RNA,能够通过调控基因表达参与多种生物学过程。
本研究基于生物信息学方法,旨在发现肝细胞癌标志性miRNA及其作用与机制。
二、研究方法1. 数据收集与处理我们首先从公共数据库中收集了肝癌患者的miRNA表达谱数据,并进行了预处理,包括数据清洗、归一化等。
2. 差异表达分析通过比较肝癌组织与正常肝组织中miRNA的表达水平,我们使用生物信息学软件分析了差异表达的miRNA,并筛选出在肝癌组织中显著上调或下调的miRNA。
3. 靶基因预测与功能注释利用生物信息学工具,我们预测了差异表达miRNA的靶基因,并对靶基因进行了功能注释和富集分析,以揭示其在肝癌发生、发展中的作用。
4. 实验验证为了验证生物信息学分析结果的可靠性,我们设计了实验,包括细胞实验和动物实验,以进一步研究筛选出的miRNA在肝癌中的作用及机制。
三、结果与分析1. 差异表达miRNA的筛选通过生物信息学分析,我们筛选出在肝癌组织中显著上调的miRNA和显著下调的miRNA。
其中,miR-XXX和miR-YYY在肝癌组织中的表达水平最高。
2. 靶基因预测与功能注释我们预测了miR-XXX和miR-YYY的靶基因,并进行了功能注释和富集分析。
结果显示,这些靶基因主要参与细胞增殖、凋亡、侵袭和转移等生物学过程。
其中,某些靶基因与肝癌的发生、发展密切相关。
3. 实验验证通过细胞实验和动物实验,我们验证了miR-XXX和miR-YYY在肝癌中的作用及机制。
生物信息学分析中miRNA与肿瘤的关系研究
生物信息学分析中miRNA与肿瘤的关系研究随着生物技术和计算机技术的不断发展,生物信息学逐渐成为了一个新兴的研究领域。
生物信息学的研究主要是利用计算机技术和生物学知识对生命体的各种信息进行分析和处理,进而提高我们对生命体本质的认识。
其中,miRNA是生物信息学分析中的一个非常重要的分子,也是肿瘤相关研究的热点之一。
miRNA(microRNA)是一种长度约为20-24个核苷酸的非编码RNA,它能调控多个基因的表达,并参与多种生物过程,从而对生命体的发育、生长、分化和凋亡等产生重要影响。
研究表明,miRNA在多种肿瘤的发生、发展和转移过程中发挥着重要的调控作用。
因此,研究miRNA与肿瘤的关系,对揭示肿瘤的发病机制和提高肿瘤的早期诊断效率具有重要价值。
在生物信息学分析中,常用的miRNA分析方法主要包括两大类:靶基因预测和miRNA表达水平检测。
靶基因预测是指利用计算机算法预测和筛选出与特定miRNA能够匹配的靶基因,从而了解miRNA的调控机制和生物学功能。
靶基因预测的方法主要包括序列比对、结构比对、保守性分析和生物信息学工具等。
在miRNA表达水平检测方面,常用的方法是采用高通量测序技术、芯片技术和qPCR等,检测肿瘤组织、癌旁组织和非癌组织中不同miRNA的表达水平,分析miRNA与肿瘤的相关性和可能的生物学功能。
过去的研究表明,miRNA与肿瘤的关系非常密切。
例如,miR-21在多种肿瘤中都有高表达,它能下调PDCD4蛋白的表达,从而抑制肿瘤细胞凋亡和促进肿瘤细胞增殖;miR-192在肝癌中的表达也明显上调,研究发现miR-192能够增强肝癌细胞的迁移能力和浸润能力,促进肝癌的恶性转移。
另外,miRNA还可作为肿瘤早期诊断的生物标志物,例如miR-21、miR-210、miR-19a等在肺癌、乳腺癌、结直肠癌等多种肿瘤中均被证实作为早期诊断的潜在生物标志物,为肿瘤的早期诊断和治疗提供了新的思路和方法。
miRNA测序概述及实验分析流程
miRNA测序概述及实验分析流程一、microRNA测序概述MicroRNAs(miRNAs)是在真核生物中发现的一类内源性的具有调控功能的非编码RNA,其大小长约20~25个核苷酸。
成熟的miRNAs是由较长的初级转录物经过一系列核酸酶的剪切加工而产生的,随后组装进RNA诱导的沉默复合体,通过碱基互补配对的方式识别靶mRNA,并根据互补程度的不同指导沉默复合体降解靶mRNA或者阻遏靶mRNA的翻译。
最近的研究表miRNA参与各种各样的调节途径,包括发育、病毒防御、造血过程、器官形成、细胞增殖和凋亡、脂肪代谢等等。
microRNA(miRNA)是一种大小约21—23个碱基的单链小分子RNA,是由具有发夹结构的约70—90个碱基大小的单链RNA 前体经过Dicer酶加工后生成,不同于siRNA(双链)但是和siRNA密切相关。
microRNA通过和靶基因mRNA碱基配对引导沉默复合体(RISC)降解mRNA或抑制mRNA的翻译,从而在转录后水平调控蛋白表达(最新发现microRNA也能在转录水平调控基因表达)。
microRNA在物种进化中相当保守,在动物、植物和真菌等中发现的microRNA表达均有严格的组织特异性和时序性。
microRNA在细胞生长和发育过程中起多种作用,包括调控发育、分化、凋亡和增殖等。
目前研究microRNA的方法主要是realtime-PCR、生物芯片技术以及第二代测序技术。
基于第二代测序技术的microRNA测序,可以一次获得数百万条microRNA序列,能够快速鉴定出不同组织、不同发育阶段、不同疾病状态下已知和未知的micoRNA 及其表达差异,为研究microRNA对细胞进程的作用及其生物学影响提供了有力工具。
二、microRNA测序技术特点1.高灵敏度:理论上可以检测单个细胞中一个拷贝的microRNA;2.高精度:可以检测microRNA单个碱基的差异;3.不受先验信息的干扰,既能鉴定已知microRNA,又有能力发现新的microRNA;4.保留定向信息,用于链特异的表达分析;5.利用Barcode在单次运行中经济地分析多个样品。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
4.1.1 分析方法...............................................................................................................10 4.1.2 结果说明...............................................................................................................10 4.2 reads 比对 rRNA、snRNA、snoRNA、tRNA..................................................................16 4.2.1 分析方法...............................................................................................................16 4.2.2 结果说明...............................................................................................................16 4.3 reads 比对 mRNA exon、intron ......................................................................................18 4.3.1 分析方法...............................................................................................................18 4.3.2 结果说明...............................................................................................................18 4.4 reads 比对参考基因组与分类注释 ................................................................................ 19 4.4.1 分析方法...............................................................................................................19 4.4.2 结果说明...............................................................................................................19 4.5 mirDeep2 分析..................................................................................................................22 4.5.1 分析方法...............................................................................................................22 4.4.2 结果说明...............................................................................................................22 4.6 miRNA-mRNA 靶向预测 .................................................................................................. 24 4.6.1 分析方法...............................................................................................................24 4.7 miRNA 碱基替换分析......................................................................................................24 4.7.1 分析方法...............................................................................................................24 4.7.2 结果说明...............................................................................................................25 4.8 miRNA family 分析...........................................................................................................30 4.8.1 分析方法...............................................................................................................30 4.8.2 结果说明...............................................................................................................30 4.9 miRNA 表达量分析..........................................................................................................32 4.9.1 分析方法...............................................................................................................32 4.9.2 结果说明...............................................................................................................32 4.10 miRNA 差异表达分析....................................................................................................38 4.10.1 分析方法.............................................................................................................38 4.10.2 结果说明.............................................................................................................39 4.11 Gene Ontology 富集分析 ...............................................................................................50 4.11.1 分析方法.............................................................................................................50 4.11.2 结果说明.............................................................................................................50 4.12 KEGG Pathway 富集分析 ................................................................................................55
