blast中文教程--参数
NCBI在线BLAST使用方法与结果详解

NCBI在线BLAST使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
下面是具体操作方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。
不同的blast程序上面已经有了介绍。
这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。
选择一个要比对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
一般的话参数默认。
3,blast参数的设置。
注意显示的最大的结果数跟E值,E值是比较重要的。
筛选的标准。
最后会说明一下。
4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。
上机实习四:BLAST序列相似性搜索工具的使用

Assessing whether proteins are homologous
RBP4 and PAEP: Low bit score, E value 0.49, 24% identity (“twilight zone”). But they are indeed homologous. Try a BLAST search with PAEP as a query, and find many other lipocalins.
cut-off parameters
BLAST search strategies
General concepts How to evaluate the significance of your results
How to handle too many results
How to handle too few results
Step 3: choose the database
nr = non-redundant (most general database) dbest = database of expressed sequence tags dbsts = database of sequence tag sites gss = genomic survey sequences htgs = high throughput genomic sequence
Sometimes a real match has an E value > 1
…try a reciprocal BLAST to confirm
Sometimes a similar E value occurs for a short exact match and long less exact match
blast验证引物教程1
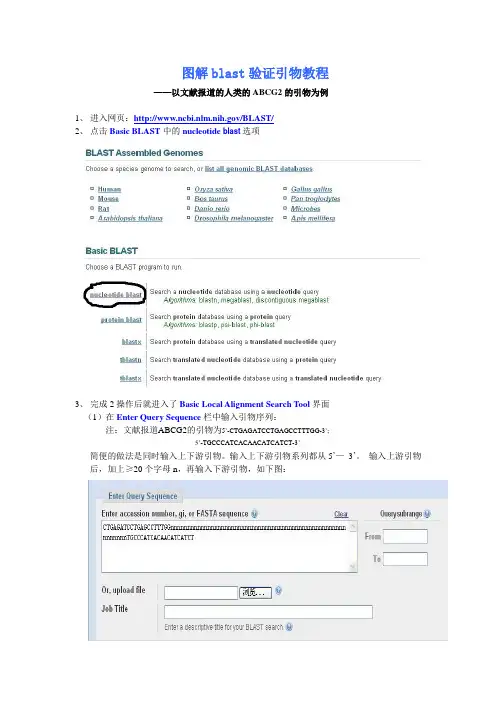
图解blast验证引物教程——以文献报道的人类的ABCG2的引物为例1、进入网页:/BLAST/2、点击Basic BLAST中的nucleotide blast选项3、完成2操作后就进入了Basic Local Alignment Search Tool界面(1)在Enter Query Sequence栏中输入引物序列:注:文献报道ABCG2的引物为5’-CTGAGATCCTGAGCCTTTGG-3’;5’-TGCCCATCACAACATCATCT-3’简便的做法是同时输入上下游引物。
输入上下游引物系列都从5’—3’。
输入上游引物后,加上≥20个字母n,再输入下游引物,如下图:(2)在Choose Search Set栏中:Database根据预操作基因的种属定了,本引物可选Human genomic + transcript或Others (nr etc.)。
本人倾向于选后者,觉得此库信息更多。
如下图:(3)在Program Selection中:选择Somewhat similar sequences (blastn)项,如下图:(4)在此界面最下面:如下图Show results in a new window项是显示界面的形式,可选可不选,在此我们选上了。
关键要点击Algorithm parameters参数设置,进入参数设置界面。
4. 参数设置:(1)在General Parameters中:Expect thresshold期望阈值须改为1000,大于1000也可以;在Word size的下拉框将数字改为7。
如下图:(2)Scoring Parameters无须修改(3)Filters and Masking中,一般来说也没有必要改5.点击最下面一栏的BLAST按钮,如图:6.点击BLAST按钮后,跳转出现如下界面:7. 等待若干秒之后,自动跳转出现显示BLAST结果的网页。
该网页用三种形式来显示blast的结果。
NCBI在线BLAST使用方法与结果详解

NCBI在线BLAST使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
下面是具体操作方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。
不同的blast程序上面已经有了介绍。
这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。
选择一个要比对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
一般的话参数默认。
3,blast参数的设置。
注意显示的最大的结果数跟E值,E值是比较重要的。
筛选的标准。
最后会说明一下。
4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。
NCBI在线BLAST使用方法与结果详解

NCBI在线BLAST使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
下面是具体操作方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。
不同的blast程序上面已经有了介绍。
这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。
选择一个要比对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
一般的话参数默认。
3,blast参数的设置。
注意显示的最大的结果数跟E值,E值是比较重要的。
筛选的标准。
最后会说明一下。
4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。
Blast:大神教你轻松搞定序列比对

Blast:大神教你轻松搞定序列比对Blast (Basic Local Alignment Search T ool) 作为一种序列相似性比对工具,被认为是生物信息分析必须掌握的一款软件。
不管你是做两序列相似性的简单比对,还是引物特异性、基因组成环等个性化分析。
因此,许多看似高大上的基因分析,都可归类于序列间的比较,因此Blast是生信分析中基础性的工具。
今天小编要放大招了,重中之重,送给还在捶胸顿足被一堆数据吓哭的你。
本地Blast本地Blast是该款软件的本地模式,用户可在离线状态下完成目标序列的相似性比对分析。
此种模式不仅可以避免在线提交序列的繁琐和不稳定性,更重要的是能够为用户提供个性化的服务。
若用户需要指定特殊数据库或大量序列的比对,本地Blast则是最优选择。
那么,如何进行本地Blast呢?接下来小编为您献上做本地Blast的基本原则,若您能掌握以下要点,不管对快速应用本地blast还是未来拓展个性化都有很大帮助。
1掌握三个基本要素分别是数据库(database)、待比对序列(query)和目标序列(subject)。
基于这三个基本元素,本地Blast运行方式即是用户选定目标序列(subject)并将其构建成数据库,然后用待比对序列(query)在数据库中搜索,待比对序列遍历数据库中的每一条目标序列后得到最终比对结果。
本地Blast概述:本地Blast是一款集成软件,其中包括blastp、blastx和blastn等模块,通过调用不同的比对模块,blast 实现了五种可能的序列比方式:blastp:蛋白序列与蛋白库作比对,直接比对蛋白序列的同源性。
blastx:核酸序列与蛋白库作比对,将核酸序列先翻译成蛋白序列,再将其与蛋白库作比对。
blastn:核酸序列与核酸库的比对,直接比对核酸序列的同源性。
tblastn:蛋白序列对核算库的比对,现将核酸库翻译成蛋白库,再将蛋白序列与翻译后的蛋白库进行比对。
NCBI在线BLAST使用方法与结果详解
NCBI在线BLAST使⽤⽅法与结果详解NCBI在线BLAST使⽤⽅法与结果详解BLAST(Basic Local Alignment Search Tool)是⼀套在蛋⽩质数据库或DNA数据库中进⾏相似性⽐较的分析⼯具。
BLAST程序能迅速与公开数据库进⾏相似性序列⽐较。
BLAST结果中的得分是对⼀种对相似性的统计说明。
BLAST 采⽤⼀种局部的算法获得两个序列中具有相似性的序列。
Blast中常⽤的程序介绍:1、BLASTP是蛋⽩序列到蛋⽩库中的⼀种查询。
库中存在的每条已知序列将逐⼀地同每条所查序列作⼀对⼀的序列⽐对。
2、BLASTX是核酸序列到蛋⽩库中的⼀种查询。
先将核酸序列翻译成蛋⽩序列(⼀条核酸序列会被翻译成可能的六条蛋⽩),再对每⼀条作⼀对⼀的蛋⽩序列⽐对。
3、BLASTN是核酸序列到核酸库中的⼀种查询。
库中存在的每条已知序列都将同所查序列作⼀对⼀地核酸序列⽐对。
4、TBLASTN是蛋⽩序列到核酸库中的⼀种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋⽩序列,再同所查序列作蛋⽩与蛋⽩的⽐对。
5、TBLASTX是核酸序列到核酸库中的⼀种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋⽩(每条核酸序列会产⽣6条可能的蛋⽩序列),这样每次⽐对会产⽣36种⽐对阵列。
下⾯是具体操作⽅法1,进⼊在线BLAST界⾯,可以选择blast特定的物种(如⼈,⼩⿏,⽔稻等),也可以选择blast所有的核酸或蛋⽩序列。
不同的blast程序上⾯已经有了介绍。
这⾥以常⽤的核酸库作为例⼦。
2,粘贴fasta格式的序列。
选择⼀个要⽐对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
⼀般的话参数默认。
3,blast参数的设置。
注意显⽰的最⼤的结果数跟E值,E值是⽐较重要的。
筛选的标准。
最后会说明⼀下。
4,注意⼀下你输⼊的序列长度。
注意⼀下⽐对的数据库的说明。
5,blast结果的图形显⽰。
NCBI在线BLAST使用方法与结果详解
NCBI在线BLAST使用方法与成果详解之袁州冬雪创作BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中停止相似性比较的分析工具.BLAST程序能迅速与公开数据库停止相似性序列比较.BLAST成果中的得分是对一种对相似性的统计说明. BLAST 采取一种部分的算法获得两个序列中具有相似性的序列.Blast中常常使用的程序先容:1、BLASTP是蛋白序列到蛋白库中的一种查询.库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对.2、BLASTX是核酸序列到蛋白库中的一种查询.先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成能够的六条蛋白),再对每条作一对一的蛋白序列比对.3、BLASTN是核酸序列到核酸库中的一种查询.库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对.4、TBLASTN是蛋白序列到核酸库中的一种查询.与BLASTX 相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对.5、TBLASTX是核酸序列到核酸库中的一种查询.此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会发生6条能够的蛋白序列),这样每次比对会发生36种比对阵列.下面是详细操纵方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列.分歧的blast程序上面已经有了先容.这里以常常使用的核酸库作为例子.2,粘贴fasta格式的序列.选择一个要比对的数据库.关于数据库的说明请看NCBI在线blast数据库的简要说明.一般的话参数默许.3,blast参数的设置.注意显示的最大的成果数跟E值,E 值是比较重要的.筛选的尺度.最后会说明一下.4,注意一下你输入的序列长度.注意一下比对的数据库的说明.5,blast成果的图形显示.没啥好说的.6,blast成果的描绘区域.注意分值与E值.分值越大越靠前了,E值越小也是这样.7,blast成果的详细比对成果.注意比对到的序列长度.评价一个blast成果的尺度主要有三项,E值(Expect),一致性(Identities),缺失或拔出(Gaps).加上长度的话,就有四个尺度了.如图中显示,比对到的序列长度为1405,看Identities这一值,才匹配到1344bp,而输入的序列长度也是为1344bp(看上面的图),就说明比对到的序列要长一点.由Qurey(起始1)和Sbjct(起始35)的起始位置可知,5'端是是多了一段的.有时也要注意3'端的.附:E值(Expect):暗示随机匹配的能够性,E值越大,随机匹配的能够性也越大.E值接近零或为零时,具本上就是完全匹配了.一致性(Identities):或相似性.匹配上的碱基数占总序列长的百分数.缺失或拔出(Gaps):拔出或缺失.用"—"来暗示.。
NCBI在线BLAST使用方法与结果详解
NCBI在线BLAST使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
下面是具体操作方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。
不同的blast程序上面已经有了介绍。
这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。
选择一个要比对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
一般的话参数默认。
3,blast参数的设置。
注意显示的最大的结果数跟E值,E值是比较重要的。
筛选的标准。
最后会说明一下。
4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。
NCBI在线BLAST使用方法与结果详解
NCBI在线BLAST使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
下面是具体操作方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。
不同的blast程序上面已经有了介绍。
这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。
选择一个要比对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
一般的话参数默认。
3,blast参数的设置。
注意显示的最大的结果数跟E值,E值是比较重要的。
筛选的标准。
最后会说明一下。
4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
-m 9:用带有注释行的列表格式显示比对结果,格式与-m 8一样,只是在每 个query的必读结果前面加了注释行用于说明列表中各列的意义
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 9”
BLAST程序的参数
-m 3:显示query在所有subjects的定位和一致性比对信息,不显示subjects之 间的差异
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 3”
4、-o output file name [File Out] (default = stdout)
输出文件的名称,默认值为stdout
BLAST程序的参数
5、 -e Expected value [Real] (default = 10.0)
期望值,这一参数控制搜索的灵敏度 (search sensitivity),可以输入整数 (如100),分数 (如1/100),小数 (如0.001)或是指数 (如5e-5),默认值是 10.0。
BLAST程序的参数
-m 1:显示query在所有subjects上的定位信息,并显示一致性比对信息,subject 之间不同的碱基/氨基酸会被标出
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 1”
BLAST程序的参数
-m 4:显示query在所有subjects上的定位信息但是不显示一致性比对信息, 不显示subjects之间的差异
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 4”
BLAST程序的参数
-m 6:显示query在所有subjects上的定位信息但是不显示每个碱基/氨基酸的 比对信息,补充“-”对齐比对区域,不显示subjects之间的差异
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 6”
BLAST程序的参数
-m 2:显示query在所有subjects上的定位信息但是不显示一致性比对信息, subject之间不同的碱基/氨基酸会被标出
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 2”
BLAST 程 序 的 参 数
徐桂霞 国春策
中国科学院植物研究所
2009.04.24
Windows系统下BLAST程序的安装
BLAST程序的的下2.2.20/
Windows系统下BLAST程序的安装
2 hits 10 hits
BLAST程序的参数
10、-E Cost to extend a gap [Integer] (default = 0)
空位延伸罚分
例:分别设定E为1和2用拟南芥的AP3基因的蛋白质序列在水稻MADS-box基因蛋白质序列库中 搜索同源基因 命令:
1) “blastall -p blastp -i AtAP3_P.txt -d OsMADS_P.txt -o OsAP3_blastp_5e-30_G12.out -e 5e-30 -E 1” 2) “blastall -p blastp -i AtAP3_P.txt -d OsMADS_P.txt -o OsAP3_blastp_5e-30_G12.out -e 5e-30 -E 2”
例:分别使用e值为0.1和0.01的两种情况下,用拟南芥的AP3基因的核酸序列在水稻MADSbox基因核酸序列库中搜索同源基因。
9 hits
7 hits
BLAST程序的参数
6、 -m Specifies alignment view (default = 0)
设定搜索结果的显示格式,m参数的选项有12个 -m 0:默认参数,显示query和subject两两比对的信息
BLAST程序的参数
-m 8:用列表格式显示比对结果。从左到右各列的意义依次是:query名, subject名,一致性百分数,比对长度,错配数,空位数,query比对起始位 点和终止位点,subject比对起始位点和终止位点,期望值,比对得分
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 8”
保存为 保存位置
ncbi.ini
C:\WINDOWS
Windows系统下BLAST程序的运行
BLAST程序的参数
1、 -p Program Name [String]
该参数p代表的是“program”,用来选择程序。其包含五个选项: blastp、blastn、blastx、tblastn和tblastx。 (1) -p blastp:用蛋白质序列搜索蛋白质序列库 (2) -p blastn:用核酸序列搜索核酸库 (3) -p blastx:核酸序列对蛋白质库的比对,核酸序列在比对之前自动 按照六个读码框翻译成蛋白质序列 (4) -p tblastn:蛋白质序列对核酸库的比对,核酸库中的序列按照六个 读码框翻译后与蛋白质序列进行比对搜索 (5) -p tblastx:核酸序列对核酸库在蛋白质质级别的比对,两者都在搜 索之前翻译成为蛋白质质进行比对
1) “blastall -p blastp -i AtAP3_P.txt -d OsMADS_P.txt -o OsAP3_blastp_5e-30_G12.out -e 5e-30 -G 12” 2) “blastall -p blastp -i AtAP3_P.txt -d OsMADS_P.txt -o OsAP3_blastp_5e-30_G12.out -e 5e-30 -G 10”
BLAST程序的参数
9、-G Cost to open a gap (zero invokes default behavior) [Integer] (default = 0)
起始空位罚分
例:分别设定G为12和10用拟南芥的AP3基因的蛋白质序列在水稻MADS-box基因蛋白质序列库 中搜索同源基因 命令:
BLAST程序的的下载:进入NCBI的FTP站点
Windows系统下BLAST程序的安装
BLAST程序的的下载:blast/executables/LATEST
Windows系统下BLAST程序的安装
BLAST程序的的下载:根据电脑系统的不同选择
blast-2.2.20-ia32-win32.exe 或 blast-2.2.20-ia32-win64.exe
-m 11:输出文件为二进制文件
BLAST程序的参数
7、-F Filter query sequence (DUST with blastn, SEG with others) [String] (default = T)
用来屏蔽简单重复和低复杂度序列的参数,有T和F两个选项,选择“T”,则 程序在比对过程中会屏蔽掉query序列中的简单重复和低复杂度序列;选择 “F”则不会屏蔽。默认值是“T”
BLAST程序的参数
-m 7:输出XML格式的blast结果
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 7”
BLAST程序的参数
-m 10:输出文件为ASN格式的文本文件
例:用水稻的OsMADS6基因的核酸序列在水稻MADS-box基因核酸序列库中搜索同源基因。 命令“blastall -p blastn -i OsMADS6_D.txt -d OsMADS_D1.txt -o OsMADS6_blastn_Ff_e-47_m1.out -F F -e 1e-47 -m 10”
win32 - windows 32位操作系统;win64 - windows 64位操作系统,目前多数是win32的
Windows系统下BLAST程序的安装
BLAST程序的的安装:解压缩到某个文件夹
自动生成三个文件夹—bin、data和doc
