InCites平台使用指南
incites数据库使用指南

incites数据库使用指南一、简介incites数据库是一种用于科学研究分析和评估的工具,旨在帮助研究人员和科学家发现重要的研究领域、评估相关研究成果的影响力以及追踪学术合作关系。
本文将为您提供incites数据库的使用指南,帮助您充分利用该数据库进行科研工作。
二、登录和界面介绍您需要通过访问incites数据库的官方网站来登录。
在登录界面,输入您的用户名和密码后,即可进入incites的主界面。
incites的主界面分为多个模块,包括“主页”、“分析”、“指标”、“合作伙伴”等。
在主页中,您可以看到最新的研究动态和相关推荐内容;在分析模块中,您可以进行研究主题的分析和数据可视化;在指标模块中,您可以查看相关研究成果的影响力指标;在合作伙伴模块中,您可以追踪和管理与其他研究人员的合作关系。
三、功能介绍1. 研究主题分析:在incites数据库中,您可以通过输入关键词或选择特定领域进行研究主题的分析。
通过分析,您可以了解该领域的研究热点、相关机构和研究人员等信息,为您的科研工作提供参考。
2. 数据可视化:incites数据库提供了丰富的数据可视化工具,帮助您更直观地了解研究成果的影响力和发展趋势。
您可以使用柱状图、折线图等图表形式展示数据,并根据需要进行筛选和排序。
3. 影响力指标:incites数据库基于学术引用数据,提供了多种影响力指标,包括引用次数、引用频次、H指数等。
这些指标可以帮助您评估研究成果的影响力和质量,为您的科研评估提供科学依据。
4. 合作伙伴追踪:在incites数据库中,您可以管理和追踪与其他研究人员的合作关系。
您可以查看与特定研究人员的合作历史、合作频率等信息,并通过数据可视化工具展示合作网络图,帮助您了解与其他研究人员的合作潜力和合作机会。
四、使用技巧1. 利用高级搜索:在进行研究主题分析时,您可以使用incites数据库提供的高级搜索功能,通过设置相关条件来精确筛选所需数据。
ESI新平台快速使用指南

Essential Science Indicators SM快速使用指南Essential Science Indicators SM (基本科学指标,简称ESI) 是一个基于Web of Science TM数据库的深度分析型研究工具。
ESI可以确定在某个研究领域有影响力的国家、机构、论文和出版物,以及研究前沿。
这种独特而全面的基于论文产出和引文影响力深入分析的数据是政府机构、大学、企业、实验室、出版公司和基金会的决策者、管理者、情报分析人员和信息专家理想的分析资源。
通过ESI,您可以对科研绩效和发展趋势进行长期的定量分析。
基于期刊论文发表数量和引文数据,ESI提供对22个学科研究领域中的国家、机构和期刊的科研绩效统计和科研实力排名。
ESI中的信息包括:•深度的收录范围:您可以访问来自于超过10,000种Web of Science核心合集(SCI/SSCI)收录的期刊,文献类型为Article和Review•提供最近十多年的滚动数据:每2个月更新一次•每一种期刊都按照22个学科进行了分类标引•提供国家、机构、论文和期刊排名•全球近5000多个规范化的机构名称•客观的科研绩效基准值ESI是对科研文献进行多角度、全方位分析的理想资源,可以帮助您轻松发现所需的信息。
通过ESI可以实现:•分析机构、国家和期刊的论文产出和影响力•按研究领域对国家、期刊、论文和机构进行排名•发现自然科学和社会科学中的重大发展趋势•确定具体研究领域中的研究成果和影响力•评估潜在的合作机构,对比同行机构新平台上的ESI在旧版的基础上开发并加强了数据及其呈现方式,使其更加全面易用。
ESI 与 InCites 数据库和 Web of Science 核心合集的数据相互连接,采用更加清晰、准确的可视化方式来呈现数据,用户可以更加轻松地创建、存储并导出报告。
登陆Essential Science Indicators SM请访问:https:///,或从新一代InCites TM平台直接登陆ESI主界面下图中的ESI主界面以红色虚线为界,分为上、下两个部分:上半部 - 数据类型与下载导出•您可以选择ESI各学科所有机构的数据指标(Indicators)、基准值(Field Baseline)或ESI阈值(Citation Thresholds)等不同数据类型•您还可以分别点击三个按钮来下载PDF、CSV或XLS格式的数据文件,将结果发送到电子信箱,或保存在本地的文件夹中下半部 - 数据筛选与分析解读您可以通过自由组合各项指标来:•查找某机构已经进入全球前1%的ESI学科•明确机构在ESI学科中的影响力排名•直接获取某机构在各ESI学科的高水平论文、高被引论文和热点论文筛选区:⏹您可以根据多个选项来筛选数据集,包括研究领域、作者、机构、期刊、国家/地区、研究前沿等;⏹您还可以选择不同的文献类型,包括高水平论文、高被引论、热点论文等;图示区:您可以查看数据的可视化结果,通过点击Show Visualization和Hide Visualization来显示或隐藏可视化地图;结果区:您可以看到分析对象的详细指标表现,通过点击Customize自定义结果区中显示的指标。
赛意数据治理服务-使用指南
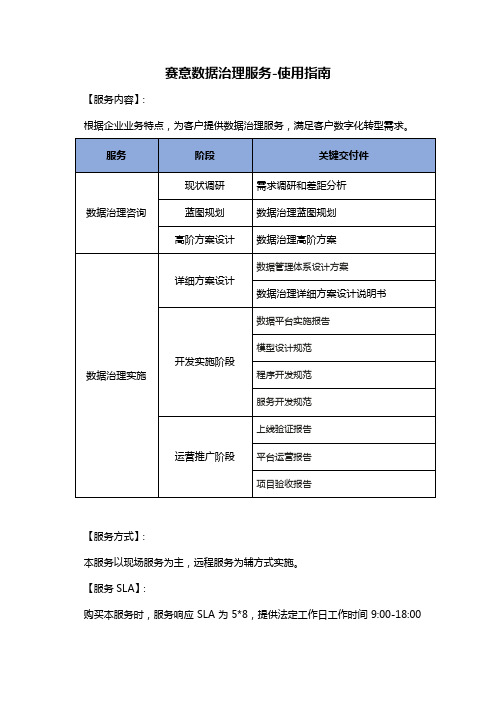
赛意数据治理服务-使用指南
【服务内容】:
根据企业业务特点,为客户提供数据治理服务,满足客户数字化转型需求。
【服务方式】:
本服务以现场服务为主,远程服务为辅方式实施。
【服务SLA】:
购买本服务时,服务响应SLA为5*8,提供法定工作日工作时间9:00-18:00
服务。
服务完成时间与用户共同协商决定,赛意承诺为客户提供优质高效的服务。
系统维保:与用户共同协商决定。
【售前热线】:
售后服务时间:7 * 24小时
售后服务内容:方案咨询、技术咨询
服务热线:
服务邮箱:
重要提示:本商品报价仅为示意,直接购买无效,请联系您的华为客户经理或经销商购买。
incites导入格式

incites导入格式使用InCite数据库进行分析自定义数据集创建自定义数据集的方法从哪里导入自定义数据集有两种方法可以创建自定义数据集:在Web of Science Core Collection中搜索文档,并使用“保存到InCites”功能创建数据集。
登录Web of Science Core Collection。
要使用“保存到InCites”功能,您必须是注册用户,并且必须登录。
执行搜索。
限制检索1980年或以后发布的文件。
选择“保存到InCites”。
在“保存到InCites”窗口中,接受默认数据集名称或创建新名称。
单击保存。
搜索检索到的所有文档都将添加到自定义数据集中(最多50,000个)。
如果只需要选定的文档,请使用“标记结果列表”功能选择单个文档。
然后从“标记结果列表”中选择“保存到InCites”。
从InCites中,上载文档标识符文件。
可以从您编译的文档标识符列表中创建自定义数据集。
有三个有效标识符:•Web of Science Core Collection登录号。
示例:WOS:000388713500004•MEDLINE ID(PubMed ID)。
示例:MEDLINE:24843332•DOI。
例如:10.1088/1478-3975/13/6/066006您上传的文件格式必须为*.csv或*.txt。
每个标识符应位于单独的行中。
例如:该文件可以包含不同类型的标识符。
例如:自定义数据集中文档的发布年份必须为1980或更高版本。
此外,自定义数据集中的最大记录数为50,000。
InCites平台使用更改版本10

2005-2014深圳大学工程学与其它机构 引用排名前10%的论文百分比
3 8
2005-2014深圳大学工程学与其它机构 高被引论文百分比情况
规范化指标
高水平论文
合作指标
WWebebofoSf cSiceinecnece 论论文文数 数 被被引引频频次次
引引文文影影响响力力
被被引引文文献献所所占占百百分分比比
HH指指数数
百百分分位位和和平平均均百百分分位位 学学科科规规范范化化引引文文影影响响力力 学学科科期期望望引引文文影影响响力力
高高被被引引论论文文 热热点点论论文文
2 6
2005-2014深圳大学高影响力论文的表现
2 7
2005-2014深圳大学 论文的平均百分位
2 8
平均百分位(Average Percentile)—示意
全球 发表 论文
论文1 被引用1365次 论文2 被引用1278次 论文3 被引用1139次 论文4 被引用987次 论文5 被引用935次 论文6 被引用821次
InCites平台
InCites中的数据内容
• 数据来源:Web of Science核心合集七大引文索
引数据库 • 出版年:1980-至今
• 全球170多个国家与地区的4,500多所研究机构进
行过变体归并的机构信息
• 每篇文献的题录和指标
• 文献类型:所有
InCites 增强功能
强大的筛选功能
• ESI分类(22个) • Web of Science学科分类(251个) • 中国:SCADC • 意大利:ANVUR • 澳大利亚:ERA 2012 • 巴西:FAPESP • OECD:Frascati • 英国:RAE 2008、2014 • GIPP
亿赛通加密软件操作说明之欧阳学创编

亿赛通加密软件操作说明时间:2021.03.03 创作:欧阳学1 客户端登录【登录】用鼠标右键单击客户端图标,选择【登录用户】,弹出【登录用户】窗口,在【用户ID】输入框中填写正确的用户ID,在【密码】输入框中填写正确的密码,用户ID、密级填写完毕后,点击【确定】按钮,完成登录。
如下图1所示。
图1 图2如果用户ID或者密码填写不正确,提示用户密码不正确。
如上图2所示。
【注销】用鼠标右键单击客户端图标,选择【注销用户】,如下图3所示,弹出提示窗口,如下图4所示,点击【确定】,完成用户注销。
图3图4【修改密码】用鼠标右键单击客户端图标,选择【修改密码】,弹出修改密码的窗口,在旧密码输入框中,填写原密码,在新密码、确认密码中填写新密码,点击【确定】后,提示密码修改成功。
如下图5所示。
图5 图6用户在没有登录时,直接修改密码,修改完成后,用户可以直接登录成功。
【策略更新】当服务端下发新的策略或者策略发生变化时,客户端需要更新策略。
操作步骤:用鼠标右键单击客户端图标,选择【更新策略】,更新的策略即可生效。
如上图6所示。
2 Web页面登录启动浏览器,在地址栏中输入服务器的IP地址。
就可以进入服务器欢迎界面。
如图所示:(我们公司地址为:http://192.168.1.251)点击“进入”,页面跳转到登录界面。
如图所示:输入用户ID和密码后点击登录按钮就可以登录服务器了,第一次登录使用默认密码登录。
3业务申请与审批输入正确的用户名和密码后就可以在web方式进行业务申请,一般用户主要有离线申请、解密申请和邮件外发解密申请。
如图所示:3.1离线申请【功能描述】➢当客户端离线、脱离服务器,想要正常操作CDG文档,可以申请离线。
➢申请离线的用户有时长和次数限制,在限定的时间和次数内,用户可以正常操作。
➢时间、次数超出后,用户的文档就没有权限打开了。
如果想继续操作文档,需要再次申请离线时长和次数。
【操作步骤】1)用鼠标右键单击客户端图标,选择【业务申请】栏下的【离线申请】,如下图所示,弹出离线申请的窗口,如下图所示,用户在离线申请窗口中输入申请时长,在备注中输入申请理由,点及确定,离线申请提交成功。
InCites数据库常用指标手册

目录图目录 (3)表目录 (3)关于本手册 (4)InCites TM数据库介绍 (5)InCites TM 数据库数据来源——Web of Science TM核心合集数据库介绍 (5)期刊评估与筛选 (5)选刊标准简介 (5)文献计量学数据要素 (6)作者 (6)机构 (6)研究领域划分模式 (7)Web of Science TM学科分类 (8)Essential Science Indicators SM学科分类 (8)GIPP 学科分类 (8)多学科及医学期刊论文的重新分类 (8)合理地使用引文指标 (9)文献计量学方法的更多细节 (9)基线(Baseline) (9)引文影响力(Citation Impact) (11)相对于全球平均水平的影响力(Impact Relative to World) (12)学科规范化的引文影响力(Category Normalized Citation Impact) (12)期刊规范化的引文影响力(Journal Normalized Citation Impact) (13)h指数(h-index) (14)平均百分位(Average Percentile) (15)论文被引百分比(% Documents Cited) (16)被引次数排名前1%的论文百分比与被引次数排名前10%的论文百分比(% Documents in Top 1% and % Documents in Top 10%) (16)合作指标(Collaboration Indicators) (18)国际合作论文(International Collaboration) (19)国际合作论文百分比(% of International Collaborations) (19)横向合作论文百分比(% of Industry Collaborations) (19)高被引论文百分比(% Highly Cited Papers) (19)热点论文百分比(% Hot Papers) (19)ESI 引文影响力排名(ESI Most Cited) (19)ESI学科收录机构 (19)附录 (20)InCites TM数据库2.x指标列表 (20)未来计划推出的指标列表(可能发生变化) (22)GIPP –Web of Science TM学科映射表 (23)区域性学科分类模式 (27)ANVUR (27)Australia FOR Level 1 & 2 (27)中国SCADC 77个二级学科 (27)FAPESP (Brasil) (27)OECD (27)UK RAE 2008 & REF 2014 (27)文献类型 (28)更多培训 (29)图目录图 1:不同学科的引文影响力表现 (10)图 2:引文分布示例 (17)图 3:某大学合作指标示例 (18)表目录表 1:基线计算示例 (9)表 2:作者层面的引文影响力 (11)表 3:作者层面的CNCI与JNCI指标示例 (14)表 4:作者层面h指数示例 (15)表 5:一组11篇文献集合的百分位计算示例 (15)关于本手册这本常用指标手册的目的在于为InCites TM数据库的数据来源提供概述。
Web of Science和InCites API客户端R包说明说明书

Package‘wosr’October12,2022Type PackageTitle Clients to the'Web of Science'and'InCites'APIsDescription R clients to the'Web of Science'and'InCites'<https:///products/data-integration/>APIs,whichallow you to programmatically download publication and citation dataindexed in the'Web of Science'and'InCites'databases.URL https://vt-arc.github.io/wosr/index.htmlBugReports https:///vt-arc/wosr/issuesVersion0.3.0License MIT+file LICENSEEncoding UTF-8LazyData trueDepends R(>=3.1)Imports httr,xml2,jsonlite,pbapply,utils,toolsRoxygenNote6.1.0Suggests testthat,knitr,rmarkdown,dplyrNeedsCompilation noAuthor Christopher Baker[aut,cre]Maintainer Christopher Baker<************************>Repository CRANDate/Publication2018-11-0205:30:03UTCR topics documented:auth (2)create_ut_queries (3)pull_cited_refs (3)pull_incites (4)pull_related_recs (5)12auth pull_wos (6)pull_wos_apply (8)query_wos (9)query_wos_apply (10)read_wos_data (11)wosr (11)write_wos_data (12)Index13 auth Authenticate user credentialsDescriptionauth asks the API’s server for a session ID(SID),which you can then pass along to either query_wos or pull_wos.Note,there are limits on how many session IDs you can get in a given period of time (roughly5SIDs in a5minute period).Usageauth(username=Sys.getenv("WOS_USERNAME"),password=Sys.getenv("WOS_PASSWORD"))Argumentsusername Your username.Specify username=NULL if you want to use IP-based authenti-cation.password Your password.Specify password=NULL if you want to use IP-based authenti-cation.ValueA session IDExamples##Not run:#Pass user credentials in manually:auth("some_username",password="some_password")#Use the default of looking for username and password in envvars,so you#don t have to keep specifying them in your code:Sys.setenv(WOS_USERNAME="some_username",WOS_PASSWORD="some_password")auth()##End(Not run)create_ut_queries3 create_ut_queries Create a vector of UT-based queriesDescriptionUse this function when you have a bunch of UTs whose data you want to pull and you need to writea series of UT-based queries to do so(i.e.,queries in the form"UT=(WOS:000186387100005ORWOS:000179260700001)").Usagecreate_ut_queries(uts,uts_per_query=200)Argumentsuts UTs that will be placed inside the UT-based queries.uts_per_query Number of UTs to include in each query.Note,there is a limit on how long your query can be,so you probably want to keep this set to around200.ValueA vector of queries.You can feed these queries to pull_wos_apply to download data for eachquery.Examples##Not run:data<-pull_wos( TS=("animal welfare")AND PY=(2002-2003) )queries<-create_ut_queries(data$publication$ut)pull_wos_apply(queries)##End(Not run)pull_cited_refs Pull cited referencesDescriptionPull cited referencesUsagepull_cited_refs(uts,sid=auth(Sys.getenv("WOS_USERNAME"),Sys.getenv("WOS_PASSWORD")),...)4pull_incitesArgumentsuts Vector of UTs(i.e.,publications)whose cited references you want.sid Session identifier(SID).The default setting is to get a fresh SID each time youquery WoS via a call to auth.However,you should try to reuse SIDs acrossqueries so that you don’t run into the throttling limits placed on new sessions....Arguments passed along to POST.ValueA data frame with the following columns:ut The publication that is doing the citing.These are the UTs that you submitted to pull_cited_refs.If one of your publications doesn’t have any cited refs,it will not appear in this column.doc_id The cited ref’s document identifier(similar to a UT).title Roughly equivalent to the cited ref’s title.journal Roughly equivalent to the cited ref’s journal.author The cited ref’sfirst author.tot_cites The total number of citations the cited ref has received.year The cited ref’s publication year.page The cited ref’s page number.volume The cited ref’s journal volume.Examples##Not run:sid<-auth("your_username",password="your_password")uts<-c("WOS:000362312600021","WOS:000439855300030","WOS:000294946900020")pull_cited_refs(uts,sid)##End(Not run)pull_incites Pull data from the InCites APIDescriptionImportant note:The throttling limits on the InCites API are not documented anywhere and are difficult to determine from experience.As such,whenever pull_incites receives a throttling error from the server,it uses exponential backoff(with a maximum wait time of45minutes)to determine how long to wait before retrying.Usagepull_incites(uts,key=Sys.getenv("INCITES_KEY"),as_raw=FALSE,...)pull_related_recs5Argumentsuts A vector of UTs whose InCites data you would like to download.Each UTis a15-digit identifier for a given publication.You can specify the UT us-ing only these15digits or you can append the15digits with"WOS:"(e.g.,"000346263300011"or"WOS:000346263300011").key The developer key that the server will use for authentication.as_raw Do you want the data frame that is returned by the API to be returned to you inits raw form?This option can be useful if the API has changed the format ofthe data that it is serving,in which case specifying as_raw=TRUE may avoid anerror that would otherwise occur during pull_incites’s data processing step....Arguments passed along to GET.ValueA data frame where each row corresponds to a different publication.The definitions for the columnsin this data frame can be found online at the API’s documentation page(see the DocumentLevelMetricsByUT method details for definitions).Note that the column names are all converted to lowercase bypull_incites and the0/1flag variables are converted to booleans.Also note that not all publica-tions indexed in WoS are also indexed in InCites,so you may not get data back for some UTs.Examples##Not run:uts<-c("WOS:000346263300011","WOS:000362312600021","WOS:000279885800004","WOS:000294667500003","WOS:000294946900020","WOS:000412659200006")pull_incites(uts,key="some_key")pull_incites(c("000346263300011","000362312600021"),key="some_key")##End(Not run)pull_related_recs Pull related recordsDescriptionPull the records that have at least one citation in common with a publication of interest.Usagepull_related_recs(uts,num_recs,editions=c("SCI","SSCI","AHCI","ISTP","ISSHP","BSCI","BHCI","IC","CCR","ESCI"),sid=auth(Sys.getenv("WOS_USERNAME"),Sys.getenv("WOS_PASSWORD")),...)Argumentsuts The documents whose related records you want to pull.num_recs Number of related records to pull for each UT.This value must be<=100.editions Web of Science editions to query.Possible values are listed here.sid Session identifier(SID).The default setting is to get a fresh SID each time you query WoS via a call to auth.However,you should try to reuse SIDs acrossqueries so that you don’t run into the throttling limits placed on new sessions....Arguments passed along to POST.ValueA data frame with the following columns:ut The publications that you passed into pull_related_recs.If one of your publications doesn’t have any related records,it won’t appear here.related_rec The publication that is related to ut.rec_num The related record’s ordering in the result set returned by the API.Records that share more citations with your UTs will have smaller rec_num s.Examples##Not run:sid<-auth("your_username",password="your_password")uts<-c("WOS:000272877700013","WOS:000272366800025")out<-pull_related_recs(uts,5,sid=sid)##End(Not run)pull_wos Pull data from the Web of ScienceDescriptionpull_wos wraps the process of querying,downloading,parsing,and processing Web of Science data.Usagepull_wos(query,editions=c("SCI","SSCI","AHCI","ISTP","ISSHP","BSCI","BHCI","IC","CCR","ESCI"),sid=auth(Sys.getenv("WOS_USERNAME"),Sys.getenv("WOS_PASSWORD")),...)Argumentsquery Query string.See the WoS query documentation page for details on how to writea query as well as this list of example queries.editions Web of Science editions to query.Possible values are listed here.sid Session identifier(SID).The default setting is to get a fresh SID each time you query WoS via a call to auth.However,you should try to reuse SIDs acrossqueries so that you don’t run into the throttling limits placed on new sessions....Arguments passed along to POST.ValueA list of the following data frames:publication A data frame where each row corresponds to a different publication.Note that each publication has a distinct ut.There is a one-to-one relationship between a ut and each of the columns in this table.author A data frame where each row corresponds to a different publication/author pair(i.e.,a ut/author_no pair).In other words,each row corresponds to a different author on a publica-tion.You can link the authors in this table to the address and author_address tables to get their addresses(if they exist).See example in FAQs for details.address A data frame where each row corresponds to a different publication/address pair(i.e.,a ut/addr_no pair).In other words,each row corresponds to a different address on a publication.You can link the addresses in this table to the author and author_address tables to see which authors correspond to which addresses.See example in FAQs for details.author_address A data frame that specifies which authors correspond to which addresses on a given publication.This data frame is meant to be used to link the author and address tables together.jsc A data frame where each row corresponds to a different publication/jsc(journal subject cate-gory)pair.There is a many-to-many relationship between ut’s and jsc’s.keyword A data frame where each row corresponds to a different publication/keyword pair.These are the author-assigned keywords.keywords_plus A data frame where each row corresponds to a different publication/keywords_plus pair.These keywords are the keywords assigned by Clarivate Analytics through an automated process.grant A data frame where each row corresponds to a different publication/grant agency/grant ID triplet.Not all publications acknowledge a specific grant number in the funding acknowledge-ment section,hence the grant_idfield can be NA.doc_type A data frame where each row corresponds to a different publication/document type pair. Examples##Not run:sid<-auth("your_username",password="your_password")pull_wos("TS=(dog welfare)AND PY=2010",sid=sid)8pull_wos_apply #Re-use session ID.This is best practice to avoid throttling limits:pull_wos("TI=\"dog welfare\"",sid=sid)#Get fresh session ID:pull_wos("TI=\"pet welfare\"",sid=auth("your_username","your_password"))#It s best to see how many records your query matches before actually#downloading the data.To do this,call query_wos before running pull_wos:query<-"TS=((cadmium AND gill*)NOT Pisces)"query_wos(query,sid=sid)#shows that there are1,611matching publicationspull_wos(query,sid=sid)##End(Not run)pull_wos_apply Run pull_wos across multiple queriesDescriptionRun pull_wos across multiple queriesUsagepull_wos_apply(queries,editions=c("SCI","SSCI","AHCI","ISTP","ISSHP","BSCI","BHCI","IC","CCR","ESCI"),sid=auth(Sys.getenv("WOS_USERNAME"),Sys.getenv("WOS_PASSWORD")),...)Argumentsqueries Vector of queries to issue to the WoS API and pull data for.editions Web of Science editions to query.Possible values are listed here.sid Session identifier(SID).The default setting is to get a fresh SID each time you query WoS via a call to auth.However,you should try to reuse SIDs acrossqueries so that you don’t run into the throttling limits placed on new sessions....Arguments passed along to POST.ValueThe same set of data frames that pull_wos returns,with the addition of a data frame named query.This data frame frame tells you which publications were returned by a given query.query_wos9 Examples##Not run:queries<-c( TS="dog welfare" , TS="cat welfare" )#we can name the queries so that these names appear in the queries data#frame returned by pull_wos_apply():names(queries)<-c("dog welfare","cat welfare")pull_wos_apply(queries)##End(Not run)query_wos Query the Web of ScienceDescriptionReturns the number of records that match a given query.It’s best to call this function before calling pull_wos so that you know how many records you’re trying to download before attempting to do so.Usagequery_wos(query,editions=c("SCI","SSCI","AHCI","ISTP","ISSHP","BSCI","BHCI","IC","CCR","ESCI"),sid=auth(Sys.getenv("WOS_USERNAME"),Sys.getenv("WOS_PASSWORD")),...)Argumentsquery Query string.See the WoS query documentation page for details on how to writea query as well as this list of example queries.editions Web of Science editions to query.Possible values are listed here.sid Session identifier(SID).The default setting is to get a fresh SID each time you query WoS via a call to auth.However,you should try to reuse SIDs acrossqueries so that you don’t run into the throttling limits placed on new sessions....Arguments passed along to POST.ValueAn object of class query_result.This object contains the number of publications that are re-turned by your query(rec_cnt),as well as some info that pull_wos uses when it calls query_wos internally.10query_wos_apply Examples##Not run:#Get session ID and reuse it across queries:sid<-auth("some_username",password="some_password")query_wos("TS=(\"dog welfare\")AND PY=(1990-2007)",sid=sid)#Finds records in which Max Planck appears in the address field.query_wos("AD=Max Planck",sid=sid)#Finds records in which Max Planck appears in the same address as Mainzquery_wos("AD=(Max Planck SAME Mainz)",sid=sid)##End(Not run)query_wos_apply Run query_wos across multiple queriesDescriptionRun query_wos across multiple queriesUsagequery_wos_apply(queries,editions=c("SCI","SSCI","AHCI","ISTP","ISSHP","BSCI","BHCI","IC","CCR","ESCI"),sid=auth(Sys.getenv("WOS_USERNAME"),Sys.getenv("WOS_PASSWORD")),...)Argumentsqueries Vector of queries run.editions Web of Science editions to query.Possible values are listed here.sid Session identifier(SID).The default setting is to get a fresh SID each time you query WoS via a call to auth.However,you should try to reuse SIDs acrossqueries so that you don’t run into the throttling limits placed on new sessions....Arguments passed along to POST.ValueA data frame which lists the number of records returned by each of your queries.read_wos_data11Examples##Not run:queries<-c( TS="dog welfare" , TS="cat welfare" )query_wos_apply(queries)##End(Not run)read_wos_data Read WoS dataDescriptionReads in a series of CSVfiles(which were written via write_wos_data)and places the data in an object of class wos_data.Usageread_wos_data(dir)Argumentsdir Path to the directory where you wrote the CSVfiles.ValueAn object of class wos_data.Examples##Not run:sid<-auth("your_username",password="your_password")wos_data<-pull_wos("TS=(dog welfare)AND PY=2010",sid=sid)#Write files to working directorywrite_wos_data(wos_data,".")#Read data back into Rwos_data<-read_wos_data(".")##End(Not run)wosr wosrDescriptionwosr12write_wos_data write_wos_data Write WoS dataDescriptionWrites each of the data frames in an object of class wos_data to its own csvfile.Usagewrite_wos_data(wos_data,dir)Argumentswos_data An object of class wos_data,created by calling pull_wos.dir Path to the directory where you want to write thefiles.If the directory doesn’t yet exist,write_wos_data will create it for you.Note,this directory cannotalready have WoS datafiles in it.ValueNothing.Files are written to disk.Examples##Not run:sid<-auth("your_username",password="your_password")wos_data<-pull_wos("TS=(dog welfare)AND PY=2010",sid=sid)#Write files to working directorywrite_wos_data(wos_data,".")#Write files to"wos-data"dirwrite_wos_data(wos_data,"wos-data")##End(Not run)Indexauth,2,4,6–10create_ut_queries,3GET,5POST,4,6–10pull_cited_refs,3pull_incites,4pull_related_recs,5pull_wos,2,6,8,9,12pull_wos_apply,3,8query_wos,2,9query_wos_apply,10read_wos_data,11wosr,11wosr-package(wosr),11write_wos_data,11,1213。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
有全球前1%的学科的大陆高校的省份分布 (2016年11月份数据)
新疆 2
西藏 0
甘肃 3
青海 0
四川 5
云南 3
16
黑龙江
6
北京 22
吉林 3
辽宁
内蒙古
11
0
宁夏 1
山西 4
河北 6
山东 10
天津 7
江苏
陕西 8
重庆4
河南
4 湖北 10
安徽 6
湖南 江西
23
浙江 11
上海 12
贵州
8
2 福建
1 广西
MOLECULAR BIOLOGY & GENETICS MULTIDISCIPLINARY
NEUROSCIENCE & BEHAVIOR
PHARMACOLOGY & TOXICOLOGY PHYSICS PLANT & ANIMAL SCIENCE PSYCHIATRY/PSYCHOLOGY
SOCIAL SCIENCES, GENERAL SPACE SCIENCE
河南理工大学ESI最新排名
21
3089/5460
22
河南理工大学工程学(Engineering)全球排名
23
河南理工大学工程学(Engineering)全球排名
24
河南理工大学工程学(Engineering)全球排名
1279/1339
25
河南理工大学5篇工程学ESI高被引论文(1-2篇)
7
百分位2% 百分位5% 百分位100%
更多角度更多指标
论文产出和 引文影响力
Web of Science论文数 被引频次
引文影响力 被引文献所占百分比
H 指数
规范化指标
百分位和平均百分位 学科规范化引文影响力
学科期望引文影响力 高被引论文 热点论文
期刊规范化引文影响力 期刊期望引文影响力
相对世界平均水平影响力
2006 23.2 18.1 33.1 23.3 26.8 8.1 19.2 12.1 28.3 24 38.7 17.4 8.47 31.9
2007 21.5 16 29.9 21.5 23.5 11.6 16.1 11.8 25.6 21 35.7 18.2 7.73 28.8
2008 19.3 13.3 27 20.9 20.9 10.6 13.1 10.7 23 19.5 32.1 16.5 7.08 25.3
200701/012017/06/30
10年6个月
2017.11
2007/01/012017/08/31
10年8个月
o 发表在11000种期刊上的10,000,000多个条目 o 每一种期刊均按照22个学科进行了分类标引 o 机构名称规范化 o 文献类型包括:Article和Review
15
22个ESI学科
32.6 29.4
22 26.4 22.8 17.7
13 8.68
4.4 1.69 0.13
36.3
34 29.5 26.5 22.5 18.1 13.8 9.59 5.51 1.87 0.24
26.4 23.6 21.3 18.5
16 12.7 10.1
7.3 4.31 1.45 0.17
18 17.2 16.8 15.4 13.7 11.4 9.71 6.99 4.47 1.77 0.26
若CNCI<1, 说明引 文影响力不及全球 平均水平
期刊所在学科: Economics
15/13.81 =1.09
规范化指标2:百分位
论文1 被引用1365次 论文2 被引用1278次 论文3 被引用1139次 论文4 被引用987次 论文5 被引用935次 论文6 被引用821次
…… 论文100 被引用4次
广东 6
3
12
海南 0
17
18
进入全球前1%的ESI学科15个以上的高校
高校
北京大学 浙江大学 中山大学 复旦大学 上海交通大学 清华大学 南京大学
全球前1%的学 科数量(个)
21 18 18 17 17 16 16
近十年 SCI/SSCI总论 文数量(篇)
55570
64047
35945
41946
*对Multidisciplinary的46种期刊中的每篇文献,按照参考文献归类到其他21个学科中,无法归类的文章计入到 Multidisciplinary中。
/incitesLiveESI/ESIGroup/overviewESI/esiJournalsList.html
6பைடு நூலகம்020
56053
36497
总被引次数
712801 649520 401716 527251 588879 642020 426324
全球排名(总 引用)
128 145 262 188 164 150 242
19
中国大陆与世界学科比例分布形态比较
中国与世界学科比例分布比较 • 比重较大的学科: 化学、物理、工程学、材料科学 • 比重较小的学科:多学科、精神病学与心理学、经济与商业、空间 科学、社会科学、免疫学等
Physics Plant & Animal Science Psychiatry/Psychology Social Sciences, general
客观数据支撑学科建设
河南理工大学ESI学科浅析
May 2017
2
定量分析 VS. 同行评议
相互补充
3
世界范围内定量分析指标的演变
发表论文的绝对 数量
问题: 发表1000篇的 机构就一定优于 发表900篇的吗?
考虑到学科和出 版年代差异后的 比较
问题: 当绝对数量差异 较大时如何比较?
发表论文的被引 次数
• Agricultural Science • Biology & Biochemistry • Chemistry • Clinical Medicine • Computer Science • Economics & Business • Engineering • Environment/ Ecology • Geosciences • Immunology • Materials Science
31.1 28.7 25.1 24.8 22.1 18.6 15.2 11.5 7.36 2.89 0.42
5
指标的标准化
不同学科的篇均被引频次有很大的差异 需要学科归一化
因为引文是动态变化的,随着时间推移会不断增长,因此不适宜将不同 时间段发表的文章放在一起比较
需要时间归一化
不同文献类型的文章其引文的行为也有所不同。通常一篇论文获得的引用没有一篇综述得到的引用 次数多
高水平论文
排名1%的论文百分比 排名前10%论文百分比
高被引论文
合作指标
国际合作论文所占百分比 国际合作论文量 与工业合作论文 所占百分比
标准化指标
8
Get it here
9
InCites平台
10
SCIE/SSCI数据应用于世界100多个国家和主要基金组织…
11
大纲
o 全球/中国大陆ESI概况 o 河南理工大学最新ESI排名 o 河南理工大学ESI学科预测 o 河南理工大学现有ESI学科概况 o ESI学科(Engineering)浅析
更新时间 时间范围 数据年限
2017.01
2006/01/012016/10/31
10年10个月
2017.03
2006/01/012016/12/31
11年
2017.05
2007/01/012017/02/28
10年2个月
2017.07
2007/01/012017/04/30
10年4个月
2017.09
2009 17.3 11.8 24.4 18.8 18.7 10.2 11.4 10.4 19.3 17.9 28.6 15.6 6.25 22.9
2010 15.1 10.4 20.5 17.3 16 8.54 9.48 9.24 16.9 14.9 24 14.7 5.44 19.9
2011 12.4 8.35 16.6 14.9 13 7.01 7.34 7.8 13.5 12.7 19.5 12.6 4.27 15.1
2015 1.48 0.98 1.8 2.21 1.49 0.73 0.58 0.97 1.42 1.35 2.16 1.89 0.45 1.72
2016 4 0.19
0.14 0.23 0.24
0.2 0.11 0.11 0.12 0.19 0.21 0.27 0.19 0.07 0.18
52.6 49.3 42.3 37.5 31.8 25.7 18.9 13.4 7.53 2.51 0.26
• Mathematics • Microbiology • Molecular Biology & Genetics • Multidisciplinary* • Neuroscience & Behavior • Pharmacology • Physics • Plant & Animal Science • Psychiatry/Psychology • Social Sciences--general • Space Science
ESI学科 Agricultural Sciences Biology & Biochemistry
Chemistry Clinical Medicine Computer Science Economics & Business
