细菌的转录机制Chapter_06_lecture
分子生物学 第5章 原核生物的转录分解
DNA
Transcription
RNA
MCesesenntgrear lRDNoAgMmosat oof fthme RoNleAcinuclaelrls is foundbiniorilboogsoymIens-f-oourrmpraottieoinn- store synthesizing minacDhinNesA flows:
s70 promoter
• -55 to +20: 全酶的结合 • -20 to +20: 聚合酶紧密结合 ,防止 DNaseΙ的
降解 • Up to position –40: 最重要 (mutagenesis
3
Coding strand
5
4. Asymmetric transcription (不对称转录)
• DNA链上只有部分的区段作为转录模板(反义链 或模板链),且模板链并非自始至终位于同一股 DNA单链上,称为转录的不对称性。
5. transcription unit(转录单元)
➢一段从启动子开始至终止子结束的DNA序列。 包括上游调控区、结构基因区、下游转录终止区 三个部分。
➢ 特异的短的保守序列 ➢ 位于转录起始位点的上游,用负数表示。 ➢ 是RNA聚合酶特异性结合和转录起始所必须的。
+1
Promoter
Terminator
DNA
Transcribed region Sense strand
Transcription
Antisense strand
RNA
Different promoters result in differing efficiencies of transcription initiation, which in turn regulate transcription.
06转录后的调节

下降10~20倍,使RNA合成水平降到正常状态的 5~10%,mRNA总合成量减少3倍,蛋白质降解
速度提高,碳水化合物、脂类合成均明显减少等。 这一系列响应称为stringent response或stringent control
2. ppGpp 的生理功能
关于ppGpp 和pppGpp的作用原理尚不清楚,目前认为 a) 它们是细菌细胞紧缩控制(反应)的信号或称警报素
(三) 细菌的应急反应/紧缩响应 (stringent response)
1. 紧缩响应(应急反应) 2. ppGpp 的生理功能 3. PPGPP对核糖体蛋白质合成的影响
1. 紧缩响应(应急反应)
当细菌处于一种或数种氨基酸全面匮乏的 “氨基酸饥饿”状态时,总之是在营养不良条件 下生长时,为了响应这种困难环境,细菌必须迅 速地关闭许多生理活动,停止包括各种RNA(特 别是rRNA)在内的几乎全部生物化学反应过程, 只保持维持生命最低限量的需要。RNA合成速度
图 2-26 二组分调节系统
二组分系统由二种不同蛋白质组成:
1.位于细胞质膜上的传感蛋白(sensor protein)该 蛋白质具有激酶活性,又称传感激酶。
微生物遗传学第六章节详解演示文稿

四、转座子插入的符号
❖ 用“::”表示各类转座子的插入,如: gal T::IS1 , gal E::IS4 , gal OP::IS1
当前18页,共22页,星期日。
第二节 细菌转座子的插入机制 和转座模型
一、插入机制
1.插入步骤 ❖ 靶序列DNA双链被交错切割 ❖ 转座因子插入到切口处,两条链的各一端共价相连 ❖ 靶序列的单链部分通过复制修补上,原来的靶序列转
微生物遗传学第 六章节详解演示
文稿
当前1页,共22页,星期日。
优选微生物遗传 学第六章节
当前2页,共22页,星期日。
当前3页,共22页,星期日。
当前4页,共22页,星期日。
一、插入序列(insertion sequence, IS )
1、两端含有反向重复序列(IR) 2、含有转座酶基因,启动子和终止区位于IR中 3、转座时需要靶序列(target sequence),转
座后变为转座子两侧的正向重复序列,其长度与Tn种 类有关,有的4bp有的9bp。 2.插入位点的专一程度 ❖ IS4表现绝对专一,要求11-13bp靶序列,Mn只要求 2bp,而Tn则在2者之间。
当前19页,共22页,星期日。
二、转座模型:复制型转座—— Tn3转座模型
❖ 转座子DNA双链被切开,切点在每一条链的3’一端。
座后靶序列重复成为DR 4、IS可独立存在,也常常作为其它转座子的一部
分Hale Waihona Puke 当前5页,共22页,星期日。
❖ 图 10-2 p.244 ❖ 表 10-1 p.243
当前6页,共22页,星期日。
当前7页,共22页,星期日。
当前8页,共22页,星期日。
二、转座子(transposon, Tn)
农杆菌转化机理

农杆菌转化机理引言农杆菌(Agrobacterium tumefaciens)是一种常见的土壤细菌,具有天然的遗传转化能力。
它通过水平基因转移的方式,将外源DNA导入到植物细胞中,并在植物细胞中稳定地整合和表达。
农杆菌转化机理的研究对于植物基因工程和农业生产具有重要意义。
本文将深入探讨农杆菌转化机理的相关内容。
农杆菌转化的基本过程农杆菌转化的基本过程可以分为以下几个步骤:1. 识别和感应农杆菌通过感受植物释放的信号物质,如植物激素和酚类物质,来识别植物细胞。
一旦识别到植物细胞,农杆菌就会感应并附着在植物表面。
2. 切伤和释放农杆菌在附着到植物表面后,通过产生细菌素酶,切伤植物细胞壁,从而释放细菌细胞外质粒(T-DNA)。
3. T-DNA传输T-DNA是农杆菌质粒中的一个片段,包含了农杆菌所携带的外源基因和调控元件。
一旦T-DNA被释放到植物细胞中,农杆菌会利用自身的转座酶将T-DNA整合到植物细胞的染色体中。
4. 基因表达和生物学效应一旦T-DNA整合到植物细胞染色体中,外源基因就可以被植物细胞转录和翻译,从而产生外源蛋白。
这些外源蛋白可以改变植物细胞的生物学过程,如激素合成、细胞分裂和生长等。
农杆菌转化机理的调控农杆菌转化机理的调控非常复杂,涉及到多个信号通路和调控因子的参与。
1. 感应和识别的调控植物激素和酚类物质在农杆菌感应和识别过程中发挥着重要作用。
激素信号通路中的激素感受器和调控因子可以调节农杆菌与植物细胞的相互作用。
2. T-DNA传输的调控T-DNA传输过程中的调控因子包括细菌素酶、转座酶和相关辅助蛋白。
这些因子可以调节T-DNA的释放、整合和稳定性。
3. 基因表达和生物学效应的调控外源基因在植物细胞中的表达和生物学效应受到植物内源基因的调控。
植物细胞中的转录因子和表观遗传修饰可以影响外源基因的转录和翻译水平。
农杆菌转化机理的应用农杆菌转化机理的研究为植物基因工程和农业生产提供了重要的理论基础和实践手段。
第六章 原核生物的转录

• 大肠杆菌RNA聚合酶在37oC时合成RNA 的速度是每秒约40nt。 • 酶的形状呈非球状结构,在一圆柱形孔 道旁有一突起。孔道的大小表明它可以 直接结合16bp的DNA。整个聚合酶所结 合的DNA区域覆盖了约60bp。
• 噬菌体T3和T7所编码的RNA聚合酶只有 一条多肽链,比细菌的多亚基酶小得多。 它们能快速合成RNA( 37oC时每秒200nt) 并识别自身特异的DNA结合序列。
• 在E.coli中,仅由一种聚合酶完成全部RNA的 转录 • 不需要引物 • 以双链DNA为模板 • 5’ 3’ • 行使合成RNA的功能时需要Mg2+ 激活 • 缺少3’ 5’ 外切酶活性,错误率为10-4 ~ 10-5 • 通常为多亚基酶
• 2.大肠杆菌RNA聚合酶 • 是大肠杆菌细胞中最大的酶之一。
• 注意区分终止子(terminator)和终止密码子 (stop coden)
• 在终止反应中,RNA-DNA杂交体分开以 重新形成双链DNA,同时RNA聚合酶和 新合成的RNA从DNA链上被释放下来。
二、大肠杆菌RNA聚合酶
• 1.概述
• RNA聚合酶和 DNA 聚合酶都可以向正在 延伸的链上添加核苷酸。它们的主要区 别为 :RNA聚合酶可以起始一条新链的 合成,而DNA聚合酶没有此功能。 因此, 在DNA的复制中需要由另一种酶先合成 一小段引物。
全酶与启动子结合很迅速
闭合复合物:聚合酶与 处于碱基配对状态的启 动子DNA所形成的最 初的复合物。
and –10 region
2.DNA解旋
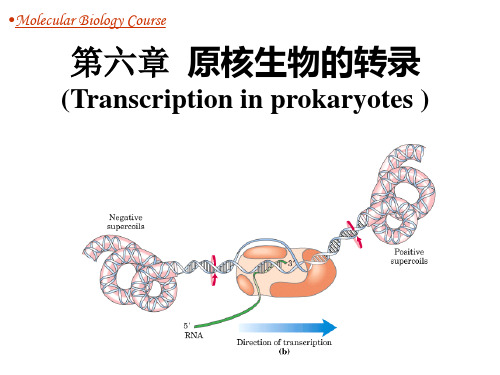
• 为了使反义链实现碱基配对,DNA双螺 旋必须被聚合酶解旋。 • 负超螺旋能增进许多基因的转录,因为 它有利于聚合酶的解旋。 • 特例:DNA促旋酶亚基基因的启动子被 负超螺旋抑制。
分子生物学

•
•
Polymerase binding sites are called promoters这个位点叫启动子
Transcription that begins at promoters is specific, directed by the s-subunit
s的重要作用是指导聚合酶在正确的位置即启动子处进行转录
6-3
6-4
6-5
Sigma as a Specificity Factor
s是一种特异性因子 • • Core enzyme without the s subunit could not transcribe viral DNA, yet had no problems with highly nicked calf thymus DNA无s时,不能转录完整的DNA,但可转录 带切口的DNA With s subunit, the holoenzyme worked equally well on both types of DNA有s时,两 种DNA均能转录
启动子复合体
•
Polymerizing the early nucleotides – polymerase at the promoter结合
最初的几个核苷酸,聚合酶仍然不动
•
Promoter clearance – transcript becomes long enough to form a stable hybrid with template启动子清
启动子复合体
6-12
Core Promoter Elements
核心启动子元件
• • •
There is a region common to bacterial promoters described as 6-7 bp centered about 10 bp upstream of the start of transcription = -10 box Another short sequence centered 35 bp upstream is known as the -35 box Comparison of thousands of promoters has produced a consensus sequence for each of these boxes分析数千个启动子的结果
《转录》 讲义

《转录》讲义在生命的微观世界里,转录是一个至关重要的过程。
它就像是一场神秘而又精准的信息传递之旅,将细胞内储存的遗传密码从 DNA 传递到 RNA,为生命活动的进行奠定基础。
那么,什么是转录呢?简单来说,转录就是以 DNA 为模板合成RNA 的过程。
这一过程发生在细胞核内,由RNA 聚合酶来催化完成。
DNA 就像是一个巨大的基因图书馆,其中存储着生物体构建和维持生命所需的所有信息。
而转录就像是从这个图书馆中挑选出特定的“书籍”(基因),并将其内容复制到一张“便签纸”(RNA)上。
要理解转录,我们首先得了解参与其中的重要角色。
RNA 聚合酶是关键的“抄写员”。
它能够识别 DNA 上的特定序列,也就是启动子区域,然后与之结合,开启转录的进程。
当 RNA 聚合酶结合到启动子上后,它会沿着 DNA 链移动,解开DNA 双螺旋结构。
就像我们打开一本书,逐行读取其中的文字一样。
在这个过程中,它以 DNA 的一条链为模板,按照碱基互补配对原则,将游离的核糖核苷酸连接起来,形成 RNA 链。
在转录过程中,碱基互补配对原则依然发挥着重要作用。
DNA 中的腺嘌呤(A)与 RNA 中的尿嘧啶(U)配对,鸟嘌呤(G)与胞嘧啶(C)配对。
转录的过程并不是一蹴而就的,它可以分为起始、延伸和终止三个阶段。
起始阶段,RNA 聚合酶识别并结合到启动子上,形成转录起始复合物,标志着转录的开始。
延伸阶段,RNA 聚合酶沿着 DNA 模板链移动,不断添加核糖核苷酸,使 RNA 链逐渐延长。
终止阶段,当 RNA 聚合酶遇到终止子序列时,转录停止,RNA 聚合酶和新合成的 RNA 从 DNA 模板上释放出来。
转录产生的 RNA 有多种类型,包括信使 RNA(mRNA)、核糖体RNA(rRNA)和转运 RNA(tRNA)等。
mRNA 是携带遗传信息的“信使”,它从细胞核中出来,进入细胞质,指导蛋白质的合成。
rRNA 则是核糖体的重要组成部分,参与蛋白质的合成过程。
细菌转化实验原理和操作步骤

细菌转化实验原理和操作步骤转化(transformation )是指一段同源或异源的DNA 转入受体细胞并得到表达的水平基因的转移过程,是现代分子生物学研究和基因工程不可缺少的重要技术。
关键词:细菌转化细菌转化一.实验目的1 .以pGLO 质粒转化大肠杆菌为例,学习转化的基本原理及方法。
2 .验证DNA 是遗传物质,加深对中心法则的理解。
二.实验原理转化(transformation )是指一段同源或异源的DNA 转入受体细胞并得到表达的水平基因的转移过程,是现代分子生物学研究和基因工程不可缺少的重要技术。
目前常用的转化方法有CaCl 2 法( 化学转化法) 和电转化法。
本实验是用pGLO 细菌转化试剂盒中提供的pGLO 质粒来转化大肠杆菌,所用方法为CaCl 2 转化法。
pGLO 质粒:pGLO 质粒上主要含有两个基因,一个为编码绿色荧光蛋白(GFP) 的基因,一个为抗生素氨苄青霉素抗性基因。
此外,该质粒还整合有一个特殊的参与受体细胞中绿色荧光蛋白表达的基因调控体系,转化细胞中的绿色荧光蛋白基因只有在培养基中存在阿拉伯糖时才能启动表达。
转化子细胞将在不含阿拉伯糖的培养基上呈白色而在含阿拉伯糖的培养基上显绿色荧光,这种荧光在长波紫外灯下即可观察到。
三.实验材料受体菌:E.coli K12 HB101质粒:pGLO plasmid转化液(CaCl2 )氨苄青霉素(amp )阿拉伯糖(ara )培养基:固体LB 、液体LB接种环、移液器等四.实验步骤1. 准备平板:每组:1 块LB 平板,2 块LB/amp 平板,1 块LB/amp/ara 平板2. 准备感受态细胞:用250 μ l 无菌水或转化液悬浮试剂盒中提供的大肠杆菌菌粉,此即感受态细胞。
3. 活化受体细胞:挑取1 环E.coli K12 HB101 菌液,于LB 培养基上37 ℃活化16-24h 。
1别标记2250 μ l3)4的一个单菌落,悬浮于两管转化液中。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
6-7
RNA Polymerase Binding
Hinkle and Chamberlin proposed: • RNA polymerase holoenzyme binds DNA loosely
at firstns along the DNA until it finds a promoter
• Complex with holoenzyme loosely bound at the promoter is a closed promoter complex as DNA is in a closed ds form
6-5
Binding of RNA Polymerase to Promoters
• How tightly does core enzyme v. holoenzyme bind DNA?
• Experiment measures binding of DNA to enzyme using nitrocellulose filters
– Holoenzyme binds filters tightly
– Core enzyme binding is more transient
6-6
Temperature and RNA Polymerase Binding
• As the temperature is lowered, the binding of RNA polymerase to DNA decreases dramatically
Chapter 6
The Mechanism of Transcription in Bacteria
6.1 RNA Polymerase Structure
By 1969 SDS-PAGE of RNA polymerase from E. coli had shown several subunits
• Holoenzyme can then melt a short DNA region at the promoter to form an open promoter complex with polymerase bound tightly to DNA
6-8
Polymerase/Promoter Binding
• With s subunit, the holoenzyme worked equally well on both types of DNA
6-3
Summary
• The key player in the transcription process is RNA polymerase
• The E. coli enzyme is composed of a core, which contains the basic transcription machinery, and a s-factor, which directs the core to transcribe specific genes
• The presence of the s-subunit permits recognition of authentic RNA polymerase binding sites called promoters
• Transcription that begins at promoters is specific, directed by the s-subunit
6-9
Summary
• The s-factor allows initiation of transcription by causing the RNA polymerase holoenzyme to bind tightly to a promoter
• Holoenzyme binds DNA loosely at first
• Complex loosely bound at promoter = closed promoter complex, dsDNA in closed form
• Holoenzyme melts DNA at promoter forming open promoter complex polymerase tightly bound
– 2 very large subunits are b (150 kD) and b’ (160 kD)
– Sigma (s) at 70 kD – Alpha (a) at 40 kD – 2 copies present in
holoenzyme – Omega (w) at 10 kD
• Was not clearly visible in SDS-PAGE, but seen in other experiments
• Not required for cell viability or in vivo enzyme activity • Appears to play a role in enzyme assembly
6-2
Sigma as a Specificity Factor
• Core enzyme without the s subunit could not transcribe viral DNA, yet had no problems with highly nicked calf thymus DNA
6-4
6.2 Promoters
• Why was the core RNA polymerase capable of transcribing nicked DNA in the previous table?
• Nicks and gaps are good sites for RNA polymerase to bind nonspecifically
