酶切反应条件的优化
载体双酶切

载体双酶切技术是分子生物学领域常用的实验技术之一,它通过利用两种不同的限制性内切酶作用于同一个质粒DNA,从而实现对目标DNA片段的精确剪切和克隆。
本文将从载体双酶切技术的原理、应用和优缺点等方面进行详细介绍。
一、原理载体双酶切技术的原理主要基于两种不同的限制性内切酶对DNA的特异性切割。
首先,选择两种能够在同一质粒上切割出相互兼容的黏性末端的内切酶,并确定其最佳反应条件。
然后,将目标DNA片段与质粒进行双酶切反应,使得目标DNA片段的末端与质粒的末端具有互补的黏性末端。
最后,通过DNA连接酶的作用,将目标DNA片段与质粒连接成重组质粒,实现目标DNA片段的克隆插入。
二、应用载体双酶切技术在分子生物学研究中有着广泛的应用。
首先,它可以用于重组质粒的构建,将外源基因插入到质粒中,用于基因克隆、表达和功能研究。
其次,还可以通过双酶切技术对质粒进行定向修饰,如引入点突变、插入序列或者删除特定片段等,用于研究基因的结构与功能。
此外,载体双酶切技术也被广泛应用于基因工程、蛋白质表达、基因组编辑等领域。
三、优缺点1. 优点(1)精准:通过双酶切技术可实现对DNA片段的精确切割和定向连接,保证了重组质粒的稳定性和可靠性。
(2)灵活:可以根据实验需要选择不同的限制性内切酶组合,实现对DNA的多样化操作。
(3)高效:相比传统的单酶切技术,载体双酶切技术能够提高DNA片段的连接效率和克隆成功率。
2. 缺点(1)操作复杂:双酶切技术需要充分考虑两种内切酶的选择、反应条件的优化及连接方法的调整,操作过程较为复杂。
(2)局限性:某些情况下可能无法找到适合的双酶切位点,导致目标DNA片段无法有效插入到质粒中。
(3)成本较高:需要购买多种限制性内切酶和连接酶,增加了实验成本。
综上所述,载体双酶切技术作为一种重要的分子生物学工具,在基因工程和分子生物学研究中发挥着重要作用。
随着技术的不断进步和完善,相信它将在更多领域展现出其巨大的潜力和价值。
酶切保护碱基表 引物设计原则
PCR引物设计原则PCR引物设计的目的是为了找到一对合适的核苷酸片段,使其能有效地扩增模板DNA序列。
因此,引物的优劣直接关系到PCR的特异性与成功与否。
要设计引物首先要找到DNA序列的保守区。
同时应预测将要扩增的片段单链是否形成二级结构。
如这个区域单链能形成二级结构,就要避开它。
如这一段不能形成二级结构,那就可以在这一区域设计引物。
现在可以在这一保守区域里设计一对引物。
一般引物长度为15~30碱基,扩增片段长度为100~600碱基对。
让我们先看看P1引物。
一般引物序列中G+C含量一般为40%~60%。
而且四种碱基的分布最好随机。
不要有聚嘌呤或聚嘧啶存在。
否则P1引物设计的就不合理。
应重新寻找区域设计引物。
同时引物之间也不能有互补性,一般一对引物间不应多于4个连续碱基的互补。
引物确定以后,可以对引物进行必要的修饰,例如可以在引物的5′端加酶切位点序列;标记生物素、荧光素、地高辛等,这对扩增的特异性影响不大。
但3′端绝对不能进行任何修饰,因为引物的延伸是从3′端开始的。
这里还需提醒的是3′端不要终止于密码子的第3位,因为密码子第3位易发生简并,会影响扩增的特异性与效率。
综上所述我们可以归纳十条PCR引物的设计原则:①引物应用核酸系列保守区内设计并具有特异性。
②产物不能形成二级结构。
③引物长度一般在15~30碱基之间。
④ G+C含量在40%~60%之间。
⑤碱基要随机分布。
⑥引物自身不能有连续4个碱基的互补。
⑦引物之间不能有连续4个碱基的互补。
⑧引物5′端可以修饰。
⑨引物3′端不可修饰。
不能选择A,最好选择T⑩引物3′端要避开密码子的第3位。
PCR引物设计的目的是找到一对合适的核苷酸片段,使其能有效地扩增模板DNA 序列。
如前述,引物的优劣直接关系到PCR的特异性与成功与否。
对引物的设计不可能有一种包罗万象的规则确保PCR的成功,但遵循某些原则,则有助于引物的设计。
1.引物的特异性引物与非特异扩增序列的同源性不要超过70%或有连续8个互补碱基同源。
双酶切连接反应之全攻略(

双酶切连接反应之全攻略(双酶切连接(Double Digestion)是一种常用的分子生物学技术,用于在DNA分子上选择性地切割两个特定的限制性内切酶位点。
它可以用于构建重组DNA,进行基因克隆,等等。
下面是一个全面的双酶切连接反应的攻略,包括实验前的准备工作,实验步骤和注意事项。
实验前的准备工作:1.获得限制性内切酶:选择两个互不相容的限制性内切酶。
确保这两个酶能够在相同的反应缓冲液中活性工作。
2.准备DNA底物:获得需要连接的DNA片段。
可以通过PCR扩增,限制性消化或DNA合成等方法获得。
3.选择连接载体:选择合适的连接载体,如质粒。
确保载体具有想要插入的目标基因的适当特性,如选择性标记物(如抗生素抗性基因)和启动子等。
4.验证限制酶位点:使用限制性内切酶图谱检测DNA片段和连接载体中的限制酶位点。
这有助于确定两个限制酶是否能够溶解目标DNA片段。
实验步骤:1.提取DNA:从细菌培养基中提取所需的DNA片段。
可以使用商用试剂盒或自制提取方法。
2.酶切反应:在适当的反应条件下,将需要连接的DNA片段和连接载体分别与两个限制性内切酶一起孵育。
反应条件包括酶的浓度,缓冲液的类型和pH值,反应温度和孵育时间。
3.酶停止反应:通过加入酶停止缓冲液或加热短暂孵育,停止酶切反应。
这样可以避免过度消化和限制酶反应继续进行。
4.凝胶电泳:将切割后的DNA片段经过琼脂糖凝胶电泳分析。
这一步骤可以检测酶切效率和特异性。
将反应样品和相应的对照样品(未经酶切)加载到琼脂糖凝胶上,然后运行电泳以分离DNA片段。
5.库仑凝胶纯化:根据所选的DNA片段大小,可以选择不同浓度的琼脂糖凝胶切片进行纯化。
将所需大小的DNA片段切割下来并进行库仑凝胶分离。
6.连接反应:将纯化的DNA片段与连接载体进行连接反应。
可以使用商业化的连接试剂盒,其中包含待连接DNA和连接载体之间的连接酶,以及其他必要的试剂。
7.转化:将连接后的DNA样品转化到合适的宿主细胞中。
限制性内切酶KpnⅠ和EcoRⅠ双酶切条件的优化

H A O F e n g
( 1 . D e p a r t m e n t o f B i o c h e mi s t r y L a b o r a t o r y , 2 . D e p a t r m e n t o f I mm u n o l o g y , J i l i n M e d i c a l C o l l e g e , J i l i n
免疫 教研 室 , 吉林 吉林
1 3 2 0 1 3 ) n I和 o I不 同的酶 切 体 系 , 酶切 含 有
摘 要 : 目的 探讨 n I 和E c o R I双 酶切 的优 化条 件 。 方法
上述 两种 限制 性 内切 酶 酶 切 位 点 的载 体 p E G F P — A n o 2 , D N A琼 脂 糖 凝 胶 电 泳检 测 酶 切 结果 , 以获 取 n I和
e r L, 1 I x L Kp n I d i g e s t i n g 1 I x g p l a s mi d f o r 1 h, t he n 1 I x L Ec o R I , 0. 01 % BS A a n d 5 L 1 0 × Bu f f e r H we r e a d de d
C i t y , J i l i n P r o v i n c e , 1 3 2 0 1 3, C h i n a )
ቤተ መጻሕፍቲ ባይዱ
A b s t r a c t :0b j e c t i v e T o i n v e s t i g a t e t h e o p t i m u m c o n d i t i o n s o f C O — d i g e s t i o n b y n I / E c o R I .Me t h o d s D i f f e r e n t
双酶切连接反应的注意要点

双酶切连接反应的注意要点1.选择适当的酶切位点:在进行双酶切连接反应之前,需要选择适当的酶切位点。
这些酶切位点应该满足以下几个要求:-位点不应该在目标DNA序列中出现,以避免酶切产生剪切产物;-两种酶切位点应该在目标DNA序列中相对靠近,以确保连接的有效性;-酶切位点的序列应该被两种酶同时识别和切割。
2.协议的优化:双酶切连接反应的协议需要进行优化,以确定最适合的条件。
一些重要的实验条件包括反应缓冲液的成分和浓度、酶的浓度和反应温度。
对于每个反应参数,应该进行范围的优化实验,以确定最佳的条件。
3.应用正确的酶切酶:双酶切连接反应需要同时使用两种酶来进行切割。
这些酶应该是互相兼容的,并且能够在相同的反应缓冲液中活性。
此外,酶的纯度和活性也应该得到保证,以确保酶切的效果。
4.反应的时间和温度:双酶切连接反应的时间和温度都需要进行优化。
反应时间应该足够长,以确保两种酶都能充分切割目标DNA序列,并且不会出现过度切割的情况。
反应温度也应该适中,通常在酶的推荐温度范围内选择。
5.质量控制:在完成双酶切连接反应之后,应该进行质量控制以确保反应的成功。
常用的方法包括琼脂糖凝胶电泳和DNA测序。
通过这些方法,可以检测连接产物的大小和纯度,并确认连接的正确性。
6.反应产物的处理:根据实验需要,对双酶切连接反应的产物进行处理。
这可能包括:-凝胶电泳分离:使用琼脂糖凝胶电泳分离不同大小的连接产物;-提取纯化:通过凝胶电泳或商业化学试剂盒,从琼脂糖凝胶中提取并纯化连接产物;-DNA测序:对连接产物进行测序,以确认连接的正确性。
总之,双酶切连接反应是一种常用的分子生物学技术,但在实验中需要注意一系列要点。
选择适当的酶切位点、优化实验条件、正确选择酶切酶、时间和温度的控制,以及进行质量控制和反应产物的处理,对于确保双酶切连接反应的成功至关重要。
这些注意要点的遵守可以确保实验结果的准确性和可靠性。
sumo酶切条件

sumo酶切条件
SUMO酶切条件可能会因酶的来源、底物和实验条件而有所不同,一般需要考虑以下几个因素:
1.酶与底物的比例:通常为1:100,即1μg酶对应
100μg底物。
2.酶切体系:通常包括融合蛋白1000μg、10x SUMO
Protease Buffer20μL、SUMO蛋白酶2μL和ddH2O (蒸馏水)定容至1000μL。
3.酶切条件:推荐在4℃下酶切过夜,用户可以根据
自己研究的目的蛋白进行摸索。
也可在25℃下酶切1h,SUMO标签的切割效率大于95%。
酶切是一项专业性较强的实验操作,需要根据实际情况进行调整和优化。
在进行酶切实验之前,建议仔细阅读酶的说明书和操作指南,严格按照实验步骤进行操作,以确保实验结果的准确性和可靠性。
酶切原理及步骤

酶切原理及步骤一、酶切原理1.酶切反应酶切反应是指使用酶作为催化剂,对底物进行切割或降解的反应。
在酶切反应中,酶的活性中心与底物特异性结合,通过催化作用将底物分解成小分子片段。
2.酶切位点酶切位点是指酶与底物特异性结合的部位。
不同的酶具有不同的酶切位点,通常由特定的氨基酸序列组成。
3.酶切动力学酶切动力学描述了酶切反应的速度和底物浓度之间的关系。
在一定条件下,当底物浓度高于某一阈值时,反应速度将达到最大值。
二、酶切步骤1.酶液准备在进行酶切实验前,需要准备适量的酶液。
根据实验需求选择合适的酶种类和浓度。
通常,酶液需要在冰箱中冷藏保存。
2.样品准备将待测样品进行预处理,以便与酶液混合后进行反应。
样品处理方法因实验而异,常见的处理方法包括细胞破碎、蛋白质提取等。
3.酶切反应设置将准备好的酶液和样品混合,加入适量的缓冲液(如pH 7.4的Tris-HCl缓冲液),设置反应温度和时间。
4.酶切反应温度和时间设置根据所选酶的活性要求和实验条件,设置适宜的反应温度和时间。
通常,适宜的反应温度为37℃,反应时间因底物种类和浓度而异。
5.反应终止和产物检测在反应结束后,需要终止反应并检测产物。
常用的终止方法包括加入酚/氯仿抽提、加热或加入抑蛋白剂。
产物检测方法因实验而异,常见的检测方法包括蛋白质印迹、电泳、光谱分析等。
三、酶切实验设计1.酶种选择2.根据实验需求选择合适的酶种类。
不同的酶具有不同的特异性,需要根据目标蛋白质序列选择具有相应酶切位点的酶。
同时还需要考虑所选酶的活性、稳定性和安全性。
3.实验条件优化在进行酶切实验前,需要对实验条件进行优化。
主要包括底物浓度、缓冲液pH值、离子强度、反应温度和时间等方面的优化。
通过调整实验条件可以提高产物的产量和质量。
4.产物检测方法选择根据实验需求选择合适的产物检测方法。
常用的检测方法包括蛋白质印迹、电泳、光谱分析等。
需要根据目标产物性质选择适宜的检测方法以便于后续分析。
分子克隆实验技术的使用中常见问题

分子克隆实验技术的使用中常见问题分子克隆技术是一种常用的实验技术,被广泛应用于分子生物学研究、基因工程以及生物医学等领域。
然而,在使用分子克隆技术进行研究或实验的过程中,常常会遇到一些问题。
本文将就分子克隆实验技术的使用中常见问题进行探讨,希望能帮助读者更好地应对这些挑战。
问题一:选择合适的克隆载体在进行分子克隆实验之前,选择合适的克隆载体是非常重要的一步。
克隆载体通常是一种容易携带外源DNA片段的可重复扩增的质粒或噬菌体。
然而,在选择合适的克隆载体时,需要考虑多个因素:1. 大小:载体的大小要适中,不宜过大或过小。
过大的载体可能导致操作不便、扩增困难,而过小的载体则可能限制载入的外源DNA片段的大小。
2. 复制起源:克隆载体应该具有一个可靠的复制起源,这样才能确保在细胞中稳定复制。
3. 选择标记:载体通常会带有一些标记基因,例如抗生素抗性基因。
在使用克隆载体进行实验时,选择合适的标记基因很重要。
问题二:限制性内切酶消化反应优化限制性内切酶消化是分子克隆实验中的重要步骤,用于切割DNA,生成所需的片段,并为之后的操作提供合适的DNA末端。
在进行限制性内切酶消化反应时,常见问题包括:1. 酶切位点不兼容:选择合适的限制性内切酶对于成功完成限制酶切非常重要。
如果酶切位点与目标DNA不兼容,将无法成功进行酶切。
2. 消化条件优化:酶切反应的条件包括酶切酶的浓度、反应温度、反应时间等。
为了获得理想的酶切效果,有时需要进行反应条件的优化。
3. 反切和星切现象:反切是指限制性内切酶在不应该切割的位点上产生切割作用,而星切是指一个酶切酶在DNA样品中的多个不同位点发生非特异性切割。
这些现象可能影响目标DNA片段的纯化和选择。
问题三:插入片段的PCR扩增在分子克隆实验中,为了获得目标DNA片段,常常需要进行PCR扩增。
然而,PCR扩增过程中可能会遇到以下问题:1. 扩增特异性:选择合适的引物是 PCR 扩增的基础。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
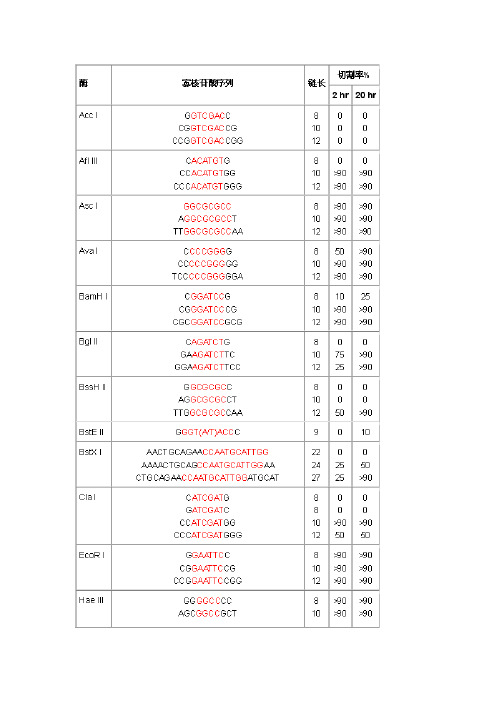
当建立内切酶酶切反应体系时有几个关键因素需要考虑。
比如如何在正确的反应体系中,加入适量的DNA、内切酶和缓冲液,就可以获得最佳酶切效果。
根据定义,在50μl体系中,1单位的限制性内切酶可以在60分钟内完全切割1μg的底物DNA。
上述酶、DNA与总反应体积的比值可以做为建立反应体系的参考数据。
但是,目前大多数科研人员会遵循下表中所列的标准反应条件,使用5-10倍的过量酶切割DNA,这样有利于克服由于DNA来源不同、质量和纯度不同而造成的实验失败。
“标准”反应体系
内切酶
•从冰箱取出后请一直置于冰上。
•酶最后加入到反应体系中。
•加入酶之前将反应混合物混匀,可以用移液枪上下吹打或轻弹管壁,然后在离心机中快速离心。
切忌振荡混匀!
•当切割超螺旋质粒和琼脂糖包埋DNA时,通常需要超过1unit/μg的酶量以达到完全酶切。
DNA
•避免酚、氯仿、酒精、EDTA、变性剂或过多盐离子的污染。
•甲基化的DNA会抑制某些酶的切割效率。
缓冲液
•使用终浓度为1X的缓冲液。
•根据实验需要加入终浓度为100μg/ml的BSA(1:100稀释)。
•在不需要BSA即可达到最佳活性的酶切反应中如果加入BSA也不会影响酶切效果。
反应总体积
•建议在50μl反应体系中消化1μg底物DNA。
•为避免星号活性,甘油浓度应<5%。
•加入内切酶(贮存于50%甘油中)的量应不超过总体积的10%。
•使用以下技术,内切酶的反应条件可能未达到最佳反应条件:克隆、基因分型、突变检测、基因定位、探针制备、测序和甲基化检测等。
•内切酶贮存液中的添加物(如:甘油和盐)和底物溶液中尚存的残余物(如:盐、EDTA 或乙醇)会导致小体积反应体系出现问题。
NEB提供了一系列高保真内切酶(方便建立反应体系。
下述为小体积反应体系反应指南。
酶切反应体系的选择
反应时间
•标准一小时。
•可加入过量的酶以缩短反应时间,或者使用能够快速酶切的内切酶。
•对于许多酶,都可以用更少单位的酶消化16小时。
终止反应
若消化后的DNA不需要进行后续的实验操作:
•用终止液终止反应【50%甘油、50mMEDTA(pH8.0)和0.05%溴酚蓝(NEB#B7021)】。
按每50μl反应体系中加入10μl的比例进行。
若消化后的DNA需要进行后续的实验操作:
•用热失活法。
•利用商业化的离心柱或酚/氯仿抽提去除内切酶。
贮存
•大多数内切酶建议保存在-20℃。
只有少数内切酶若贮存时间超过30天建议保存在-70℃。
详细的贮存信息请参阅内切酶使用说明书或目录。
•10X反应缓冲液和BSA贮存液也应保存在-20℃。
•不要将BSA直接加入到10X反应缓冲液中再冻存,因为这样BSA会产生沉淀。
稳定性
•在任何情况下,都应尽可能的避免将酶置于高于-20℃的温度下。
对照反应
如果在切割底物DNA时遇到了问题,建议加入以下对照实验:
•没有加入内切酶的实验DNA,以检测DNA制备和反应缓冲液中是否存在污染。
•加入了内切酶的对照DNA(含有多个已知内切酶切割位点的DNA,如LambdaDNA或腺病毒-2DNA),以检测内切酶的活性。
•如果对照DNA能够被切割而实验中的底物DNA不能,可以将这两种DNA混合在一起再进行酶切,以检测实验用的底物DNA中是否存在抑制反应的物质。
如果确实存在某种抑制因子(通常为盐、EDTA或酚),则混合之后对照DNA也不能被切割。
注意:由于某些酶与DNA结合的亲和性非常高,所以不能很好地与产物分离。
这样在进行琼脂糖凝胶电泳时就会出现弥散现象。
若出现这种情况,可以在反应结束后加入终浓度为0.1-0.5%的SDS,带型会更加清晰。
原文地址:/biotech/exp/molbio/DNA/2010/o8194426106.html。
