NCBI查基因 编码区 启动子区 外显子
基因启动子序列的查找

启动子序列的查找
3、确定启动子区与第一外显子区的长度; 进入P53基因mRNA的详细信息界面
.
启动子序列的查找
3、确定启动子区与第一外显子区的长度; 进入P53基因mRNA的详细信息界面,找到exon(第一外显子)的位置:1-174bp
.
启动子序列的查找
3、确定启动子区与第一外显子区的长度; •进入P53基因FASTA信息的界面,确定第一外显子的长度: •第一外显子位于基因5’端1-174bp的位置,由于基因为从右向左反向转录,第一外 显子位于启动子( 7687550-7689550bp )的左侧174bp:7687376--7687550bp; •第一外显子+启动子: 7687376--7689550bp
.
启动子序列的查找
4、下载启动子区与第一外显子区的序列;下载启动子区与第一外显子区的序列; 数据保存为FASTA格式,用word打开。
.
启动子序列的查找
4、下载启动子区与第一外显子区的序列; 去除序列中的段落标记:Word-查找替换-特殊格式-段落标记-全部替换
.
启动子序列的查找 1、在NCBI中查找所需的基因(以p53为例)
选择特定物种的P53基因,例如human.
.
启动子序列的查找
1、在NCBI中查找所需的基因(以p53为例) 获得human p53基因的详细信息
.
启动子序列的查找
2、确定此基因在染色体上的位置和转录方向(正向/反向); •在染色体上的位置: Chromosome 17 - NC_000017.11 •转录方向:从右向左,反向转录,说明启动子区与第一外显子区位于 此段基因的右侧。
.
基因序列查找
基因组区域,转录,产出:p53基因在不同转录本中的信息—点击GenBank
一步一步教你使用NCBI查找DNA

一步一步教你使用NCBI 查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST序列比对等最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html 在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
应用UCSC_Ensembl查找基因启动子(promoter)、内含子、外显子序列-表观遗传学论坛-生物秀论坛
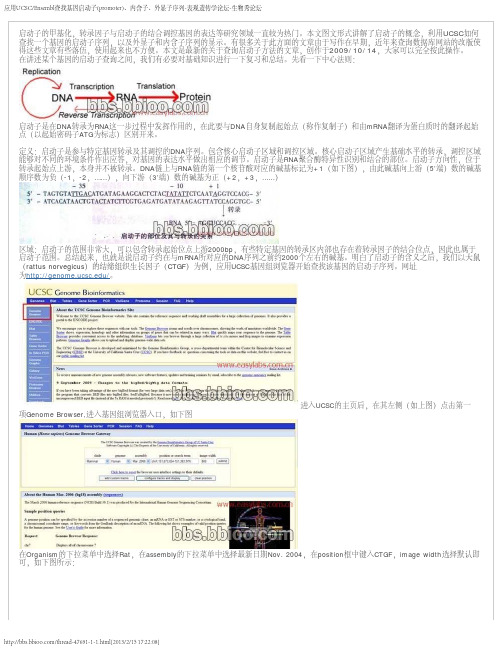
在讲述某个基因的启动子查询之间,我们有必要对基础知识进行一下复习和总结。
先看一下中心法则:启动子是在DNA转录为RNA这一步过程中发挥作用的,在此要与顺序数为负(-1,-2,……),向下游(3’端)数的碱基为正(+2,+3,……)区域:启动子的范围非常大,可以包含转录起始位点上游2000bp,有些特定基因的转录区内部也存在着转录因子的结合位点,因此也属于为/。
进入项Genome Browser,进入基因组浏览器入口,如下图在Organism的下拉菜单中选择Rat,在assembly的下拉菜单中选择最新日期Nov. 2004可,如下图所示:然后点击Submit,返回的页面如下:结果显示该基因的已知序列和相关mRNA序列,点击Known Gene中的第一个序列,出现包含这序列的图解概要。
为了获得这个区域更清晰的图像,可以点击紧靠zoom out的1.5X按钮,如下图:对于Known Genes(已知基因)和预测的基因路径来说,一般的惯例是以一个高的垂直线或块状表示每个编码外显子,以短的垂直线或块状表示5′端和3′端非翻译区。
起连接作用的内含子以非常细的线条表示。
翻译的方向由沿着细线的箭头指示。
本例的搜寻目的来说,默认设置不是理想的设置。
按照视图利用页面底部的Track Controls按钮,将一些路径设置为hide模式(即不显示),其他设置为dense模式(所有资料密集在一条直线上);另一些路径设置为full模式(每个特征有一个分开的线条,最多达300)。
在考虑这些路径内究竟存在那些资料之前,对这些路径的内容和表现做一个简要的讨论是必要的,许多这些讨论是由外界提供给UCSC的。
Ensembl Gene Predictions路径由Ensembl提供。
Ensembl基因通过许多方法来预测,包括与已知mRNA和蛋白质进行同源性比较。
若查询启动子区域,我们需要将Ensembl Genes选择为dense 或full模式,点击Refresh,即刷新,出现下图:图中多出了Ensembl Genes的预测路径,我们在红框中圈出。
利用Pubmed Map viewer查找基因序列

利用Pubmed Map viewer查找基因序列、mRNA 序列、启动子(Promoter)来源:dxy/lovelifeangel 发布时间:2009-09-22 查看次数:2234下面以大鼠(rattus norvegicus)的结缔组织生长因子(C T G F)为例讲述一下具体的操作步骤1.打开Map viewer页面,网址/mapview/index.html在search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的一个序列。
我也推荐大家使用这个序列。
4.点击上述三条序列第一条序列(即reference)对应的"Genes seq",出现新的页面,页面下方为:5.点击上图出现的“Download/View Sequence/Evidence ”,即下载查看序列等功能,结果如图所示:先对上面这张图做点简要的说明,在Sequence Format(序列输出格式)后面是一个下拉式选择菜单,默认的为FASTA 格式,还有一个是GenBank 格式。
我推荐大家选择GenBnak格式,因为这个格式提供了很多该基因的信息,而FASTA 格式只有基因序列。
6.在Sequence Format 后选择GenBank,然后点击下面的Display,目的基因的相关信息和序列就出现在眼前了。
一步一步教你使用 NCBI 查找DNAmRNAcDNAProteinpromoter引物设计BLAST 序列比对等

一步一步教你使用NCBI 查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST 序列最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
寻找启动子区域和预测转录因子结合位点

寻找promoter区域
•
genomes
• 在clade选择Mam的数据库,gene中输入ANKH
• 点击Tables
• 在track中选择RefSeq Genes,在output format中选择sequence
• 复制白底黑色区域即为promoter区域。
白底黑字 为启动子
区域
紫底黑字 为基因区
域
粉底黑字为编 码区,ATG为 启示密码子
寻找promoter区域
• 在前两张幻灯片中选择FASTA • 在右边Change region shown输入到 • Display options选择Show reverse complement • 可以直接得到FASTA格式的promoter核苷酸序列(似乎有一个bp的差距,可
• 点击get output。 选择genomic。
寻找promoter区域
• 选择Promoter/Upstream by 2000 bases • Exons in upper case, everything else in lower case外显子大写,其他小写
寻找promoter区域
寻找promoter区域
• /index.htmlhuman 输入 ankh • 选择Gene,点击 GeneID ENSG • 点击左边的Export data
寻找promoter区域
• 5 Flanking sequence 输入2000 • Options for FASTA sequence中Genomic选5 Flanking sequence, • deselect all • 点击Next(不管正反此法都适用)
• 选择Promoter/Upstream by 2000 bases • Exons in upper case, everything else in lower case外显子大写,其他小写
NCBI_功能详细介绍

NCBI_功能详细介绍NCBI(National Center for Biotechnology Information,国家生物技术信息中心)是一个提供生物医学和基因组学研究数据的资源库和数据库。
NCBI的主要目标是促进和推动生物医学研究的发展,并为科学家、医生和公众提供相关信息。
NCBI提供了各种各样的数据库和工具,用于存储、检索和分析生物医学和基因组学数据。
下面是一些NCBI提供的主要功能的详细介绍:2. GenBank:GenBank是一个基因序列数据库,存储了全球范围内的基因序列数据。
研究人员可以通过GenBank获得基因序列和相关信息,用于基因功能研究、进化分析和生物信息学研究。
3. BLAST:BLAST(Basic Local Alignment Search Tool)是一种常用的序列比对工具,用于比对给定的DNA、RNA或蛋白质序列与NCBI数据库中的序列。
BLAST可以帮助研究人员确定新序列的相关性,并找到与其相似的序列。
4. Entrez:Entrez是一个综合性的引擎,可以对NCBI的不同数据库进行全文。
研究人员可以通过Entrez进行文献检索、基因和蛋白质注释、序列比对等操作,方便地获取各类生物学数据。
5. PubChem:PubChem是一个化学物质数据库,存储了数百万种化合物的化学结构和相关信息。
研究人员可以通过PubChem化合物的属性、药理学和毒理学数据,以及相关的文献信息。
6. OMIM:OMIM(Online Mendelian Inheritance in Man)是一个遗传疾病数据库,提供了人类遗传疾病的基因和表型信息。
研究人员可以通过OMIM了解各种遗传疾病的发病机制、遗传模式以及相关基因的功能。
7. RefSeq:RefSeq是一个参考序列数据库,存储了各个物种的基因组和转录组序列。
RefSeq提供了基因的注释信息,包括基因的外显子、内含子、启动子、终止子等区域的序列。
NCBI使用方法介绍

NCBI使用方法介绍一、Map viewer查找基因序列,RNA,启动子下面以人的IL6(白细胞介素6)为例讲述一下具体的操作步骤1、A. 打开Map viewer页面,网址为在search的下拉菜单里选择物种,for后面填写你的目的基因。
2、B. 点击“GO”:C. 在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter:说明一下:1.1、染色体的红色区域即为你的目的基因所处位置。
1.2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的一个序列。
我也推荐大家使用这个序列。
1.3、点击上述三条序列第一条序列(即reference)对应的"Genes seq",出现新的页面,1.4、点击上图出现的“Download/View Sequence/Evidence ”,即下载查看序列等功能,先对上面这张图做点简要的说明,在Sequence Format(序列输出格式)后面是一个下拉式选择菜单,默认的为FASTA格式,还有一个是GenBank 格式。
我推荐大家选择GenBnak格式,因为这个格式提供了很多该基因的信息,而FASTA格式只有基因序列。
1.5、在Sequence Format后选择GenBank,然后点击下面的Display,目的基因的相关信息和序列就出现在眼前了。
在上述打开的网页中,你可以看到基因长度,基因序列,以及这个基因是如何被报道出来的等各种信息。
你会看到: mRNA join(3598..3678,3841..4031,5090..5203,5911..6057, 7803..8394) 这代表了从基因的3598位开始就是转录区了,即我们常说的mRNA 片断,由于内含子的存在,所以mRNA在DNA序列上分成了几段。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
问题:NCBI中怎样查找编码区/非编码区、起始密码子、启动子、外显子/内含子。
启动子
一般定义启动子,都是upstream 1000bp,downstream1000bp的那段序列。
或者根据你的实验。
你去ensemble,输入基因,找到exon,点开,在configuration里面选好flank多少bp 的序列,选好之后自动刷新,就出来了。
在序列里面,ensemble用不同的颜色标出来不同区域,5‘UTR之类的,还有exon,intron,转录起始位点等等,flank的区域就是你选的promoter 了。
开放阅读框
在分子生物学中,开放阅读框(Open Reading Frame, ORF)从起始密码子开始,是DNA序列中具有编码蛋白质潜能,一段无终止密码子打断的碱基序列。
