蛋白灰度分析总结-sssholy
蛋白质CD光谱定性解析_总结.
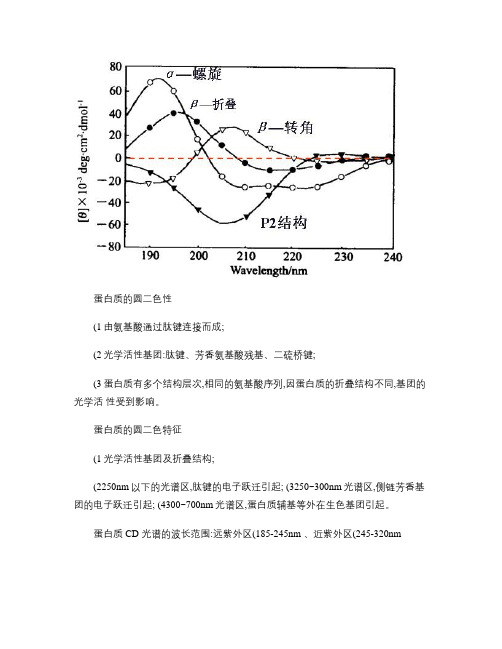
蛋白质的圆二色性(1由氨基酸通过肽键连接而成;(2光学活性基团:肽键、芳香氨基酸残基、二硫桥键;(3蛋白质有多个结构层次,相同的氨基酸序列,因蛋白质的折叠结构不同,基团的光学活性受到影响。
蛋白质的圆二色特征(1光学活性基团及折叠结构;(2250nm以下的光谱区,肽键的电子跃迁引起; (3250~300nm光谱区,侧链芳香基团的电子跃迁引起; (4300~700nm光谱区,蛋白质辅基等外在生色基团引起。
蛋白质 CD 光谱的波长范围:远紫外区(185-245nm 、近紫外区(245-320nm分子构象电子跃迁形式极值的波长[θ]*103 Deg.cm 2/dmolα-螺旋(α-helixn → π﹡π→ π﹡221~222nm(- 207~210nm(- 191nm(+ -38~-40 -36~-40 72~86 β-折叠(β-sheet n → π﹡π→ π﹡217~218nm(- 195~197nm(+-19~-21 28~42 β-转角(β-turnn → π﹡π→ π﹡227nm(- 弱 200~205nm(+ 192.5(-强无规卷曲 (random coilπ→ π﹡230nm(- 215~218nm (+195~202nm(-强-1.6 1~2 -25~-50圆二色谱法分析多肽二级结构圆二色谱是一种特殊的吸收谱, 它通过测量蛋白质等生物大分子的圆二色光谱,从而得到生物大分子的二级结构,简单、快捷,广泛应用在蛋白质折叠,蛋白质构象研究,酶动力学等领域。
圆二色谱紫外区段 (190-240nm , 主要生色团是肽链, 这一波长范围的 CD 谱包含了生物大分子主链构象的信息。
α-螺旋构象的 CD 谱在 222nm 、 208nm 处呈负峰,在 190nm 附近有一正峰。
β-折叠构象的 CD 谱,在 217-218nm 处有一负峰,在195-198nm 处有一强的正峰。
无规则卷曲构象的 CD 谱在 198nm 附近有一负峰,在220nm 附近有一小而宽的正峰。
蛋白质双向电泳图像分析

技术与方法蛋白质双向电泳图像分析3贾宇峰1) 林秋霞1) 郭尧君2)33 郭 鹞3) 刘少君1)33(1)军事医学科学院基础医学研究所神经生物学研究室,北京100850;2)中国科学院生物物理研究所,北京100101;3)第四军医大学预防医学系放射医学教研室,西安710032)摘要 随着人类基因组计划的接近完成,蛋白质组(proteome)研究成为新的热点.其中高分辨率的双向电泳(two2dimensional gel electrophoresis,22DE)技术使对组织或细胞的整个蛋白质组的综合分析成为可能.近年来这一技术有了很大的改进和提高,特别是图像分析系统,算法更为先进,功能日益强大,操作也更简便,为大规模研究提供了良好的工具.使用新一代的2D图像分析系统,对离体培养的雪旺氏细胞的蛋白质样品双向电泳结果进行了初步分析,探讨了在图像扫描、点检测、背景消除、匹配、结果报告和数据分析各步中的技术问题,并报告了进行2D图像分析的体会.关键词 双向电泳,蛋白质组,图像分析,软件,雪旺氏细胞学科分类号 Q51 双向电泳(two2dimensional gel electrophoresis, 22DE)技术是蛋白质组研究中最为广泛应用的蛋白质分离方法[1].而2D图像分析作为22DE的重要一步,其作用是评价和量化电泳结果.在蛋白质组研究中,从样品制备、第一向等电聚焦(IEF)、第二向SDS聚丙烯酰胺凝胶电泳(SDS2PA GE)、2D图像分析到基质辅助激光解析电离2飞行时间质谱仪(MALDI2TOF2MS)或液相色谱2串联质谱连用(LC2MS/MS)分析,整个技术过程环环相扣,更加复杂.在各个环节中,图像分析既要对上游各步的结果做出定性和定量的评价,将一个直观的蛋白质二维图谱数字化,还要为下一步的分析提供依据.特别是在表达蛋白质组学研究中,需要用图像分析软件通过正常和异常细胞、组织等电泳图谱间的匹配,找出差异蛋白质点:哪些点消失了?哪些点是新出现的?哪些点是相同蛋白,而表达水平有差异等等.将这些目标点切下,通过氨基酸组成分析、N端测序或质谱分析,确定是何种蛋白质,再结合图像分析数据和与内部和外部数据库比较的结果,做出结论.由此可见,2D图像分析在蛋白质组研究中是不可或缺的.正当人类基因组计划即将完成之时,越来越多的实验室开始涉足后基因组(post2genome)领域,蛋白质组研究正在升温.在一些科研机构和生物技术公司的努力下,22DE技术不断出现突破性进展, 2D凝胶的分辨率也越来越高.在这种情况下,简单的图像比较是远远不能满足研究需要的,图像采集系统和分析软件必须能够检测到最小的差别和获取最多的信息[1].随着22DE技术和计算机技术的发展,2D图像分析系统的性能有了很大的提高,高分辨率高速度的扫描仪如ImageScanner、STORM scanner和Model GS2710Imaging Densitometer等,再加上新一代功能强大的软件如ImageMaster2D、PDQuest[2]、MelanieⅡ[3,4]等,为大规模分析做好了准备.我们的实验室正在利用22DE技术开展神经生物学方面的蛋白质组研究工作,已经建立了全套22DE系统,并且应用图像扫描系统和2D图像分析软件对提取的雪旺氏细胞蛋白质样品电泳结果进行了初步分析研究. 3国家自然科学基金资助项目(39928015). 33通讯联系人. 刘少君Tel:010*********,E2mail:liusj@ 郭尧君Tel:010*********,E2mail:yaojun.g@ 收稿日期:2000204224,接受日期:20002062071 材料与方法111 22DE本文中22DE使用蛋白质样品提取自原代培养的雪旺氏细胞,上样量80μg(Bradford法[5], Unicam UV300紫外2可见分光光度仪定量);第一向IEF使用Immobiline TM p H3~10线性IP G预制凝胶条(购自Amersham Pharmacia Biotech Inc.),在IP G phor 电泳仪(APB公司)上完成,第二向SDS2PA GE使用自制SDS凝胶(250mm×180mm ×015mm,T=13%)[6,7],在MultiphorⅡ 电泳仪(L K B公司)上进行;Hoefer凝胶自动染色仪(APB公司)银染[8];染色完成后玻璃纸覆盖,室温下放置数天干胶.112 软件与硬件ImageMaster2D Elite3101(图像分析软件)和LabScan(扫描控制和分析前处理软件)是由Nonlinear Dynamics Ltd.开发,购自APB公司.工作站为联想奔月2000电脑,(Pentium II 350MHz CPU,64M内存,30G硬盘空间,17寸显示器,显示分辨率1024×768),Windows98操作系统.ImageScanner扫描仪(最高分辨率为9600dpi(H)×9600dpi(V)),购自APB公司. 113 图像采集与分析使用Labscan控制ImageScanner扫描图像, ImageMaster2D Elite3110分析.2 结果与讨论211 分析过程21111 图像扫描:在LabScan中,执行扫描.大多数情况下,300DPI(dot per inch)比较合适,但对于过大或过小的凝胶,就应该根据实际情况调节DPI值.不过相对于凝胶而言,缩小的图像会导致图像质量的损失或无法分辨;而放大的图像则会由于多余的象素而出现内插值(interpolated values),甚至会出现象素效应(pixellation),影响测定.因此扫描时图像与凝胶大小最好是1∶1.本文分析采用300DPI扫描的图像(图1a). 应该强调的是,强度校正(intensity calibration)是很重要的,特别是要使用图像做光密度测定时,一般是分析凝胶图像的第一步.强度校正可把以任意单位测得的强度值与实际的光密度(optical density)或散射密度(diffuse density)值匹配.这可以解决图像扫描设备的非线性应答问题,即图像中的象素值和实际光密度之间无线性关系.由于光密度仪能够直接提供图像的校正数据,用此种设备扫描时通常不需要校正[8].另外,也可用扫描软件校正图像.校正一般使用已知光密度的灰度尺(step wedge),即一组灰度呈梯度变化的条带.在软件中可定义灰度尺列表,添加和删除值.21112 点检测:可自动执行,同时也可调节灵敏度和算子大小,在高级设置中更有灵敏度、算子大小、背景和噪声四项供调节.使用点检测向导很方便,首先在整个图像上框选一片区域,进入下一步,出现九格窗口,各个窗格显示的是根据不同灵敏度和算子大小检测得到的效果图,选择最满意的窗格或直接调节下侧和右侧的灵敏度和算子大小,直到中间窗格的检测结果达到最佳而其他各窗格结果也较为满意为止,进入再下一步,就是消除背景,最后显示检测结果.上述过程中有两点需要说明,一是框选区域越小,图像处理时间越短,反之亦然;二是ImageMaster2D除有图像翻转功能外,不提供任何其他图像调整工具,因此提交分析的基本是扫描后的原始图像,一些杂质点和明显的瑕疵也会被误认为是蛋白质点,这些将在下面的手工编辑和背景消除中得到纠正.初步检测结果,包括蛋白质点的编号、面积、量、以及峰值用表格的形式列出,图像上所有检测到的点都可用同形色块覆盖、轮廓线圈围、十字线或标记等显示(图1b).自动检测完成后,仍有一些点未被识别出,还有一些是“假点”,另有一些因距离过近被识别成一个点,需要手工添加、删除或分割. ImageMaster2D中提供了一些很好用的编辑工具:画点、删除点、提高峰值、边缘增加、分割点、选择点等(图2).21113 背景消减:扫描后的图像一般都有不同程度的背景,从而影响蛋白质点的精确检测.一些2D图像分析软件有较强大的图像调整功能,可在分析前对图像作平滑、对比增强、消减背景等处理.而ImageMaster2D原则上在检测前不对图像作任何处理(除了图像翻转),而是在点检测完成后消减背景,共有五种方式:手工消减、边界最低强度消减、边界平均象素值消减、非点模式消减,后三种均是完全自动的,如果上述四种方式均不能满足要求,也可以直接编辑某个点的背景水平. 21114 匹配:上文已经提到,匹配是2D图像分析中很重要的一步.在ImageMaster2D Elite中,这也是一个交互式的过程.匹配时首先要创建参考凝胶,参考凝胶可以是要分析比较的一组凝胶中的一张,也可以是几张凝胶合并而成的平均胶.用户可以通过改变向量框(vector box)和搜索框(search box)的大小来操纵匹配.由于电泳过程中的一些影响因素的作用,即使是同一样品两次电泳图像之间也存在移位或扭曲.在这种情况下,可以调整上述两个参数得到较好的匹配效果(图3). 21115 结果报告:在图像分析的过程中,会产生大量的数据.数据的存储和输出是很重要的. ImageMaster2D在整个分析过程中,无论是图像窗口还是测量窗口的内容变化,随时都可以拷贝到剪贴板和文件,其中数据列表还可拷贝到Excel文档.检测结果还可生成凝胶报告或蛋白质点报告,包括图像和列表,并可存储为rtf文件.另外,软件还可将分析数据自动创建成网页,点击参考胶任何一个蛋白质点就可以访问相关数据.21116 1D和2D校正:可通过已知分子质量的标准蛋白确定凝胶上蛋白质点的大致的分子质量(M r),等电点(p I)可通过在第一向加入的等电聚焦标准蛋白(一系列分子质量相同而等电点不同的蛋白).2D 校正首先要创建一个蛋白质列表,这些蛋白质的特性,主要是分子质量和等电点都是已知的,并将这些蛋白质与凝胶上的点关联起来(即该种蛋白质在凝胶上形成的点),软件据此进行校正.21117 数据分析和数据库查询:在因特网上有许多蛋白质数据库和22DE 数据库,这些数据库中存储着不同来源的和不同形式的蛋白质信息.2D 分析软件基于分析所产生的蛋白质点的数据,再通过数据库中的查询,可以做出更全面的分析.软件现提供Biobase 、Swiss 2D Page 等5个数据库的查询.212 问题与展望21211 图像调整:扫描时,杂质和蛋白质点的拖尾会影响图像的质量,使分析效率与准确性大大降低.一些2D 图像分析软件有较强大的图像调整工具,可对图像作平滑、对比增强、消减背景等处理,如PDQuest 在点检测时,生成2D scan 、gel image 和gel spots 三幅图像,其中2D scan 是扫描的原始图像,gel spots 是经图像调整后提交检测的图像,gel image 是用于编辑的图像,也经过了图像调整.而ImageMaster 2D 原则上不对图像作任何处理.在实验初期,我们为了提高图像分析的效率,尝试使用Photoshop 510的图像调整工具和噪声滤镜消减背景和去除杂质点,但是发现图像调整后蛋白质点的量值(volume )明显变化.同时我们发现,即使不调整图像,通过优化灵敏度、算子大小、背景和噪声等变量的设置,在点检测时就可排除一些杂质点,其后的手工编辑也可再作甄别.21212 校正和标准化:影响双向电泳重复性的因素很多,包括样品制备与上样方式的不同,染色方法和扫描方式的差异等等.为了准确地比较分析,必须采用校正标准化的方法.首先是强度校正(intensity calibration ),在扫描前,使用灰度尺解决图像扫描设备的非线性应答问题.同时可把图像中的象素值转换为实际的光密度(optical density ,OD );在匹配时,可建立平均凝胶(averaged gel ).所谓平均凝胶,并非是真的凝胶,而是通过把一组凝胶经统计学处理合并在一起生成的模拟凝胶.创建平均凝胶可在同一样品的一组凝胶中生成一张有代表性的凝胶.如果将其作为参考凝胶进行匹配,则能得到更多的信息.21213 自动化:但是我们也应该看到,一张凝胶上大约总有一些点未被检测到,另外一些检测到的却是“假点”等等,这些都必须手工调整,非常耗时间费精力.另外,手工匹配也需要大量时间,而且会导致错配发生.因此,22DE分析软件的自动化程度还需提高,应向高分辨率、高灵敏度、智能化的方向发展.参 考 文 献1 Quadroni M,James P.Proteomics and automation.Electro2 phoresis,1999,20:664~6772 Collins P J,J uhl C,Lognonn′e J L.Image analysis of2D gels: considerations and in2sights.Cell Mol Biol,1994,40(1):77~833 Appel R D,Palagi P M,Walther D,et al.MelanieⅡ:a third2 generation software package for analysis of two2dimensional electrophoresis images:Ⅰ.Features and user interface.Electrophoresis,1997,18(15):2724~27344 Appel R D,Vargas J R,Palagi P M,et al.MelanieⅡ:a third2 generation software package for analysis of two2dimensional electrophoresis images:Ⅱ.Algorithms.Electrophoresis,1997, 18(15):2735~27485 Bollag D M,Rozucki M D,Edelstein S J.Protein Methods.2nd.USA:Wiley2Liss,Inc,626 G rg A,Boguth G,Obermaier C,et al.Two2dimensional polyacrylamide gel electrophoresis with immobilized p H gradients in the first dimension(IPG2Dalt):The state of the art and the controversy fo vertical versus horizontal systems.Electrophoresis, 1995,16:1079~10867 G rg A,Boguth G,Obermaier C,et al.Two2dimensional electrophoresis of proteins in an immobilized p H4~12gradient.Electrophoresis,1998,19:1516~15198 郭尧君.蛋白质电泳实验技术.北京:科学出版社.1999.142 Guo Y J.Experimental Techniques of Protein Electrophoresis.Beijing:Science Press,1999.142The Image Analysis of Two2Dimensional G el Electrophoresis3J IA Yu2Feng1),L IN Qiu2Xia1),GUO Yao2J un2)33,GUO Yao3),L IU Shao2J un1)33(1)Instit ute of Basic Medical Sciences,Academy of Military Medical Sciences,Beiji ng100850,China;2)Instit ute of Biophysics,The Chi nese Academy of Sciences,Beiji ng100101,China;3)Depart ment of Radiation Medici ne,The Fourth Military Medical U niversity,Xi’an710032,China)Abstract Proteome research has become a new hot spot in the post2genome era.High2resolution two2 dimensional gel electrophoresis(22DE),which provides the most comprehensive analysis system of the whole proteome,was highly improved in recent years.With the development of computer techniques,the powerful and user2friendly image analysis systems appeared to help high2throughput,large2scale proteomic ing new generation two2dimensional image analysis software,ImageMaster2D Elite,the2D gels of proteins extracted from cultured Schwann’s cells were processed.The analysis procedure,including image acquirement, spot detection,match,background subtraction,p I/M r calibration,analysis results report and database query, were reported and discussed.K ey w ords two2dimensional gel electrophoresis,proteome,image process,software,Schwann’s cell 3This work was supported by a grant from National Nature Science Foundation of China(39928015). 33Corresponding author.Liu S J,Tel:86210266930304,E2mail:liusj@ Guo Y J,Tel:010*********,E2mail:yaojun.g@ Received:April24,2000 Accepted:J une7,2000。
灰度值分布 python-概述说明以及解释

灰度值分布python-概述说明以及解释1.引言1.1 概述在数字图像处理中,灰度值分布是指图像中各个灰度级别的频率分布情况。
灰度值是指图像中每个像素点的亮度,通常以0(黑)到255(白)之间的整数表示。
灰度值分布描述了图像中不同灰度级别出现的次数,可以帮助我们了解图像的亮度分布情况和特征。
在本文中,我们将探讨灰度值分布的概念及其在Python中的处理方法。
通过分析灰度值分布,我们可以更好地理解图像的特征和信息,为图像处理和分析提供重要的参考。
Python作为一种强大的编程语言在处理灰度值分布方面具有灵活性和便捷性,我们将介绍一些常用的处理方法和技巧,并通过实例分析展示其应用效果。
通过本文的学习,读者将能够深入了解灰度值分布的重要性及其在图像处理中的应用,为进一步探索图像处理领域打下基础。
1.2 文章结构:本文主要分为引言、正文和结论三个部分。
在引言部分,我将对灰度值分布这一主题进行概述,并介绍文章的结构和目的。
在正文部分,我将详细介绍灰度值分布的概念,以及在Python中处理灰度值分布的方法。
最后,通过实例分析帮助读者更好地理解和应用所学的知识。
在结论部分,我将对全文进行总结,探讨灰度值分布在实际应用中的前景,以及展望未来可能的发展方向。
整个文章结构清晰,逻辑性强,旨在帮助读者全面了解和掌握灰度值分布的相关知识。
1.3 目的本文旨在探讨灰度值分布在图像处理中的重要性和应用。
通过深入分析灰度值分布的概念和Python中处理灰度值分布的方法,读者将能够更好地理解和应用这一概念。
通过实例分析的方法,我们将演示如何利用Python来处理和分析灰度值分布,以便读者能够在实际应用中运用这些知识。
最终,希望本文能够帮助读者更好地理解和利用灰度值分布这一重要的图像处理概念,为他们在实际应用中取得更好的效果提供帮助。
2.正文2.1 灰度值分布的概念在数字图像处理中,灰度值分布是指图像中每个像素点的灰度级别在整个图像范围内的分布情况。
10分钟Get!大牛教你用imageJ对Westernblot条带进行灰度分析!
10分钟Get!大牛教你用imageJ对Westernblot条带进行
灰度分析!
科研人员做的western blot实验一般需要对其结果扫描后进行灰度分析,一般使用的软件为Image 。
采用Image J 软件对蛋白质印迹实验结果(Western Blot)进行灰度半定量分析,是分子生物学实验中比较常用的方法,可实现对WB实验结果进行准确、简单和快速的分析。
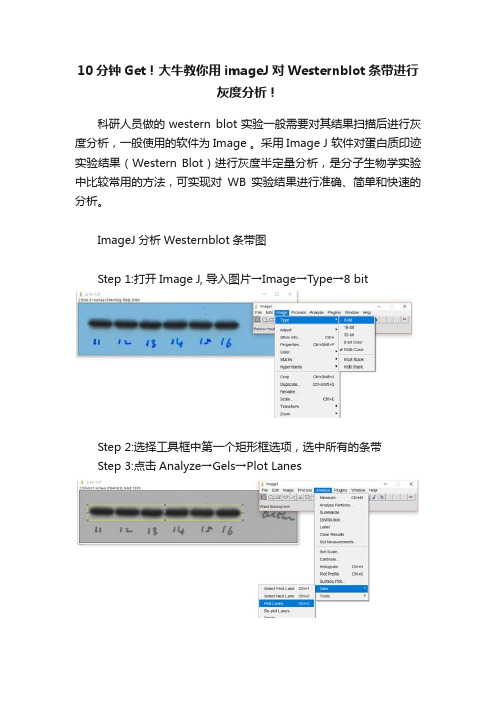
ImageJ分析Westernblot条带图
Step 1:打开Image J, 导入图片→Image→Type→8 bit
Step 2:选择工具框中第一个矩形框选项,选中所有的条带
Step 3:点击Analyze→Gels→Plot Lanes
Step 4:选用工具框中的直线工具,将开口波峰封闭
Step 5:选用工具框中的魔棒工具,点击相应的波峰,相应area值
ImageJ分析Westernblot条带图
Step 1:打开Image J, 导入图片。
Image→Type→8 bit
Step 2:扣除背景P→rocess→Subtract Background→选择50 pixels和Lightbackground
Step 3:设定参数→Analyze→Set Measures→按照下图勾选参数
Step 4:设定参数→Analyze→Set scale→unit of length选项改为pixels
Step 5:图像分割→Edit→Invert→选用椭圆或自定义工具选中条带
Step 6:Analyze→Measure→得到 IntDen值Step 7:重复Step5、6,得到所有条带的IntDen值
以上实用干货来源于课程
30分钟精通Image J图片处理。
Western Blot图片灰度分析 -Photoshop
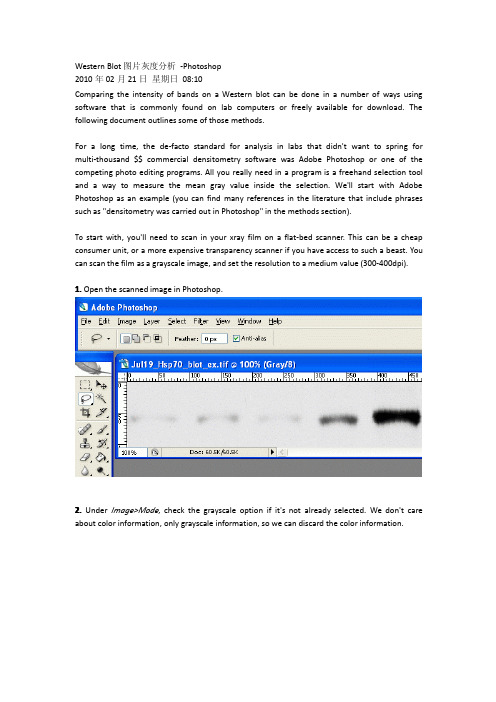
Western Blot图片灰度分析-Photoshop2010年02月21日星期日08:10Comparing the intensity of bands on a Western blot can be done in a number of ways using software that is commonly found on lab computers or freely available for download.The following document outlines some of those methods.For a long time,the de-facto standard for analysis in labs that didn't want to spring for multi-thousand$$commercial densitometry software was Adobe Photoshop or one of the competing photo editing programs.All you really need in a program is a freehand selection tool and a way to measure the mean gray value inside the selection.We'll start with Adobe Photoshop as an example(you can find many references in the literature that include phrases such as"densitometry was carried out in Photoshop"in the methods section).To start with,you'll need to scan in your xray film on a flat-bed scanner.This can be a cheap consumer unit,or a more expensive transparency scanner if you have access to such a beast.You can scan the film as a grayscale image,and set the resolution to a medium value(300-400dpi).1.Open the scanned image in Photoshop.2.Under Image>Mode,check the grayscale option if it's not already selected.We don't care about color information,only grayscale information,so we can discard the color information.3.Under Image>Adjustments,select Invert(or press Ctrl+I).Now the dark parts of the film are light,and the light parts are dark.This is useful later,as the high-expression bands,which are dark on the film,will have high numerical values when we measure them.When photo programs report darkness/lightness values,the dark points have values near zero,and the light points have values that max out at255.The inverted scan4.Choose the lasso tool from the tool palette.5.On the first band,use the lasso tool to draw a line all the way around the edges of the first band.This is where your judgement comes in to play,determining where the edges of the band are,and what is simply background.6.For Photoshop CS2/3:If the histogram window is not open by default,go to Window>Histogram to open the histogram.In newer versions of Photoshop(CS2,CS3)there is a small arrow in a circle in the upper right corner of the histogram window.Click on this and choose'expanded view'to show the values for your selection.For Photoshop v.5+6:Go to Image>Histogram to display the histogram for the current selection.7.The histogram information includes a"Mean"value and a"Pixels"value.Record these two numbers for your selection.The Mean value is the average gray value(from0to255)for the area inside your selection.The Pixels value is the number of pixels contained in your selection area. Bands with high expression are typically darker,but also often larger in size,so we want to know both of these attributes for our comparison later.8.On your picture,use the lasso tool to draw around the next band.Record the Mean and Pixel values for this selection.Repeat for the rest of your bands,including your standard.。
(整理)WesternBlot图片灰度分析-Photoshop.
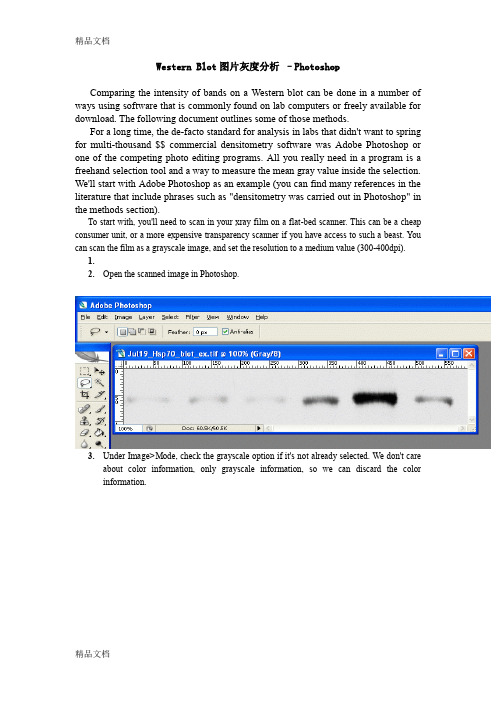
Western Blot图片灰度分析–PhotoshopComparing the intensity of bands on a Western blot can be done in a number of ways using software that is commonly found on lab computers or freely available for download. The following document outlines some of those methods.For a long time, the de-facto standard for analysis in labs that didn't want to spring for multi-thousand $$ commercial densitometry software was Adobe Photoshop or one of the competing photo editing programs. All you really need in a program is a freehand selection tool and a way to measure the mean gray value inside the selection. We'll start with Adobe Photoshop as an example (you can find many references in the literature that include phrases such as "densitometry was carried out in Photoshop" in the methods section).To start with, you'll need to scan in your xray film on a flat-bed scanner. This can be a cheap consumer unit, or a more expensive transparency scanner if you have access to such a beast. You can scan the film as a grayscale image, and set the resolution to a medium value (300-400dpi).1.2.Open the scanned image in Photoshop.3.Under Image>Mode, check the grayscale option if it's not already selected. We don't careabout color information, only grayscale information, so we can discard the color information.4.5.Under Image>Adjustments, select Invert (or press Ctrl+I). Now the dark parts of the filmare light, and the light parts are dark. This is useful later, as the high-expression bands, which are dark on the film, will have high numerical values when we measure them.When photo programs report darkness/lightness values, the dark points have values near zero, and the light points have values that max out at 255.The inverted scan6.Choose the lasso tool from the tool palette.7.8.On the first band, use the lasso tool to draw a line all the way around the edges of the firstband. This is where your judgement comes in to play, determining where the edges of the band are, and what is simply background.6.For Photoshop CS2/3: If the histogram window is not open by default, go toWindow>Histogram to open the histogram. In newer versions of Photoshop (CS2, CS3) there is a small arrow in a circle in the upper right corner of the histogram window. Click on this and choose 'expanded view' to show the values for your selection.For Photoshop v.5+6: Go to Image>Histogram to display the histogram for the current selection.9.The histogram information includes a "Mean" value and a "Pixels" value. Record thesetwo numbers for your selection. The Mean value is the average gray value (from 0 to 255)for the area inside your selection. The Pixels value is the number of pixels contained in your selection area. Bands with high expression are typically darker, but also often larger in size, so we want to know both of these attributes for our comparison later.8. On your picture, use the lasso tool to draw around the next band. Record the Mean and Pixel values for this selection. Repeat for the rest of your bands, including your standard.。
三种分析蛋白结构域的方法
三种分析蛋白结构域的方法蛋白质是生物体中最重要的分子之一,它在细胞功能和生物过程中起着关键作用。
了解蛋白质的结构和功能对于揭示其生物学功能以及药物设计和治疗疾病的机制至关重要。
在过去的几十年里,科学家们开发了多种方法来分析蛋白质的结构域。
在本文中,将会介绍三种常见的方法:X射线晶体结构学、核磁共振(NMR)结构学和电子显微镜(EM)。
首先,X射线晶体结构学是分析蛋白质结构的金标准方法之一、该方法利用蛋白质晶体对X射线的衍射反射来解析其结构。
通过测量反射强度和角度,可以确定蛋白质中原子的位置。
X射线晶体结构学具有高分辨率和高精度的优点,可以得到详细的原子级别的结构信息。
然而,该方法需要获得高质量的晶体样品,并且晶体的生长和数据采集过程相对复杂和耗时。
其次,核磁共振(NMR)结构学是另一种常用的蛋白质结构分析方法。
NMR方法使用核磁共振信号来确定蛋白质中原子的位置和相对于周围环境的动力学信息。
与X射线晶体结构学不同,NMR方法可以在溶液中研究蛋白质结构,而无需晶体。
NMR结构学在研究小分子或无法结晶的蛋白质方面具有优势。
然而,由于蛋白质的体积和复杂性,NMR在解析大型蛋白质结构上仍然具有挑战性。
此外,NMR的分辨率相对较低,对于一些较小的结构域的分析可能不够准确。
总结而言,X射线晶体结构学、NMR结构学和电子显微镜是目前常见的分析蛋白质结构的方法。
每种方法都有其独特的优势和限制,需要根据研究的需求和样品的特性选择合适的方法。
随着技术的不断进步,这些方法的分辨率和解析能力将不断提高,为我们进一步理解蛋白质结构和功能提供更多的工具和方法。
磷酸化wb灰度值
磷酸化wb灰度值摘要:1.磷酸化wb 灰度值的背景和意义2.磷酸化wb 灰度值的处理方法和流程3.磷酸化wb 灰度值的具体应用场景4.磷酸化wb 灰度值在实验中的优势和局限性5.磷酸化wb 灰度值的未来发展方向正文:磷酸化wb 灰度值是指在蛋白质磷酸化Western Blot(WB)实验中,通过仪器测量得到的灰度值。
磷酸化Western Blot(WB)是研究蛋白质磷酸化修饰的一种重要技术手段,通过该技术可以检测特定蛋白质在样本中的表达情况。
然而,在实验过程中,磷酸化wb 灰度值的处理和分析成为实验结果准确性的关键环节。
首先,磷酸化wb 灰度值的处理方法和流程包括以下几个步骤:(1)实验样本的收集和处理(2)WB 实验操作(3)仪器测量灰度值(4)灰度值的处理和分析(5)结果的呈现和解读磷酸化wb 灰度值的具体应用场景包括:(1)磷酸化蛋白质的表达和调控研究(2)药物筛选和作用机制研究(3)信号通路的研究(4)疾病相关蛋白质磷酸化修饰的研究磷酸化wb 灰度值在实验中的优势和局限性:(1)优势:灰度值可以定量磷酸化蛋白质的表达水平,为实验结果提供客观、准确的量化数据。
(2)局限性:实验过程中可能出现的操作误差、仪器误差等因素,会影响磷酸化wb 灰度值的准确性。
未来,磷酸化wb 灰度值的研究方向将主要集中在:(1)提高实验操作的标准化程度,降低误差(2)开发新的数据分析方法,提高结果的准确性和可靠性(3)结合其他实验技术,深入研究磷酸化蛋白质的功能和作用机制综上所述,磷酸化wb 灰度值作为研究蛋白质磷酸化修饰的重要指标,在实验过程中具有重要作用。
ELISA灰区的设置及质控方法
ELISA灰区的设置及质控方法ELISA灰区的设置及质控方法ELISA测定的阳性判断值,通常是将大量阴性和阳性人群的测定数据进行统计分析后确定的,其确定依据为判断结果的假阳性和假阴性率最低。
在阳性判断值周围一定范围内之外的测定结果可归为可疑,亦即ELISA测定的“灰区”。
“灰区”的大小可用统计学方法确定。
测定结果处于“灰区”的样本,可通过确认试验或追踪检测来确定到底是阳性还是阴性。
”。
“灰区”的大小可用统计学方法确定??具体是怎么确定的,。
或者大概粗略的方法计算是怎么样的呢??高手在哪呢ELISA“灰区”是把定量分析的正常值范围引入定性分析而建立的概念。
灰区的设置有二种:(1)C.O×(1±C),C为该试剂的批内C (一般在15-20%间);(2)C.O±2S,S为实验室做室内质控ROC的S。
另外,个人认为:ELISA“灰区”对于不同项目和应用的领域不同,其设置不能一概而论。
根据美国疾病控制中心(CDC)对美国Ortho 的抗HC检测的ELISA试剂盒进行研究,发现只有检验结果S/CO≥3.8时,高度预示HC抗体真阳性状态;若检验结果S/CO<3.8,存在较高的假阳性。
在血站系统的献血员抗HC筛查用上述两种灰区的设置方法没有什么不可;如果临床实验室抗HC的灰区也按前者设置,势必存在较多的假阳性,如何设置灰区应该是值得研究和商榷的。
这位老师好,不好意思,本人愚昧,还不是很清楚这两种方法在实际的应用,可否举一个小例子。
比如HBsAg>0.5是阳性,如果测量得吸光度是0.4的话怎么判断,结果是阴性,但是是有很明显颜色的。
在血站系统这样的血就不敢用,怎么解决呢,我们经常遇到测量阴性但是有颜色的标本,用不同厂家的试剂或是同种试剂重复实验还是一样,又担心存在弱抗体的问题。
怎么解决呢,万分感激国内试剂测HBSAG的cutoff值一般不会达到0.5,除非不做空白。
其cutoff的公式一般为NC*2.1。
ImageJ和Graphpad如何对WesternBlot条带灰度分析【干货】每日生物评论
ImageJ和Graphpad如何对WesternBlot条带灰度分析【干货】每日生物评论摘要手把手教你利用Image J和Graphpad软件对Western Blot条带进行灰度分析和柱状图构建,看艾美捷干货分享。
WB是研究蛋白表达的一个经典方法。
对于一些时间点或者是不同组织蛋白表达量的分析就涉及到量的变化。
一些凝胶成像软件带有此分析工具,比如Quantity One,Bandscan,Gel-Pro Analyzer等成像系统专用软件。
除了这些软件,还有一个比较简单的综合性质图像处理软件Image J可以很方便的进行灰度分析。
而且Image J是开源性质的免费软件,可以在其官网直接下载使用。
对Western Blot条带进行数值化有两种方法——灰度分析和光密度分析。
灰度是计算机图像分析仪根据图像颜色深浅分为256个级别。
灰度值越小物体颜色越深。
光密度是指光线通过溶液或某一物质前的入射光强度IO与该光线通过溶液或物质后的透射光强度Ib比值的对数OD=log(IO/Ib);图像分析中,入射光和透射光强度分别被切片上最亮区域的平均灰度值和待测目标平均灰度值取代,则图像分析系统的光密度值=log(切片标本最亮区域的平均灰度值(g0)/待测目标的平均灰度值(g))。
光密度值越大,物体颜色越深,阳性物质相对含量越大。
对于灰度值来说,切片标本制作时染色时间长短,以及测量时显微镜照明光源电压的大小对其影响很大。
而光密度是一个比值。
是通过计算一张切片标本最亮区域的平均灰度值与该切片标本中待测目标的平均灰度值的比值,再根据数学公式计算而来的,所以受切片标本染色时间长短及照明光源电压关系很小。
然而有研究实践显示,灰度值与光密度值二者之间存在着线性关系,都可以用来进行WB定量分析。
同样,Image J软件进行WB条带定量分析时也存在两种方法,此处只列举一种方法,具体步骤如下。
1、下载安装,并打开Image J软件。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
蛋白灰度分析总结
By sssholy 2012-08-26
(stejun@)
蛋白灰度分析软件有:Image-Pro Plus,Quantity One,Image J等~~
但总的来说:
(1
能准确反应蛋白灰度~~
但需要靠魔棒或轨迹法选取目的蛋白区域,有一定的主观性,不过蛋白灰度测定本身就是半定量分析-----测量值本身就没有一定标准,不同软件测量值不同,即使相同软件重复测量数值也不一定相同,因此只要测定数值能反应目的蛋白变化趋势,测量值相对偏差不大即可~~~
(2)Quantity One 有泳道-轨迹测定法,等高线/手绘选取目的区域测定法等方法。
ⅰ.泳道-轨迹测定法:
过程:先剔除一系列背景,再选择泳道(lane),然后分析条带(band),最后使用高斯建模得出灰度分析值(Gauss Model trace)
特点:感觉比较科学,重复性好,但十分繁琐,而且有时不能正确反应灰度(我亲测过,亲~~)。
ⅱ.等高线-手绘选取目的区域测定法:通过等高线或手绘选取目的蛋白区域,最简便,但重复性不太好。
(3)Image J 最坑爹~~~,它用魔棒选取区域测定灰度,可是有时很难选中所需区域,而且方法繁琐,不推荐!!
在此,只总结综合性能最好的Image-Pro Plus(IPP)蛋白灰度分析方法~~~~
Image-Pro Plus 分析蛋白灰度教程
说明:目的蛋白的灰度=目的蛋白IOD值/ 相应内参IOD值
1.使用Photoshop剪切出目的区域(只含western bolt条带,如图),保存为
TIFF格式文件。
2.Image-Pro Plus打开文件,剔除背景:
(1)放大镜放大目的区域
(2)M easure---calibration---intensity---options----image,
选择背景最大value值------ok-------ok----system----close
(3) Measure---count/size---measure---select measurements,选择IOD为测量值,调整结果显示方式:Options-label style改为mesurement,选择IOD为显示值。
(4)接下来有2种方法选择目的蛋白区域(即找出AOI,area of interest, IPP核心元件):魔棒和手绘轨迹法。
魔棒:首选方法,客观科学,适合于蛋白条带轮廓清楚且各蛋白条带不融合
目的蛋白区域)。
手绘轨迹法:主观性较强,当魔棒无法选择单一条带时才使用。
(5)count/size---edit-----convert AOI to object---得出IOD值
(6)若测下一条带,则点NEW AOI按钮,再按以上方法选择AOI
(7) 可以count/size---view---measurement data显示或输出测量值
图示过程如下:
魔棒法:
Or
手绘轨迹法:
得出测量IOD值:
下图显示的即为目的蛋白IOD值
重复测量其他条带:
相关介绍:
255。
