质谱蛋白质互作结果峰值数据处理
Co-IP质谱分析

百泰派克生物科技
Co-IP质谱分析
Co-IP质谱分析可以用来鉴定天然状态下蛋白质间的相互作用,也可以用来发现与
已知蛋白互作的未知蛋白。
百泰派克生物科技针对Co-IP样品蛋白的提取,表达和纯化的优化方案可确保获得高质量的诱饵和猎物蛋白,为客户提供优质的Co-IP蛋白互作分析服务及对免疫共沉淀CO-IP获取的蛋白进行蛋白质谱分析。
Co-IP(免疫共沉淀)
免疫共沉淀(co-immunoprecipitation,简称Co-IP)是一种检测蛋白质与蛋白质
相互作用的实验方法。
在实验中,首先要使用非离子型表面活性剂来裂解细胞以更好的保留蛋白质与蛋白质的相互作用,然后加入某一蛋白(诱饵蛋白)的专一性抗体,抗体会与诱饵蛋白结合使诱饵蛋白沉淀,同时细胞内与诱饵蛋白相互作用结合在一起的其它蛋白也会沉淀来下,即共沉淀。
由此可以看出,Co-IP可以用来检测
体内蛋白质之间的互作,也可以用来发现体内与某已知蛋白有相互作用的新蛋白质。
Co-IP质谱分析
当Co-IP实验中需要确认已知互作蛋白是否是预想的蛋白质,或需要鉴定发现的新的互作蛋白时,可以结合质谱进行分析。
Co-IP质谱分析指的就是对Co-IP实验中
沉淀下来的蛋白质进行质谱分析。
免疫共沉淀(Co-IP)结合质谱可以用于分析稳
定或者强烈的蛋白质间的相互作用,可以鉴定某个目标蛋白质(诱饵蛋白)与另一个蛋白质之间的相互作用或者寻找与某个目标蛋白质(诱饵蛋白)存在相互作用的未知蛋白质。
第二章 4 蛋白质鉴定技术--质谱数据分析 end 2
(1)固定基线算法
这种固定基线方法简单易行,但最大的 不足就在于没有根据用户当前提供的数 据寻找基线,而固定一个基线,无法考 虑当前图谱的强度分布。
(2)固定峰数法
这个方法可以看作是有效峰挑选的方法.这 种方法将谱峰按照强度高低进行排序,保留 固定数日的峰作为信号峰(如200个),其他的 都作为噪音舍弃
(3)窗口基线法(b)
基线—采用局部线性拟合的方法来去除
(3)窗口基线法(b)
(4)窗口基线法和固定峰数法 的结合
例如,在图谱的质量方向上,每隔50Da 开一个窗口,在每个窗口中选择峰强最 高的5个谱峰作为信号峰,其他谱峰作为 嗓声而舍弃。 如 Mascot 通过在每一个固定范围内 (14Da)保留一个质峰的方法加强了对 小丰度值质峰的选取,以获得与离子 系列相对应的质峰阶梯。
识别质谱中的容易区分的噪声
4)谱峰中心化(Peak centroiding)
轮廓质谱图--->棒状质谱图
质谱数据的预处理
1 原始质谱数据的预处理 2 谱峰中心化后的质谱数据的预处理
2 谱峰中心化后的质谱数据的预处理 串联质谱中对肽序列鉴定有用的信息只 是很少量的几十个重要离子的单同位素 (monoisotope)谱峰 但通常一个Q-TOF类型的串联质谱对应 的.dta文件里包含300-5000甚至更多的 谱峰。 质谱中绝大多数的谱峰对应仪器的物理 噪声以及离子的同位素谱峰。 因此,有必要对质谱数据进行预处理
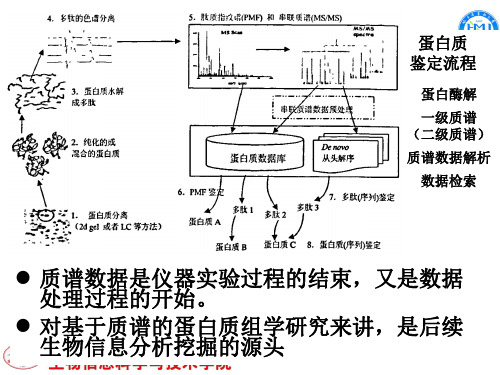
蛋白质 鉴定流程
蛋白酶解
一级质谱 (二级质谱) 质谱数据解析 数据检索
质谱数据是仪器实验过程的结束,又是数据 处理过程的开始。 对基于质谱的蛋白质组学研究来讲,是后续 生物信息分析挖掘的源头
植物ip-ms原理

植物ip-ms原理
植物IP-MS(Interaction Partner-Mass Spectrometry)技术是一种用于研究植物分子互作的实验方法。
IP-MS原理主要包括以下几个步骤:
1. 表达载体构建:将目标基因与荧光标签(如GFP)融合,构建表达载体。
将构建好的载体转化到植物材料中,使目标基因表达并产生荧光信号。
2. 植物材料处理:将转化后的植物材料进行适当处理,如切片、破碎等,使细胞内的目标蛋白暴露出来。
3. 蛋白提取:利用蛋白提取试剂盒从植物材料中提取目标蛋白。
提取过程中,要注意保持蛋白的完整性,以便后续实验。
4. 免疫沉淀:利用特异性抗体与目标蛋白结合,将其沉淀下来。
此步骤可用于筛选与目标蛋白相互作用的蛋白。
5. 质谱分析:将沉淀下来的蛋白进行质谱分析,获取蛋白质组分及其相对丰度信息。
6. 数据处理与分析:通过质谱数据分析,找出与目标蛋白相互作用的蛋白,并建立分子互作网络。
IP-MS技术具有较高的准确性,可以同时研究多个基因之间的相互作用。
与其他分子互作技术相结合,IP-MS技术有助于深入了解植物分子互作的机制,为研究植物生长发育、抗病性、逆境响应等生物学问题提供重要线索。
蛋白质互作网络的构建与调节

蛋白质互作网络的构建与调节蛋白质是生命体内最为重要的分子之一,其功能与规模十分复杂。
蛋白质之间不仅仅是单一的相互作用,而是一个动态的“互作网络”,因此,构建并调节蛋白质互作网络变得至关重要。
一、构建蛋白质互作网络蛋白质互作网络是指蛋白质之间的相互作用关系,包括但不限于蛋白质-蛋白质相互作用、蛋白质-核酸相互作用、蛋白质-小分子相互作用等。
在构建蛋白质互作网络时,我们可以运用多种技术手段,如蛋白质质谱、蛋白质数组、蛋白质结构生物学等。
1. 蛋白质质谱技术蛋白质质谱技术是指将蛋白质样品经过多种分离技术处理后进行质谱分析,从而得到蛋白质组成成分、分子量和其它信息。
蛋白质质谱技术已广泛应用于建立蛋白质相互作用的“互作图谱”。
2. 蛋白质数组技术蛋白质数组技术,又称为功能性蛋白质芯片技术,是指将大量可溶性或膜蛋白质或其片段在芯片表面上固定,通过其特异性配对,对多组蛋白质之间的相互作用进行高通量的分析。
蛋白质数组技术可用于发现新的蛋白质相互作用,特别是用于分析细胞位点相互作用,有助于研究蛋白质-蛋白质相互作用的抑制剂。
3. 蛋白质结构生物学技术蛋白质结构生物学技术是指运用X射线衍射、核磁共振、电子显微镜等技术对蛋白质的原子结构进行测定,以获得在“识别-配对-响应”过程中发生的蛋白质相互作用的详细信息。
二、调节蛋白质互作网络蛋白质互作网络的调节主要包括以下几方面:调节蛋白质的表达水平、酶促作用和小分子抑制剂等。
1.调节蛋白质的表达水平调节蛋白质的表达水平是指通过基因调控、转录后修饰和翻译后修饰等方式,对蛋白质表达量进行调节。
这种调节方式可以制约蛋白质互作网络的构建和演变,并最终影响组织与器官等高等生物层次的生理行为。
2.调节蛋白质酶促作用蛋白质的酶促作用是指分解或合成蛋白质时参与反应的酶的作用。
酶促作用可以影响蛋白质在互作网络上的分布与演变情况。
此外,调节酶促作用可以降低或提高蛋白质互作网络中的特定蛋白质浓度,从而实现对蛋白质互作网络的调控。
蛋白质互作网络的分析方法

蛋白质互作网络的分析方法随着生物学研究的不断深入,人们逐渐认识到,生物体内的蛋白质之间相互作用是一个极为复杂的网络系统。
在这个复杂的系统中,蛋白质的互作网络扮演着至关重要的角色。
因此,如何分析蛋白质互作网络成为了当下研究的热点之一。
本文将介绍一些常用的蛋白质互作网络的分析方法。
1. 蛋白质互作网络构建方法在分析蛋白质互作网络之前,我们需要先构建一个蛋白质互作网络。
蛋白质互作网络的构建有两种方法,一种是实验方法,即利用生化实验手段鉴定蛋白质之间的相互作用;另一种是计算机模拟方法,使用已有的大量数据,通过计算机模拟得到网络图。
在实验方法中,有很多技术可用来鉴定蛋白质之间的相互作用,如酵母双杂交技术、质谱联用技术、蛋白质芯片技术等。
这些技术可以在不同的尺度下对蛋白质进行特定的分析和鉴定,从而获得关于蛋白质互作网络的具体信息。
而在计算机模拟方法中,利用大量的生物学数据建立蛋白质互作网络模型。
这个模型是通过先验信息和计算机算法进行构建的,并可以通过进一步的数据挖掘和分析不断完善。
2. 蛋白质互作网络的基本特征构建蛋白质互作网络后,我们需要对其进行分析和挖掘,以了解蛋白质互作网络的基本特征。
简单来说,分析蛋白质互作网络可以分为三个方面:网络的统计学特征、网络的拓扑结构和节点的生物学意义。
网络的统计学特征可以描述网络的规模、密度、直径、平均短路径等性质,有助于研究网络的规律和本质。
网络的拓扑结构可以描述网络的层级、聚类系数、中心度等等,反映了网络的组织和结构特点。
而节点的生物学意义则是探究蛋白质在其所处网络中的作用和功能。
3. 蛋白质互作网络的应用蛋白质互作网络的研究不仅有助于深入了解复杂的生物系统,而且具有多种实际应用。
其中,最常见的应用之一是疾病研究。
人类疾病的发生往往是多个基因和蛋白质的互作导致的,通过对蛋白质互作网络的分析可以研究疾病发生的机理和治疗方法。
此外,蛋白质互作网络还可以用于药物筛选、基因编辑、进化分析等方面,具有广泛的科研和医学价值。
蛋白质互作网络的拓扑与功能分析方法

蛋白质互作网络的拓扑与功能分析方法蛋白质是生物体内功能最为重要的分子之一,它们通过与其他蛋白质相互作用形成复杂的互作网络,调控生物体内的各种生理过程。
理解蛋白质互作网络的拓扑和功能在揭示细胞活动的机制和生物学过程中具有重要的意义。
本文将介绍一些常用的蛋白质互作网络拓扑与功能分析方法。
首先,研究者通常使用高通量的蛋白质相互作用筛选实验,例如酵母双杂交法或质谱联用技术,来鉴定蛋白质互作网络中的相互作用关系。
这些方法可以帮助我们了解蛋白质网络的组成和拓扑结构。
然而,由于实验技术的局限性和复杂性,筛选出的相互作用数据可能存在一定的假阳性和假阴性结果。
因此,为了准确地分析蛋白质互作网络,需要进行数据质量控制和筛选,以排除不可靠的数据。
一种常用的蛋白质互作网络拓扑分析方法是节点中心性分析。
节点中心性是衡量网络中节点(蛋白质)重要性的一种指标。
常用的节点中心性指标包括度中心性、介数中心性和接近中心性。
度中心性表示节点与其他节点直接连接边的数量,是最直观的节点中心性指标。
介数中心性表示节点在网络中的中介性,即节点在其他节点间的传递信息上起到的桥梁作用。
接近中心性表示节点与其他节点之间的平均最短距离,其值越小,说明节点在网络中更为集中或更为重要。
通过计算这些指标,可以识别出网络中的重要节点,从而深入研究其功能和调控机制。
另一种常用的分析方法是模块性分析。
蛋白质互作网络通常是由多个功能相关的子网络(模块)组成的。
模块性分析方法可用于发现网络中的模块,并对模块内的蛋白质进行功能注释。
其中一个常用的模块性分析方法是基于模块度的算法,其基本思想是通过比较网络内部和网络随机模型中的边分布来度量模块化结构的好坏。
此外,还有一些基于聚类分析或社区检测的算法,可以将网络中相互作用密切的蛋白质聚合到一起形成模块。
此外,功能富集分析也是分析蛋白质互作网络功能的重要方法之一。
功能富集分析通过比较网络中的蛋白质与数据库中的已知功能关联,来识别蛋白质互作网络中的富集功能。
itc 蛋白互作
itc 蛋白互作摘要:一、ITC蛋白互作技术简介1.蛋白互作研究的重要性2.ITC技术的原理二、ITC实验流程1.样品准备2.ITC反应液的配制3.ITC实验装置的搭建4.实验条件的设置5.实验数据的处理与分析三、ITC技术的优缺点1.优点a.高灵敏度b.高分辨率c.操作简便d.可重复性好2.缺点a.对蛋白质的大小和形状要求较高b.需要标准品进行校准c.存在非特异性结合四、ITC技术在生物医学研究中的应用1.蛋白质复合物的研究2.药物筛选与开发3.信号通路的研究4.疾病相关蛋白互作的研究正文:一、ITC蛋白互作技术简介蛋白质是生命活动的基本单位,它们通过相互作用与其他蛋白质或分子共同完成生命过程。
了解蛋白质之间的相互作用对于研究生命现象具有重要意义。
免疫沉淀结合质谱(IP-MS)和热转换化(ITC)等技术被广泛应用于蛋白质互作研究。
其中,ITC技术以其高灵敏度、高分辨率、操作简便和可重复性好等优点受到科研工作者的青睐。
ITC技术基于热力学原理,通过测量蛋白质结合过程中热量的变化来检测相互作用。
当两个蛋白质结合时,如果它们之间存在热力学稳定的相互作用,那么在加热过程中,反应混合物的热量会发生变化。
通过测定热量变化,可以计算出蛋白质之间的结合常数Ka和Kd,从而评估它们之间的相互作用强度。
二、ITC实验流程1.样品准备:首先需要从细胞或组织中提取蛋白质,然后通过凝胶电泳或离心等方法纯化蛋白质。
2.ITC反应液的配制:准备一定浓度的蛋白质样品,然后与等量的已知浓度的其他蛋白质混合,形成反应液。
3.ITC实验装置的搭建:将反应液放入ITC仪器中,设置温度和体积等实验条件。
4.实验条件的设置:根据实验需求,设置一系列不同的温度和时间条件,进行实验。
5.实验数据的处理与分析:实验过程中,ITC仪器会记录热量变化,通过数据处理和分析,得到蛋白质结合常数Ka和Kd。
三、ITC技术的优缺点1.优点a.高灵敏度:ITC技术可以检测到纳摩尔级别的蛋白质结合,对于研究低亲和力相互作用具有重要意义。
蛋白质谱结果
蛋白质质谱是一种用于研究蛋白质组成、结构和功能的技术,它可以通过分析蛋白质在不同条件下的表达量、修饰状态、相互作用等信息,揭示生物体内蛋白质的动态变化和功能。
蛋白质质谱的结果通常以蛋白质的质量和数量为基础,可以通过以下几个方面来描述:
1.蛋白质的定量:蛋白质质谱可以测定蛋白质的相对丰度和绝对丰度,通过比较不同实验组之间的蛋白质表达量,可以发现差异表达的蛋白质,从而揭示生物体内蛋白质的动态变化。
2.蛋白质的鉴定:蛋白质质谱可以鉴定蛋白质的种类和亚型,通过比较不同实验组之间的蛋白质组成,可以确定哪些蛋白质发生了变化,并进一步探究其功能和相互作用。
3.蛋白质的修饰:蛋白质质谱可以检测蛋白质的修饰状态,例如磷酸化、甲基化、乙酰化等,通过分析蛋白质的修饰状态,可以揭示蛋白质的功能和调控机制。
4.蛋白质的相互作用:蛋白质质谱可以检测蛋白质之间的相互作用,例如蛋白质复合物的组成和结构,从而揭示蛋白质之间的相互作用网络和功能。
综上所述,蛋白质质谱的结果可以提供关于蛋白质组成、结构和功能的重要信息,有助于深入研究生物体内的蛋白质动态变化和功能。
蛋白质互作网络及其功能分析
蛋白质互作网络及其功能分析蛋白质是细胞机体中最重要的分子之一,其作用几乎贯穿生物体内所有的生理活动。
在细胞内,蛋白质并不是孤立存在的,而是形成一种复杂的网络。
这个网络被称为蛋白质互作网络。
蛋白质互作网络是由许多蛋白质之间的相互作用构成的,这些相互作用形成了包括酶促反应、信号转导、分子运输等各种进程在内的许多生命活动。
蛋白质互作网络的发现最早可以追溯到20世纪50年代。
之后,随着技术的不断进步和大规模测序的出现,人们发现蛋白质的相互作用关系远比最初想象的要复杂。
为了更好地理解蛋白质互作网络的功能,人们采用了许多方法来研究这个网络。
蛋白质互作网络的研究方法通常包括以下几种:1. 晶体学方法:通过蛋白质晶体学技术,研究蛋白质中基本单位的构成和蛋白质之间的相互作用。
2. 方法:利用质谱技术,分析蛋白质相互作用时的质量变化,进一步发现蛋白质之间的相互作用。
3. 免疫共沉淀方法:利用特定的抗体沉淀靶蛋白质,然后进行蛋白质间的筛选。
4. 两杂交法:通过将两类蛋白质基因分别融合成一个新的融合蛋白,从而检测相互作用的发生。
5. 生物信息学方法:运用计算机技术,建立计算模型,对组成蛋白质互作网络的蛋白质相互作用进行预测和分析。
这些研究方法为我们更加深入地理解蛋白质互作网络的功能提供了基础。
随着技术的进步,越来越多的蛋白质被发现,并逐渐加入到这个庞大的网络中。
这个网络呈现出复杂的结构,包括层次分明的网络,互通有无的互联关系以及动态演化的特性。
其中,许多蛋白质被认为具有“中枢性”,是整个蛋白质互作网络的关键节点。
这些关键节点在蛋白质网络中起着极其重要的作用,例如传递信号、参与代谢等生物过程。
此外,蛋白质互作网络还具有许多其他的特性。
比如,网络中的许多蛋白质形成了基因家族,这些基因家族具有相同或相似的蛋白质结构和组成。
在网络中,这些基因家族经常出现在同一个模块中,从而实现某些特定的生物功能。
总体来说,蛋白质互作网络的研究可以帮助我们更好地理解细胞或整个生物体的基本生物学过程,并可用于寻找药物靶标、疾病治疗和其他生物学应用。
质谱分析蛋白质的相互作用
蛋白质间的相互作用存在于生物体每个细胞的生命活动过程中,互交叉形成网络,成细胞中一系列重要生理活动的基础。
其中,多数蛋白质是通过与配体分子结合或者是作为1个大的生物复合体的一部分,与细胞完整性维持、遗传物质复制、基因表达调控、信号转导、免疫应答等一系列生命过程。
研究蛋白质间相互作用的方式和程度,将有助于蛋白质功能的分析、疾病致病机理的阐明和治疗和新型药物的开发等众多难题的解决。
因此,确定蛋白质间相互作用关系、绘制相互作用图谱已成为蛋白质组学研究的热点。
近年来有许多方法被用于蛋白质相互作用的研究,酵母双杂交技术,免疫共沉淀技术,串联亲和纯化技术,化学交联技术,蛋白质芯片技术,荧光共振能量转移技术,噬菌体展示技术等。
酵母双杂交技术Fields和song等首先在研究真核基因转录调控中建立起来的,是在真核细胞中检测蛋白质与蛋白质之间的相互作用的方法。
该系统是通过两个分别称之为“诱饵蛋白”和“捕获蛋白”的融合蛋白形成一个完整的转录激活因子,从而激活报告基因的表达,通过在营养缺陷型培养基上生长或呈现显色反应来检测系统的功能。
酵母双杂交系统可在全基因组规模上进行蛋白质一蛋白质相互作用高通量的研究。
免疫共沉淀技术免疫共沉淀是利用抗原和抗体的特异性结合以及细菌的Protein A或G特异性地结合到免疫球蛋白的Fc片段的现象开发出来的方法。
其基本原理是,在细胞裂解液中加入抗兴趣蛋白的抗体,孵育后再加入与抗体特异结合的结合于Agarose珠上的Protein A或G,若细胞中有与兴趣蛋白结合的目的蛋白,就可以形成这样一种复合物:“目的蛋白—兴趣蛋白—抗兴趣蛋白抗体—Protein A或G”,经变性聚丙烯酰胺凝胶电泳,复合物又被分开。
然后经免疫印迹或质谱检测目的蛋白。
这种方法得到的目的蛋白是在细胞内与兴趣蛋白天然结合的,符合体内实际情况,得到的结果可信度高。
这种方法常用于测定两种目标蛋白质是否在体内结合;也可用于确定一种特定蛋白质的新的作用搭档。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
质谱蛋白质互作结果峰值数据处理
质谱蛋白质互作结果峰值数据处理通常包括以下步骤:
峰识别:使用质谱软件进行峰识别,提取出每个样品中的峰。
峰对齐:对于多个样品的数据,需要进行峰对齐,使得相同化合物的峰在所有样品中的位置一致。
峰定量:通过计算峰面积或峰高度,对每个峰进行定量。
峰过滤:过滤掉信号噪声较大的峰,以及信号强度较弱的峰。
数据归一化:对于多个样品的数据,需要进行归一化,使得每个样品的总离子流量一致。
统计分析:使用统计软件进行差异分析,找出不同样品中的显著性差异峰。
在处理质谱蛋白质互作结果峰值数据时,需要根据实际情况选择合适的质谱软件和统计软件,并且注意数据的质量控制,确保数据的准确性和可靠性。
