组织或细胞的Real-time PCR protocol
realtimepcr的名词解释

realtimepcr的名词解释Real-time PCR的名词解释Real-time PCR,即实时定量聚合酶链反应,是一种在生物分子研究领域被广泛应用的技术。
它能够在短时间内检测和定量分析DNA或RNA的含量,因此在基因表达研究、微生物检测、病原体诊断等领域具有重要的意义。
首先,我们来了解PCR的基本原理。
聚合酶链反应(PCR)是一种能够在体外扩增DNA分子的技术。
它利用DNA聚合酶酶活性,在一系列变温反应过程中,通过酶催化,把待测DNA片段的特定区域进行放大,从而获得足够数量的目标DNA。
这种技术的应用使得我们能够快速、准确地研究和分析DNA的相关信息。
与传统PCR不同,Real-time PCR在PCR过程中实时监测反应体系中的荧光信号,以实现对DNA量的定量和计量。
这主要通过添加合成的DNA探针或荧光标记的引物,以及具有对应荧光信号的探针来实现。
在PCR反应进行过程中,荧光信号将与DNA量成正比。
PCR结束后,计算机软件会分析和显示实时荧光信号的扩增曲线,从而确定样品中目标DNA的起始量。
Real-time PCR的优势在于其高灵敏度、高选择性和高分辨率。
相对于传统PCR,Real-time PCR不需要后续的电泳分析,节省了实验时间,减少了实验操作和处理的复杂性。
此外,由于实时监测PCR反应过程中的信号变化,Real-time PCR能够提供更精确的计量和定量结果。
Real-time PCR有多种应用,其中包括基因表达研究。
通过实时监测特定基因的表达水平,我们可以了解基因在不同条件下的转录变化情况,从而理解其在生物过程中的功能和调控机制。
此外,Real-time PCR还可以用于微生物检测。
通过设计特异性的引物,可以快速、精确地检测病原微生物的存在与否,为临床诊断和疾病防控提供帮助。
除了这些应用外,Real-time PCR还在病原体诊断和检测方面发挥着重要作用。
传统的病原体检测方法往往需要培养和鉴定,周期较长,诊断结果需要等待数天甚至更长时间。
Real-Time PCR操作手册 0904

7 合成引物: 送往 Sangon 合成引物,大约需三个工作日。
8 引物稀释: 原液 100 μM 工作液 5 μM mixture Forwad primer Reverse primer ddH2O
5 μl 5 μl 90 μl
三) 推荐反应体系
试剂 2 × SYBR Green PCR Master mix cDNA (1-100 ng RNA 反转录所得 cDNA) H2O 5 μM Primer mix 终体积
此时可以设计 Real-time PCR 反应程序,点击兰色行的加号添加反应循环步骤,点击白色行加号添加每 一个循环内的步骤。 上图为 real-time PCR 的默认程序, ① 将第二个循环中第一步 95℃的时间调整为 15 秒,第二步改为 60℃(这些条件都需要根据所用的试 剂不同做出相应的调整), ② 另外我们需要添加熔解曲线步骤,点击红圈标出的加号,添加第三个循环,并把 dwell time 改为 1, setpoint 列改为 95℃, ③ 再点击加号添加第四个循环,并把 dwell time 改为 1,setpoint 列改为 55℃,
5 选择引物序列: 打开 Map 标签页,查看找到的引物序列,此时引物有红蓝两色显示,在 optimal primer pairs only 前
打勾,则只显示红色并基本上按得分高低排列引物,此时尽可能选择靠近 3’端的引物;再打开 Result 标签页,复制 Forwad primer 和 Reverse primer 序列到 Word 文档中,比对引物查看其是否跨越至少 1 个 外显子,再打开 Primer 标签页查看选中的序列的罚分值,若引物正好跨越外显子,且得分比较低(红 色基本可用)则可用。
Real-Time PCR 操作手册
实时荧光定量PCR(Real-Time-PCR)实验流程

实时荧光定量PCR(Real-Time PCR)实验流程一、RNA的提取(详见RNA提取及反转录)不同组织样本的RNA提取适用不同的提取方法,因为Real-Time PCR对RNA样品的质量要求较高,所以,正式实验前要选择一款适合自己样品的提取方法,在实验过程中要防止RNA 的降解,保持RNA的完整性。
在总RNA的提取过程中,注意避免mRNA的断裂;取2ug进行RNA的甲醛变性胶电泳检测,如果存在DNA污染时,要用DNase I进行消化(因为在处理过程中RNA极易降解,建议体系中加入适量RNA酶抑制剂)。
二、DNase I 消化样品RNA 中的DNA用DNase I 消化DNA组份加量模板(RNA) 10ugRNase Inhibitor 4ulDNase I buffer 10ulDNase I 10ulDEPC处理H2O 至100ul混匀,37℃90min三、RNA琼脂糖凝胶电泳1.1%的琼脂糖凝胶电泳凝胶的配制:1)称取琼脂糖0.45g放入三角瓶中,向其中加入4.5ml的10×MOPS缓冲液和39.5ml 的DEPC水,放微波炉里溶化。
2)待冷却到60摄氏度左右时,加入1ml甲醛,摇匀(避免产生气泡)。
倒入凝胶板上凝固30min。
2.取各个RNA样品4µl,加入6×RNA电泳上样缓冲液2µl混匀,加入变性胶加样孔中。
3.120V电压下电泳25min。
用凝胶紫外分析仪观察,照相保存。
4.RNA电泳结果如下图所示。
可见28S和18S两条明亮条带,无DNA条带污染。
四.RNA反转录为cDNA反转录程序(以MBI的M-MLV为例) 组份加量(20ul体系) 加量(40ul 体系)模板(RNA) 0.1~2.5ug(根据条带的亮度适当调整) 3ug(根据条带的亮度适当调整)引物T18(50uM)(或其他引物) 2.0ul 4.0ulDEPC处理H2O 至12.5ul 至25ul混匀,70℃5min,立即冰浴5*buffer 4.0ul 8.0uldNTP(10mM) 2.0ul 4.0ulRNase Inhibitor 0.5ul 1.0ul混匀,37℃5minM-MLV 1.0ul 2.0ul42℃60min ,70℃10min反转录引物的选择与Real-Time PCR引物设计的要求1)随机六聚体引物:当特定mRNA由于含有使反转录酶终止的序列而难以拷贝其全长序列时,可采用随机六聚体引物这一不特异的引物来拷贝全长mRNA。
RealTimePCRProtocol

RealTimePCRProtocolReal Time PCR 基本操作步骤及注意事项1、质粒标准品的制备1、1实验材料人粪便基因组DNA2X Taq PCR Master Mix (NP公司)小量质粒抽提试剂盒(OMEGA公司)pEASY-T3克隆试剂盒(全式金公司)感受态细胞DH-5α(全式金公司)LB培养基(按《分子克隆》配)氨苄青霉素Amp: 100mg/mlX-Gal: 20mg/mlIPTG: 24mg/mlTrans 2K Plus DNA Marker(全式金公司)1、2过程1、2、1 以人粪便基因组DNA为模板,目的菌的16s rRNA 引物为实验引物,PCR产生该菌的目的片段,并通过琼脂糖凝胶电泳检测PCR结果。
PCR体系:20ul 体系2X Taq PCR Master Mix 10ulForward primer 1ulReverse primer 1ulTemplate 1uldd H2O 7ulPCR热循环参数:Tem Time step cycle1 94℃5min2 94℃30s3 60℃30s4 72℃1min5 72℃7min 2 306 4℃∞PCR结束后,取出PCR产物,通过琼脂糖凝胶电泳(1%)检测PCR结果,样品和Marker 上样量均为4ul,100V约40min。
注:(1)PCR体系中所加上游引物和下游引物的量根据引物浓度和实验要求调整,所加模板的量根据模板和实验的具体要求调整,不足的用水补齐(2)PCR热循环参数中,具体的温度、时间和循环数根据所扩增片段大小调整。
(3)若目的条带单一,可直接进行下游实验,若有非特异条带,先进行切胶回收并纯化,用纯化的PCR产物进行下游实验。
1、2、2 采用pEASY-T3 Cloning Kit,取PCR阳性样品,把目的片段连入pEASY-T3载体。
克隆反应体系的建立如下:在1.5ml离心管中依次加入溶液:PCR产物2ul (根据PCR产物量可适当增减,最多不超过4ul)pEASY-T3 Cloning V ector 1ul轻轻混合,室温(20-37℃)反应5分钟。
real-time PCR手册

PCR手册(2010-07-13 23:29:38)转载标签:杂谈由于Real-time qPCR的众多优点,现在已经是生命科学领域的一项常规技术。
越来越多的研究文章中涉及RT-PCR的实验,也基本上被real-time qPCR所代替。
由于real-time aPCR 输出的数据不同于常规的PCR 电泳检测,很多没有做过real-time qPCR的研究者常常感到高深莫测,不知从何入手;甚至一些做过次实验的研究者也会对数据处理分析感到迷惑,不知所措。
本文就从real-time qPCR的发展史说起,包括real-time qPCR的原理,实验设计,实际操作,数据分析,常见问题解答五个方面,手把手教你从各个方面了解real-time qPCR,彻底的从菜鸟到高手!一、Real-time qPCR发展史Real-time qPCR就是在PCR扩增过程中,通过荧光信号,对PCR进程进行实时检测。
由于在PCR扩增的指数时期,模板的Ct 值和该模板的起始拷贝数存在线性关系,所以成为定量的依据。
由于常规的PCR的缺点,real-time qPCR 由于其操作简便,灵敏度高,重复性好等优点发展非常迅速。
现在已经涉及到生命科学研究的各个领域,比如基因的差异表达分析,SNP检测,等位基因的检测,药物开发,临床诊断,转基因研究等。
在Real-time qPCR技术的发展过程中,定量PCR仪的发展起了至关重要的作用。
1995年,美国PE公司(已经并入Invitrogen公司)成功研制了Taqman 技术,1996年推出了首台荧光定量PCR检测系统,通过检测每个循环的荧光强度,通过Ct值进行数据分析。
从而荧光定量PCR获得广泛应用。
现在的定量PCR 仪有ABI7000、7300、7500,7700、7900HT、StepOnePlusTM、StepOneTM、PRISM@StepOneTM系列;BIO-RAD的CFX96、iCycler iQ5@、MyiQ@、MJ Research Chromo4TM Opticon 系列;Stratagene MxTM系列;Roche LightCycler@系列;Eppendorf Masercycler@;Corbett Rotor-GeneTM;Cepheid SmartCycler@和BIOER的LineGene系列。
Real-time_PCR_原理介绍
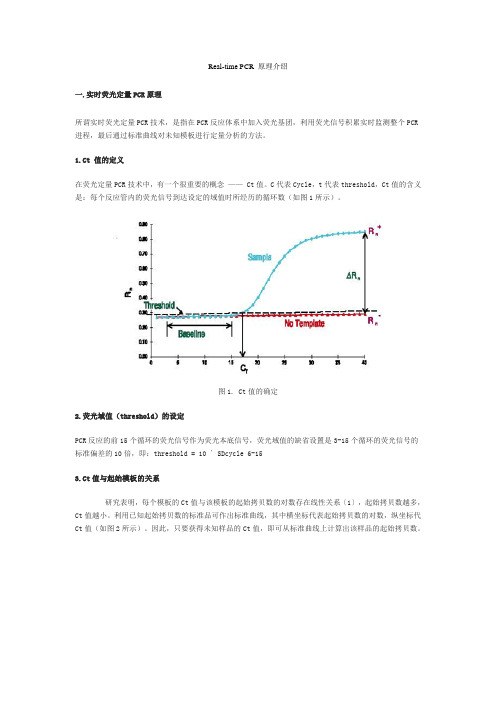
Real-time PCR 原理介绍一.实时荧光定量PCR原理所谓实时荧光定量PCR技术,是指在PCR反应体系中加入荧光基团,利用荧光信号积累实时监测整个PCR 进程,最后通过标准曲线对未知模板进行定量分析的方法。
1.Ct 值的定义在荧光定量PCR技术中,有一个很重要的概念—— Ct值。
C代表Cycle,t代表threshold,Ct值的含义是:每个反应管内的荧光信号到达设定的域值时所经历的循环数(如图1所示)。
图1. Ct值的确定2.荧光域值(threshold)的设定PCR反应的前15个循环的荧光信号作为荧光本底信号,荧光域值的缺省设置是3-15个循环的荧光信号的标准偏差的10倍,即:threshold = 10 ´ SDcycle 6-153.Ct值与起始模板的关系研究表明,每个模板的Ct值与该模板的起始拷贝数的对数存在线性关系〔1〕,起始拷贝数越多,Ct 值越小。
利用已知起始拷贝数的标准品可作出标准曲线,其中横坐标代表起始拷贝数的对数,纵坐标代Ct 值(如图2所示)。
因此,只要获得未知样品的Ct值,即可从标准曲线上计算出该样品的起始拷贝数。
图2. 荧光定量标准曲线4.荧光化学荧光定量PCR所使用的荧光化学可分为两种:荧光探针和荧光染料〔2〕。
现将其原理简述如下:1)TaqMan荧光探针:PCR扩增时在加入一对引物的同时加入一个特异性的荧光探针,该探针为一寡核苷酸,两端分别标记一个报告荧光基团和一个淬灭荧光基团。
探针完整时,报告基团发射的荧光信号被淬灭基团吸收;PCR扩增时,Taq酶的5’-3’外切酶活性将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接收到荧光信号,即每扩增一条DNA链,就有一个荧光分子形成,实现了荧光信号的累积与PCR产物形成完全同步。
如图3所示。
图3. TaqMan 荧光探针工作原理2)SYBR荧光染料:在PCR反应体系中,加入过量SYBR荧光染料,SYBR荧光染料特异性地掺入DNA双链后,发射荧光信号,而不掺入链中的SYBR染料分子不会发射任何荧光信号,从而保证荧光信号的增加与PCR产物的增加完全同步。
RealTime_RT-PCR实验流程
RealTime RT-PCR实验流程1.样本Total RNA提取- Trizol法提取组织、细胞Total RNA(1)样本处理(细菌、细胞不用研磨);在铝箔中将1cm2大小左右的组织样本敲碎,移至已加入钢珠的Eppendorf管中,使用研磨仪(30 1/s,8min)进行研磨;注:此步操作应尽可能在低温液氮环境下操作,细菌、细胞样本不用研磨。
(2)在研磨后的Eppendorf管中加入1ml Trizol,振荡混匀;(3)将混匀后溶液移至一新Eppendorf管中,加入200μl氯仿,振荡混匀;(4)离心:4℃,12000rpm,15min;(5)将离心后上清液移至一新Eppendorf管中,加入500μl异丙醇,轻轻混匀,室温静置15min;(6)离心:4℃,12000rpm,15min;(7)倒掉上清液,加入1ml 75%乙醇,振荡混匀;(8)离心:4℃,7500rpm,5min;(9)倒掉上清液,并在超净台中将乙醇挥发干净;(10)加入40~60μl DEPC H2O,65℃ 5min助溶;(11)-20℃冷冻保存。
2.Total RNA质量检测(1)NanoDrop测定Total RNA浓度(2μl Total RNA上样)(2)1.5%甲醛变性琼脂糖凝胶电泳检测RNA质量Total RNA 500ng5×Loading Buffer 2μlDEPC H2O to 7~8μl65℃变性5min,冰浴5minEB(500倍稀释)1μl总体积约为8~9μl甲醛变性琼脂糖凝胶:30ml 1×TBE Buffer中加入0.45g琼脂糖,微波炉中加热溶化,轻轻摇动使琼脂糖充分混匀(肉眼观察无颗粒状悬浮物),冷却至60℃左右时加入600μl甲醛,混匀,倒入RNA专用的制胶器中(7.5×5.5cm),室温静置30min左右即可使用。
电泳条件:120~130V,15~20min。
real-time pcr名词解释
real-time pcr名词解释
外文名【Real-time Quantitative polymerase chain reaction】
中文名【实时荧光定量多聚核苷酸链式反应】
中文简称【实时荧光定量PCR】
外文简称【RT PCR】
【实时荧光定量PCR技术】,是指在PCR反应体系中加入荧光基团,利用荧光信号积累实时监测整个PCR进程,最后通过标准曲线对未知模板进行定量分析的方法。
实时荧光定量PCR (Quantitative Real-time PCR)是一种在DNA扩增反应中,以荧光化学物质测每次聚合酶链式反应(PCR)循环后产物总量的方法。
通过内参或者外参法对待测样品中的特定DNA序列进行定量分析的方法。
·
Real-timePCR是在PCR扩增过程中,通过荧光信号,对PCR进程进行实时检测。
由于在PCR扩增的指数时期,模板的Ct值和该模板的起始拷贝数存在线性关系,所以成为定量的依据。
【技术原理】
将标记有荧光素的Taqman探针与模板DNA混合后,完成高温变性,低温复性,适温延伸的热循环,并遵守聚合酶链反应规律,与模板DNA互补配对的Taqman 探针被切断,荧光素游离于反应体系中,在特定光激发下发出荧光,随着循环次数的增加,被扩增的目的基因片段呈指数规律增长,通过实时检测与之对应的随扩增而变化荧光信号强度,求得Ct值,同时利用数个已知模板浓度的标准品作对照,即可得出待测标本目的基因的拷贝数。
realtimepcr简易操作流程
Real-time PCR(实时聚合酶链反应)是一种用于检测和定量DNA或RNA的技术。
它可以在PCR(聚合酶链反应)进行的实时监测PCR 产物的累积量。
real-time PCR技术适用于病毒检测、基因表达分析、SNP检测等许多领域。
real-time PCR技术的操作流程相对简单,但需要一定的实验操作技巧和严格的操作规范。
以下是real-time PCR的简易操作流程:1. 样品准备- 从实验对象中收集所需的组织样品或细胞样品。
- 样本的处理和提取要遵循标准的生物安全操作规程,确保样品的纯度和完整性。
2. DNA/RNA提取- 根据实验需求选择合适的DNA/RNA提取试剂盒和方法,从样品中提取目标DNA或RNA。
- 保证提取的DNA/RNA质量和浓度满足real-time PCR的要求。
3. Primer和探针设计- 根据目标基因序列设计引物和探针,确保其特异性和有效性。
- 引物和探针的设计应遵循一定的原则和标准,可以使用专业的设计软件或委托专业实验室进行设计。
4. PCR体系设置- 根据实验设计和引物/探针的特性设置PCR反应体系,包括引物和探针的最佳浓度、PCR反应缓冲液、酶和模板DNA/RNA的加入量等。
- 严格按照实验方案中的要求和比例进行PCR反应体系的设置,确保反应的准确性和稳定性。
5. PCR反应- 将PCR反应体系加入到real-time PCR仪器中,根据实验方案设置好PCR程序。
- 在PCR过程中,实时监测PCR产物的累积量,记录和分析反应曲线。
6. 数据分析- 根据实验目的和方法选择合适的real-time PCR数据分析软件,对实验数据进行处理和分析。
- 分析数据并计算目标基因的相对表达量或浓度,进行统计学分析和结果解读。
7. 结果呈现- 将实验结果以图表或表格的形式清晰呈现出来,配以必要的文字解释。
- 结果的呈现应简洁明了,突出实验的关键发现和结论。
real-time PCR技术的操作流程虽然相对简单,但在实际操作中仍然需要严格遵循操作规范和注意实验细节。
Real-timePCR实验原理与技术
实时荧光定量PCR技术原理
01
在实时荧光定量PCR反应中, DNA模板被扩增时,与荧光探 针结合,产生荧光信号。
02
随着DNA的扩增,荧光信号逐 渐增强,通过对荧光信号的实 时监测和分析,可以计算出 DNA的起始浓度。
03
通过标准曲线或内参基因的校 准,可以将起始浓度转化为目 标基因的表达量或基因拷贝数 。
实时监测荧光信号
实时监测荧光信号,确保PCR产物在指数扩增阶段被检测到。
标准化实验流程
建立标准化实验流程,确保实验结果的可靠性和可重复性。
未来展望
新技术应用
01
随着新技术的不断发展,如数字PCR、微流控PCR等,Real-
time PCR技术将得到进一步优化。
高通量检测
02
通过多重PCR和高通量检测平台,实现大规模样本的快速检测。
03
Real-time PCR 实验数 据分析
数据收集与整理
数据收集
在实时PCR实验中,数据收集通常涉 及记录每个循环的荧光信号强度。这 些数据通常以图表形式表示,其中横 轴表示循环数,纵轴表示荧光信号强 度。
数据整理
收集到的原始数据需要经过整理,包 括去噪、去除异常值和标准化等步骤, 以确保数据分析的准确性。
自动化与智能化
03
实现Real-time PCR的自动化和智能化,提高实验效率,减少人
为误差。
THANKS FOR WATCHING
感谢您的观看
real-timepcr实验原 理与技术
contents
目录
• Real-time PCR 实验原理 • Real-time PCR 实验技术 • Real-time PCR 实验数据分析 • Real-time PCR 实验应用 • Real-time PCR 实验注意事项与优化建
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
总RNA的提取
1)匀浆处理:
a. 组织将组织在液氮中磨碎,每50~100mg组织加入1mlTRIzol,用匀浆仪进行匀浆处理。
样品体积不应超过TRIzol体积10℅。
b. 单层培养细胞直接在培养板中加入TRIzol裂解细胞,每10cm2面积(即3.5cm 直径的培养板)加1ml,用移液器吸打几次。
TRIzol的用量应根据培养板面积而定,不取决于细胞数。
TRIzol加量不足可能导致提取的RNA有DNA污染。
c. 细胞悬液离心收集细胞,每5~10×106动物、植物、酵母细胞或1×107细菌细胞加入1mlTRIzol,反复吸打。
加TRIzol之前不要洗涤细胞以免mRNA降解。
一些酵母和细菌细胞需用匀浆仪处理。
2)每使用1mlTRIzol加入0.2 ml氯仿,剧烈震荡20 sec,室温静置10 min;
3)4℃12000 rpm 15 min,分三相,RNA在上层水相中;
4)小心吸取上清于新的EP管中,加入等体积的异丙醇,颠倒混匀,室温静置10-20 min;
5)4℃12,000×g 15 min离心;
6)小心弃上清,加1 ml 75%冷乙醇(DEPC水配置)洗涤沉淀,上下颠倒使沉淀重悬;
7)4℃7500×g 5 min 离心;
8)小心弃上清并尽量将液体吸干,沉淀在室温晾干;
9)加入30-50 μl RNase-free的水溶解,轻弹或短暂离心,50℃5 min促溶;取1 μl RNA稀释后测浓度并进行琼脂糖凝胶电泳分析RNA质量。
消化RNA中的DNA
参照RNase-free DNase (Promega)说明书步骤。
1)10 μl体系加入1 μg RNA、1 μl RQ1 RNase-free DNase、1 μl RQ1 DNase buffer,因RNA的量会有所损失,选择处理15 μg RNA,DNase及buffer均加15 μl,H2O补齐至150 μl,混匀;
2)37℃处理30 min;
3)补水至200 μl,加入100 μl氯仿,振荡混匀,室温静置10 min;
4)4℃顶速离心10-15 min,小心吸取上层水相溶液于新的EP管中;
5)加入0.1倍体积的NaAc(3 M,pH5.2)及等体积的异丙醇,室温沉淀半小时;
6)4℃顶速离心15 min,去上清,沉淀用75% DEPC乙醇洗涤;
7)4℃离心5 min,尽量去干净上清,室温干燥3-5 min;
8)加入20 μl RNase-free H2O溶解,轻弹或短暂离心,50℃5 min促溶;
9)取1 μl RNA稀释后测浓度并进行琼脂糖凝胶电泳分析RNA质量。
RNA样品要求OD260/280为1.8~2.2,完整性良好;28S、18S条带清晰,且28S比18S亮或至少与18S相同,5S很弱;
附Total RNA 电泳图,电泳时RNA总量在200ng-800ng左右效果比较好。
反转录
参照SuperScript II reverse transcriptase (Invitrogen)说明书步骤。
1)取3 μg RNA,加入Oligo(dT)primer 0.5 μl 、dNTP 1 μl,补H2O至12 μl,混匀;
2)65℃5 min,冰上5 min;
3)加入5×Reaction Buffer 4 μl、0.1 M DTT 2 μl、RNase OUT 1 μl,混匀;
4)42℃2 min,加入SuperScript II reverse transcriptase 1 μl,混匀;
5)42℃50 min,72℃15 min,4℃待用。
定量PCR
参照TaKaRa SYBR Premix Ex Taq试剂及7500 real-time PCR instrument (Applied Biosystems) 说明书进行。
1)引物设计:SYBR Green I荧光嵌合法进行Real-time PCR反应对引物的特异性及效率要求很高,设计原则可参考Takara公司Real-time PCR实验及解析方法介绍,其关键点如下:
●Tm值和GC含量在适当范围内以保证引物与目的DNA片段的稳定
退火;
●引物内部及上下游引物之间无互补序列以保证引物利用效率;
●使用BLAST以保证引物的特异性,3’端特异尤为重要。
设计好的引物需提前进行预实验,扩定量PCR熔解曲线为单一且锐利的锋,产物进行琼脂糖凝胶电泳只有特异扩增条带而无引物二聚体和非特异带的引物则可用;
2)进行PCR反应:相对定量PCR目前用的较多,最好检测至少四个内参,内参一般选择表达水平高的管家基因(如ACTIN,TUBULIN,GAPDH,eEF-1等)。
PCR反应体系如下(20 μl):
SYBR Premix Ex Taq(2×)10 μl
Rox Reference Dye II(50×)0.4 μl
ddH2O 8.5 μl
cDNA template 0.3 μl
Primer mix(2.5 μM,终浓度100 nM)0.8 μl
可将前三种溶液混到一起,之后按模板分装,引物可直接加到PCR反应板上。
检测同一个基因的不同样品最好放在同一竖排,Real-time PCR检测非常灵敏,同样的模板及引物需重复扩3个孔,结果必须来自至少三次独立实验所得的数据;
3)选用ABI 7500 Real-time PCR仪进行扩增,程序如下:
Stage 1:95℃10 sec,
Stage 2:95℃3 sec,60℃30 sec,72℃34 sec(读板),循环40次;
熔解曲线程序为:95℃15 sec,60℃ 1 min,95℃15 sec;
4)结果分析:在PCR反应的对数增长期中间左右的位置设定荧光阈值(不同批次实验的同一个检测基因需设定一样的荧光阈值),读取并输出Ct值。
计算公式:
2 -△Ct WT=2 –(Ct WT目的基因-Ct WT内参基因)
2 -△Ct mutant=2 –(Ct mutant目的基因-Ct mutant内参基因)
2 - △△Ct =2 -△Ct mutant/2 -△Ct WT
先计算不同内参之间的相对水平,并选择一个在野生型和突变体之间稳定表达的内参。
通过计算2 - △△Ct检测目的基因在野生型和突变体中的差异:野生型为1.0,突变体所得数值即为目的基因在突变体中的表达水平是野生型表达水平的倍数;不同批次实验可用2 -△Ct WT计算野生型的标准差。
