全基因组选择育种值估计
基因组选择育种遗传评估模型

基因组选择育种遗传评估模型
基因组选择育种遗传评估模型是一种基于全基因组信息进行选择育种的模型,通过对个体的基因组信息进行精确测量和分析,预测其表型表现和育种价值。
该模型主要分为三个步骤:
1. 基因型数据准备:对个体的基因组进行测序或基因分型,获取个体的基因型数据。
2. 关联分析:通过关联分析方法,将基因型数据与表型数据进行关联,找出与表型性状相关的基因位点。
3. 预测育种值:利用与表型性状相关联的基因位点信息,构建预测模型,对个体的育种值进行预测。
基因组选择育种遗传评估模型具有以下优点:
1. 高精度:通过对全基因组信息的分析,可以更准确地预测个体的表型表现和育种价值。
2. 高效率:相较于传统的育种方法,基因组选择育种遗传评估模型可以大大缩短育种周期和成本。
3. 广泛应用:该模型适用于各种农作物和动物,具有广泛的应用前景。
总的来说,基因组选择育种遗传评估模型是一种基于全基因组信息进行精确育种的模型,具有高精度、高效率和广泛应用等优点。
未来随着基因组学技术的发展,该模型将得到更广泛的应用和推广。
育种与全基因组选择-19页PPT资料

利用表型数据和分子数据的组合,减少田间试验 数量,提高育种效率
利用MAS的潜在优势
Meuwissen & Goddard, 2019 (GSE)
三种不同类型的分子标记
直接的标记 功能性 的变异- 已知的基因
LD 标记
分子标记与数量性状位点的关联 存在于整个群体当中
LE 标记 分子标记与数量性状位点的关联 不存在于整个群体当中,而只存 在于家系当中
分子标记贡献ˆk 量的估计值
利用分子标记 贡献量的估计 值估计其他基 因型的表现型
GE BV ˆkgik
The infinitesimal model (Fischer 1918) vs. The finite loci model
统计方法
Ridge Regression
ˆX 'XI 1X 'y
+50
550
500
GQ GQ Aq Aq Aq
目标:
找到主要基因或者 和主要基因联系紧 密的分子标记
用分子标记辅助选 择育种 MAS
Marker-Assisted Selection
分子辅助选择策略
数量遗传性状黑箱
基因
数量遗传性状灰箱
重要基因 分子标记 数量位点QTL
表现型数据 分子数据
分子辅助选择
杂交1
杂交2
杂交3
杂交4
MAS应用受限制的原因
分子标记的数量过少 分子标记只能解释非常少的遗传方差分量 基因分型的成本过高 分子标记/数量性状位点(QTL)的贡献量估计值
不稳定
贡献量大的位点倾向于被高估 分子标记贡献量之在家系和控制试验中有意义 分子标记/QTL的贡献量与遗传背景 和(或者) 环境的
科技成果——利用基因组检测评估奶牛育种值构建高质量奶牛群技术
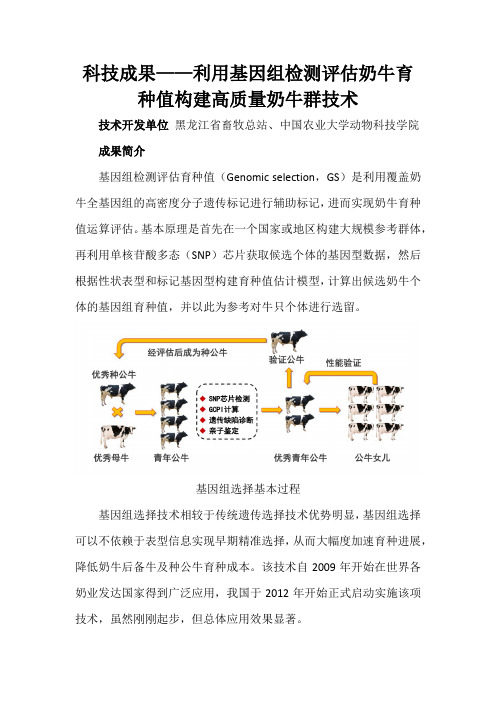
科技成果——利用基因组检测评估奶牛育种值构建高质量奶牛群技术技术开发单位黑龙江省畜牧总站、中国农业大学动物科技学院成果简介基因组检测评估育种值(Genomic selection,GS)是利用覆盖奶牛全基因组的高密度分子遗传标记进行辅助标记,进而实现奶牛育种值运算评估。
基本原理是首先在一个国家或地区构建大规模参考群体,再利用单核苷酸多态(SNP)芯片获取候选个体的基因型数据,然后根据性状表型和标记基因型构建育种值估计模型,计算出候选奶牛个体的基因组育种值,并以此为参考对牛只个体进行选留。
基因组选择基本过程基因组选择技术相较于传统遗传选择技术优势明显,基因组选择可以不依赖于表型信息实现早期精准选择,从而大幅度加速育种进展,降低奶牛后备牛及种公牛育种成本。
该技术自2009年开始在世界各奶业发达国家得到广泛应用,我国于2012年开始正式启动实施该项技术,虽然刚刚起步,但总体应用效果显著。
基因组选择基本原理应用情况目前基因组选择技术已经得到广泛应用,奶业发达国家的青年公牛选择已经100%应用基因组选择技术,截止到2019年美国和加拿大基因组参考群体已达到448007头荷斯坦牛(其中公牛群体56970头)。
荷兰、德国和北欧三国也相继成立了基因组选择技术平台,参考群体规模也在不断扩大。
我国奶牛基因组选择技术平台是从2008年开始研发,中国农业大学奶牛分子育种团队于2012年申请《中国荷斯坦牛基因组选择分子育种技术体系的建立与应用》项目,已通过教育部科技成果鉴定,开始在全国推广应用,实现了青年公牛基因组选择全覆盖。
截至2020年12月,中国荷斯坦牛基因组选择参考群体累计3497头,荷斯坦青年公牛全部完成了基因组检测和评估。
技术效果2009年我国奶牛育种开始应用基因组选择技术,该技术显著提高了奶牛遗传世代进展,降低种公牛及母牛核心群培育成本。
参测牛群主要经济性状的遗传进展得到加速提升,产奶量的平均遗传进展提升58.1kg/年;乳脂量的平均遗传进展提升1.42kg/年;乳蛋白量的平均遗传进展提升 1.76kg/年,在产奶性能遗传进展方面增加直接效益4.6亿/年。
估计育种值的方法概述

国际瞭望GLOBAL NEWS海外文摘对公猪肉质异味质量控制的研究最近,德国哥廷根大学的Johanna Trautmann博士对公猪肉质异味的感官质量控制进行了深入研究,其研究结果总结如下。
需要谨慎选择公猪异味评估者选择程序的第一部分是进行气味测试,主要目的是客观地描述公猪肉质异味评估员的遴选过程。
该测试主要是分析雄烯酮和粪臭素的检测阈值以及评估员通过易于使用的纸质气味条区分和鉴定各种浓度水平的物质的能力。
随后,对25个脂肪样品进行异味检测,以评估嗅觉性能对肉质感官质量控制的影响。
评估员对雄烯酮和粪臭素的嗅觉敏锐度显示出相当大的个体间变异性。
由此可知,评估员的嗅觉性能显著影响脂肪样品评级为公猪肉质异味的概率。
热铁是公猪异味检测的最佳选择第二部分的主要目的是填补先前研究中用于公猪肉质异味评价的样品制备差异。
在本次研究中,通过3个常用的工具加热脂肪样品以检测公猪肉质异味,即微波、热铁和热水法。
审核小组由10个评估员组成,对72个脂肪样品进行比较。
加热方法显著影响偏差评级的概率。
与假定的“金标准”(化学分析)相比,当考虑灵敏度和特异性时,通过热铁处理的肉品质量更好一些。
此外,结果显示,与单个评估者的评估结果相比,审核组的评估结果更准确。
恒定噪声不影响人们嗅觉第三部分的主要目的是质疑广泛的建议,即感官测试应该在没有外来噪声的环境中进行。
然而,当在屠宰环境中进行评估时,大家对评估结果有所质疑。
在本研究中,选择了两组成员:通常在无声条件下工作的大学小组和通常在屠宰场工作的屠宰场小组。
我们针对40个公猪肉质样品研究了气味辨别、气味识别和气味检测阈值。
结果表明,噪声不影响评估员对肉品有无异味的结果判断。
雄甾烯酮和粪臭素相互作用影响肉品的感官判断第四部分的主要目的是深入分析雄烯酮和粪臭素的气味及气味相互作用。
因此,采用气相色谱质谱法对1 043只公猪的脂肪样品进行感官评价和公猪肉质异味化合物的定量测定。
每个样品由10个训练有素的评估者评价,得到11 000个以上的个体评分,对其进行统计分析。
全基因组选择育种(GS)简介

全基因组选择育种(GS)简介全基因组选择(Genomic selection, GS)是⼀种利⽤覆盖全基因组的⾼密度标记进⾏选择育种的新⽅法,可通过早期选择缩短世代间隔,提⾼育种值(Genomic Estimated Breeding Value, GEBV)估计准确性等加快遗传进展,尤其对低遗传⼒、难测定的复杂性状具有较好的预测效果,真正实现了基因组技术指导育种实践。
原理常规育种⼿段主要利⽤性状记录值、基于系谱计算的个体间亲缘关系,通过最佳线性⽆偏估计(best linear unbiased predication,BLUP)来估计各性状个体育种值(EBVs),通过加权获得个体综合选择指数,根据综合选择指数⾼低进⾏选留。
标记辅助选择(marker assisted selection, MAS)育种,利⽤遗传标记,将部分功能验证的候选标记联合BLUP计算育种值,这样不仅可以提⾼育种值估计的准确性,⽽且可以在能够获得DNA时进⾏早期选择,缩短世代间隔,加快遗传进展。
⽽GS则通过覆盖全基因组范围内的⾼密度标记进⾏育种值估计,继⽽进⾏排序、选择,简单可以理解为全基因组范围内的标记辅助选择,主要⽅法是通过全基因组中⼤量的遗传标记估计出不同染⾊体⽚段或单个标记效应值,然后将个体全基因组范围内⽚段或标记效应值累加,获得基因组估计育种值(GEBV),其理论假设是在分布于全基因组的⾼密度SNP标记中,⾄少有⼀个SNP能够与影响该⽬标性状的数量遗传位点(quantitative trait loci, QTL)处于连锁不平衡(linkage disequilibrium, LD)状态,这样使得每个QTL的效应都可以通过SNP得到反映。
相⽐BLUP⽅法,全基因组选择可以有效降低计算个体亲缘关系时孟德尔抽样误差的影响;相⽐MAS⽅法,全基因组选择模型中包括了覆盖于全基因组的标记,能更好地解释表型变异。
技术路线植物GS路线动物GS路线GS预期效果:1. 缩短育种周期,实现待选群体的低世代选留2. 提⾼育种值估计准确性3. 降低育种成本,减少表型鉴定的数量4. 预测亲本杂交后代,选择最佳杂交优势组合统计模型统计模型是GS的核⼼,极⼤地影响了基因组预测的准确度和效率。
猪育种新技术

C
B
A
全基因组选择
育种目标的变化
测定技术的变化
育种新技术
全基因组选择
什么叫全基因组选择为了进一步提高育种的效率,T.H,Meuwissen等提 出 了 一 种新的标记辅助选择方法,即全基因组选择(Genomic selection,GS)。该方法假设覆盖全基因组上的高密度SNPs标记中至少有1个SNP与 QTL处于连锁不平衡关系,利用SNP估计每个QTL的效应,从而获得个 体 的 全 基 因 组 估 计 育 种 值 (Genomic estimated breeding value, GEBV)。
对猪体进行一系列扫描之后,可以精确预测胴体的组成成分。与传统的超声波技术相比,用这种技术预测胴体肌肉、脂肪和骨骼产量的准确率分别可提高6.4%、5.6%和15.0%。
测定技术的变化
对于屠宰商和零售商来说,CT还有一个更加突出的优点,那就是可以测出不同大分切部位(背脊部、后腿部、腹胁部和肩胛部)当中的肌肉产量。
01
02
03
全基因组选择的优点不仅可以提高选择的准确性,尤其是一些低遗传力性状、难以测量的性状、限性性状、生长后期测定的性状、屠宰性状和免疫力等,还可以在动物出生时或者胚胎期即可预测 GEBV,从而缩短世代间隔,大大提高遗传进展。
全基因组选择商业化应用在猪的全基因组测序完成后,A.M.Ramos等对商业品种杜洛克(34头)、皮特兰(23头)、长白猪(29头)、大白猪(36头)和野猪(36头)共5个品种的DNA分别混池,采用全基因组重测序方法鉴定出数十万个SNPs,并从中选择设计出PorcineSNP60 Beadchip。该SNP芯片共有64232个SNPs,检出率可达97.5%,SNP信息可靠。
全基因组选择及其在玉米育种中的研究进展

报告从20世纪80年代开始,分子标记系统的开发使植物育种者和分子生物学家获得多态性标记的数量大大增加。
单核苷酸多态性(Single nucleotide polymorphisms,SNPs)已经在数量性状基因座(Quantitative trait locus,QTL)中广泛使用。
目前已有多项研究结果表明,超过10 000个不同标记系统的QTL 应用于12种植物中,旨在改善具有重要经济价值的数量性状。
最初,通过应用MAS 将分子标记整合到传统的表型选择(Phenotypic selection,PS)中。
对于简单的性状,MAS 只选择具有主要作用的QTL 相关标记的个体,不使用与性状无显著相关的标记的个体。
由于QTL 与环境相互作用,难以在多种环境中或不同的遗传背景下找到相同的QTL,通过使用QTL 相关标记检测来改善多基因控制的复杂数量性状是不可行的,因此,新的MAS 技术-基因组选择(GS)应运而生。
Meuwissen 等首次提出了GS 育种策略,GS 育种分为两步。
第一步主要是利用训练群体的基因分型结果和表型建立最佳线性无偏预测(Best linear unbiased prediction,BLUP )模型,得到训练的育种值(Breeding value,BV)。
第二组是育种群体的基因型数据,但群体中的个体均没有表型,基于BLUP 模型和与训练群体中的表型相关的基因组的等位基因同一性来预测育种群体的各种性状的表现,从而得到GEBV ,GEBV 来源于预测群体中每个个体的基因组中发生的有用基因组的组合,并且提供了每个个体具有优良表型的估计值,即高育种值。
可以根据GEBV 选择新的育种亲本。
GS 与传统的MAS 相比有以下优点:①GS 不需要QTL 定位。
GS 不同于连锁和关联作图的策略,它不是映射单个基因效应,而是基于大量分子标记对有效育种值进行估计,理想地覆盖全基因组。
②GS 更精确,特别是对于早期选择。
育种与全基因组选择

G
Q
550
A q
A
q
500
A q
分子辅助选择策略
数量遗传性状黑箱
表现型数据 基因
分子辅助选择 数量遗传性状灰箱
重要基因 分子标记 数量位点QTL 数量位点
分子数据
利用表型数据和分子数据的组合,减少田间试验 数量,提高育种效率
利用MAS的潜在优势 利用MAS的潜在优势
Meuwissen & Goddard, 1996 (GSE)
用贡献量显著的 分子标记进行MAS
可以找到更多的 LD 分子标记 但是所有的显著的分子标记也 只能解释很少遗传方差。大量 的微小贡献率被忽略
解决方案:全基因组选择 解决方案:
利用全部的SNPs 利用全部的SNPs 同时估计50000个SNPs的贡献量 同时估计50000个SNPs的贡献量 解大型线形方程组
贡献量大的位点倾向于被高估 分子标记贡献量之在家系和控制试验中有意义 分子标记/QTL的贡献量与遗传背景 分子标记/QTL的贡献量与遗传背景 和(或者) 环境的 交互作用显著 分子标记与QTL的 分子标记与QTL的Linkage Disequilibrium 只存在于独立 群体
2000年开始的分子技术革命 2000年开始的分子技术革命
选择 数量遗传性状黑箱 h2
基因
表现型
环境
现代的选择策略
选择 数量遗传性状黑箱 h2
基因
表现型
育种值估计
环境
具有亲缘关系 的基因型的表 型数据
BLUP: Best Linear Unbiased Prediction
美国玉米育种的成功
数据来源 Richard Johnson, Illinois Corn Breeders School, March 1-2, 12010 1964 Yield/acre Product price Seed corn price 80 bu $1.10/bu $10/Unit 2010 180 bu $3.85/bu $350/Unit
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
全基因组选择育种值估计是一种利用覆盖全基因组的高密度分子标记进行选择育种的方法。
其原理是通过构建预测模型,根据基因组估计育种值(Genomic Estimated Breeding Value,GEBV)进行早期个体的预测和选择,从而缩短世代间隔,加快育种进程,节约大量成本。
统计模型是全基因组选择的核心,影响着全基因组预测的准确度和效率。
传统预测方法基于线性回归模型,但难以捕捉基因型和表型间的复杂关系。
相较于传统模型,非线性模型(如深度网络神经)具备分析复杂非加性效应的能力,人工智能和深度学习算法为解决大数据分析和高性能并行运算等难题提供了新的契机,深度学习算法的优化将会提高全基因组选择的预测能力。
全基因组选择已应用于奶牛、生猪的品系选育中,但在家禽育种方面的研究和应用相对较少。
随着分子标记检测技术不断发展,分子育种进入了全基因组选择时代,这将推动现代育种向精准化和高效化方向发展。
