常用核酸探针标记方法
常用核酸探针标记方法
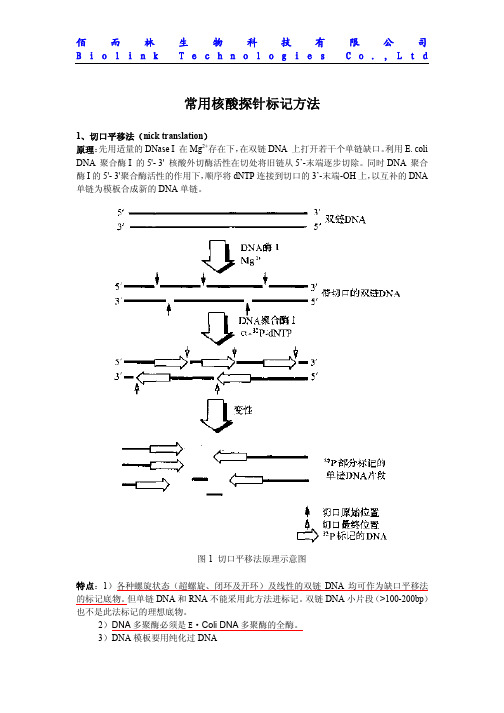
常用核酸探针标记方法1、切口平移法(nick translation)原理:先用适量的DNase I 在Mg2+存在下,在双链DNA 上打开若干个单链缺口。
利用E. coli DNA 聚合酶I 的5'- 3' 核酸外切酶活性在切处将旧链从5’-末端逐步切除。
同时DNA 聚合酶I的5'- 3'聚合酶活性的作用下,顺序将dNTP连接到切口的3’-末端-OH上,以互补的DNA 单链为模板合成新的DNA单链。
图1 切口平移法原理示意图特点:1)各种螺旋状态(超螺旋、闭环及开环)及线性的双链DNA均可作为缺口平移法的标记底物。
但单链DNA和RNA不能采用此方法进标记。
双链DNA小片段(>100-200bp)也不是此法标记的理想底物。
2)DNA多聚酶必须是E·Coli DNA多聚酶的全酶。
3)DNA模板要用纯化过DNA2、随机引物法(random priming)原理:将待标记的DNA探针片段变性后与随机引物一起杂交,然后以此杂交的寡核苷酸为引物,大肠杆菌DNA聚合酶I大片段(E·Coli DNA polymerase I Klenow Fragment)的催化下,合成与探针DNA互补的DNA链。
当反应液中含有标记的dNTP时,即形成标记的探针。
图2 随机引物标记法原理示意图特点:1)除了能进行双链DNA标记外,也可用于单链DNA和RNA探针的标记。
2)所得到的标记产物是新合成的DNA单链,而所加入的DNA片段本身并不能被标记。
3)新形成的标记DNA单链的长度与加入寡核苷酸引物的量成反比,因为加入的寡核苷酸数量越多,合成起点也越多,得到的片段的长度也越短。
按标准方法得到的标记产物长度一般为200-400bp。
3、末端标记与切口平移法和随机引物法不同,DNA末端标记法并不将DNA片段的全长进行标记,而是只将其一端(5’或3’端)进行部分标记。
其特点是可得到全长DNA片段,DNA片段并非均匀标记,标记活性不高。
[说明]核酸分子杂交及PCR技术
![[说明]核酸分子杂交及PCR技术](https://img.taocdn.com/s3/m/cd08778f50e79b89680203d8ce2f0066f53364c4.png)
核酸的分子杂交技术一、核酸分子杂交用标记的已知DNA或RNA片段(探针)来检测样品中未知核酸序列,通过核苷酸间碱基互补的原则发生异源性结合,再经显影或显色的方法,将结合核酸序列的位置或大小显示出来。
待测的核酸序列,可以是克隆的基因片段,也可以是未克隆化的基因组DNA和组织细胞的RNA。
二、核酸分子杂交的分类液相杂交核算分子杂交印记杂交固相杂交原位杂交1.固相杂交:将需要杂交的一条核酸链先固定在固体支持物上,另一条核酸链游离在液体中。
2.液相杂交:参与反应的两条核酸链都游离在液体中。
常用固相杂交类型:Southern印迹杂交、Northern印迹杂、菌落原位杂交、斑点杂交、狭缝杂交、组织原位杂交、夹心杂交等。
三、核酸分子杂交的基本原理1、变性:在某些理化因素的作用下,核酸双链分子碱基对的氢键断裂,疏水作用被破坏,双链螺旋或发夹结构被拆开,有规则的空间结构被破坏,形成单链分子,称为核酸的变性。
﹡引起核酸变性的因素:热、酸、碱、化学试剂(如:尿素、甲酰胺、甲醛等)。
﹡加热变性是最常用的方法,一般加热80-100℃数分钟即可使核酸分子氢键断裂,双链分开。
﹡变性的核酸分子失去了生物活性,同时理化性质也随之改变,其紫外吸收值(A260)也随之升高。
可用紫外吸收的变化来跟踪DNA的变性过程。
以A260吸收值对应温度作图,得到DNA的变性曲线或熔解曲线。
增色效应:DNA变性后对260nm紫外光收增加的现象。
DNA热变性现象双螺旋结构即发生解体,两条链分开形成无规则线团。
同时,一系列物化性质发生改变:260nm处紫外吸收值升高,粘度降低,浮力密度升高。
由于二级结构的丧失,也失去了部分或全部生物活性。
增色效应和减色效应当DNA分子加热变性后,其260nm的紫外吸收会急剧增加的现象称为增色效应。
变性DNA复性后,在260nm处的吸收值减少的现象称为减色效应。
A260值达到最大值1/2时的温度称为解链温度或熔解温度(melting temperature,Tm),此时50%的DNA分子发生了变性。
Fish探针标记方法大全

探针标记方法大全1、DNA的缺口平移法缺口平移是一种快速、简便、成本相对较低以及生产高比活性均一标记DNA的方法。
该技术可以制备序列特异的探针。
当使用重组质粒探针时,虽然任何形式的双链DNA都可以进行缺口平移标记。
但通常使用限制性核酸内切酶将插入片段进行酶切和凝胶电泳纯化后再进行缺口平移标记。
此外,用这种探针进行杂交可产生较低的背景信号。
2、DNA的随机引物法这种方法是使用寡核苷酸引物和大肠杆菌DNA聚合酶Ⅰ的Klenow片段来标记DNA片段。
这种方法可代替缺口平移产生均一的标记探针外,还在许多方面优于缺口平移法,比如标记核苷酸在DNA探针中的掺入率可达50%以上。
由于在反应过程中加入的DNA片段不被降解,故在随机引物反应中加入的DNA可少于200 ng,甚至10 ng也能有效地标记。
DNA片段的大小不影响标记的结果。
标记物沿所加入的全长DNA被均等地掺入。
标记的探针可以直接使用而不需要去除未掺入的核苷酸;单链双链DNA都可作为随机引物标记的模版。
随机引物可用于较小的DNA片段(100~500 bp),而缺口平移法对大DNA片段(>1000 bp)效果最好。
随机引物法的主要缺点是产生的标记探针量比缺口平移的要少些,此外环状DNA不能有效地标记,必须先用限制性内切核酸酶线形化或用碱法或DNA酶Ⅰ产生缺口。
3、RNA的体外转录法体外转录法标记RNA探针可用于商品化转录质粒来制备。
这些质粒中应该包括SP6、T3、或T7 RNA聚合酶的RNA启动位点和,而这些启动位点则与多克隆位点(multiple cloning sites,MCS)相邻。
4、RNA的寡脱氧核苷酸法克隆质粒pSP64、pGEM-3、pGEM-4等含邻近MCS的SP6RNA启动子,可作为从DNA寡核苷酸模版产生RNA探针的基础。
这两种标记方法产生探针量大,而且产生的探针不受载体序列的影响,所需要的线性化的质粒DNA大约1 ug,但需要高纯度的模板。
核酸探针的原理及应用

核酸探针的原理及应用1. 导言核酸探针是一种用于检测和鉴定核酸分子的分子探针。
它通过特异性识别目标核酸序列,可广泛应用于基因组学、生物医学研究、临床诊断等领域。
本文将介绍核酸探针的原理和应用。
2. 核酸探针的原理2.1 核酸杂交原理核酸探针的原理基于核酸的互补配对特性。
当目标核酸序列与探针的互补序列相遇时,它们之间会发生杂交反应。
杂交后,探针会与目标核酸形成稳定的双链结构,从而实现对目标核酸的特异性识别和检测。
2.2 核酸标记技术核酸探针通常需要标记以便于检测。
常用的核酸标记技术包括荧光标记、放射性标记和酶标记等。
这些标记可以通过特定的检测方法,如荧光显微镜、放射性测量和酶反应等,来检测探针与目标核酸之间的杂交情况。
2.3 核酸探针的设计核酸探针的设计需要考虑多个因素,包括目标序列的长度、杂交条件、标记方式等。
探针的长度应足够长以确保与目标序列的特异性结合,同时要避免与非目标序列的杂交。
此外,探针和目标序列的杂交温度、盐浓度等条件也需要进行优化。
3. 核酸探针的应用3.1 基因组学研究核酸探针在基因组学研究中扮演着重要角色。
通过使用特异性的核酸探针,可以对基因组进行定位、定序和变异等研究。
同时,核酸探针也可用于基因表达分析和基因功能研究,例如通过反转录聚合酶链式反应(RT-PCR)技术检测目标基因的表达水平。
3.2 生物医学研究核酸探针可用于生物医学研究中的疾病诊断和治疗。
例如,在肿瘤学研究中,核酸探针可以用于检测肿瘤相关基因的异常改变,从而实现早期诊断和治疗监测。
此外,核酸探针还可用于检测病毒和细菌感染,诊断遗传性疾病等。
3.3 临床诊断核酸探针在临床诊断中有着广泛应用。
通过对特定的核酸序列进行检测,可以实现对疾病的早期筛查和诊断。
常见的应用包括艾滋病病毒检测、乙肝病毒检测、人类乳头瘤病毒(HPV)检测等。
核酸探针的应用具有高度特异性和灵敏性,可以提供准确的诊断结果。
4. 总结核酸探针作为一种重要的生物技术工具,具有广泛的应用前景。
简述核酸分子探针的分类和标记方法

简述核酸分子探针的分类和标记方法下载提示:该文档是本店铺精心编制而成的,希望大家下载后,能够帮助大家解决实际问题。
文档下载后可定制修改,请根据实际需要进行调整和使用,谢谢!本店铺为大家提供各种类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by this editor. I hope that after you download it, it can help you solve practical problems. The document can be customized and modified after downloading, please adjust and use it according to actual needs, thank you! In addition, this shop provides you with various types of practical materials, such as educational essays, diary appreciation, sentence excerpts, ancient poems, classic articles, topic composition, work summary, word parsing, copy excerpts, other materials and so on, want to know different data formats and writing methods, please pay attention!核酸分子探针是一种用于检测和识别特定核酸序列的工具,它在生物学研究、医学诊断和药物开发中具有重要作用。
原位杂交-经典方法-地高辛标记探针

原位杂交(In situ hybridization)一、目的掌握核酸探针原位杂交操作技术,并利用该技术对单细胞的靶目标进行定位,用于细胞生物学基础研究。
二、原理原位杂交技术(in situ hybridization)是分子生物学和组织化学成功结合的产物,是特定标记的已知序列核酸作为探针与细胞或组织切片中核酸进行杂交并对其实行检测的方法。
其基本原理是含互补序列的标记DNA或RNA片段,即探针,在适宜的条件下与细胞内特定的DNA或RNA形成稳定的杂交体。
原位杂交能在成分复杂的组织中进行单一细胞的研究而不受同一组织中其它成分的影响,因此对于那些细胞数量少且散在于其他组织中的细胞内DNA或RNA研究更为方便;由于原位杂交不需要从组织中提取核酸,对于组织中含量极低的靶序列有极高的敏感性,并可完整地保持组织与细胞的形态,更能准确地反映出组织细胞的相互关系及功能状态。
三、仪器设备烘箱,切片机,展片机,染色缸,湿盒,原位PCR仪,显微镜、镊子、量筒、烧杯、吸水纸、枪与枪头、载玻片、盖玻片、冰盒等。
四、材料和试剂1.材料:带有病原体的水生动物,如感染WSSV病毒的对虾、感染虹彩病毒的水生动物等。
2.试剂:Davidson’s AFA固定液:330 ml 95%乙醇220 ml 福尔马林(37~39%甲醛水溶液)115 ml 冰醋酸335 ml H2O混匀后封口,室温放置;DIG标记与检测试剂盒(Roche公司);TNE:50 mmol/L Tris-HCl 6.57 g Tris Base10 mmol/L NaCl 0.58 g NaCl1 mmol/L EDTA 0.37 g EDTAddH2O 900 ml (定容至1L)用HCl调pH至7.4,高压灭菌,4℃保存;0.4% 甲醛:37~39%% 甲醛 5.4 mlddH2O 495 ml;蛋白酶K溶液(Pr.K,10 mg/ml):10 mg Pr.K 溶于1 ml TNE中(用时现配);20×SSC:3 mol/L NaCl 175.32 g NaCl0.3 mol/L柠檬酸钠88.23 g 柠檬酸钠ddH2O 900 ml (定容至1L)调pH至7.0,高压灭菌,4℃保存;2×SSC:20×SSC 100 mlddH2O 900 ml0.45 µm 滤膜过滤,4℃保存;20×Denhardt’s溶液:0.4%牛清血蛋白0.4 g牛清血蛋白0.4%聚蔗糖0.4 g聚蔗糖0.4%聚乙烯吡咯烷酮0.4 g聚乙烯吡咯烷酮ddH2O 100 ml0.45 µm 滤膜过滤,4℃保存;25%硫酸葡聚糖:25 g硫酸葡聚糖溶于80 ml ddH2O(定容至100 ml),-20℃保存;杂交液:4×SSC50% 甲酰胺1×Denhardt’s5% 硫酸葡聚糖0.5 mg/ml鲑精DNA4℃保存;BufferI:100 mmol/L Tris-HCl 12.11 g Tris Base150 mmol/L NaCl 8.77 g NaCl用HCl调pH至7.5,定容至1L,高压灭菌,4℃保存,;BufferII:0.5% Blocking agent 0.5 g Blocking agentBufferI 100 ml低温加热溶解,4℃保存一星期;BufferIII:100 mmol/L Tris-HCl 12.11 g Tris Base100 mmol/L NaCl 5.84 g NaCl50 mmol/L MgCl2 10.16 g MgCl2•6H2OddH2O 990 ml(定容至1L)用HCl调pH至9.5,0.45 µm 滤膜过滤,4℃保存;BufferIV:10 mmol/L Tris-HCl, pH8.0, 1 mmol/LEDTA;显色液(NBT/BCIP使用液):20 µl NBT/BCIP + 1ml BufferIII。
核酸探针描述课件

斑点印迹法是一种简单快速的核酸检测方法,其基本原理是将核酸样品直接点到 膜上,然后通过与标记的探针进行杂交,检测目核酸序列。该方法具有操作简 便、快速、高通量等优点,广泛应用于基因诊断、基因表达分析等领域。
微孔板印迹法
总结词
一种将核酸结合到微孔板上的方法,用 于高通量检测多个样本中的特定核酸序 列。
等领域的研究提供了有力支持。
THANKS
感谢观看
核酸探针的应用领域
基因检测与诊断
用于检测基因突变、遗传病、癌症等 疾病相关的基因序列变化,为疾病的
预防、诊断和治疗提供依据。
生物多样性研究
用于检测和鉴定物种的基因组序列, 研究物种的进化、分类和系统发育等
。
食品安全与环境监测
用于检测食品和环境中存在的有害微 生物、病毒和其他病原微生物的核酸 序列,保障食品安全和环境卫生。
探针的纯化与保存
探针的纯化
通过凝胶电泳、亲和层析等方法对标记后的核酸 探针进行纯化,去除杂质和未标记的核酸分子。
探针的保存
将纯化的核酸探针进行分装,并保存在-20℃或80℃冰箱中,以延长探针的保存时间并保持其稳 定性。
03
核酸探针的检测方法
Southern印迹法
总结词
一种将DNA从凝胶转移到膜上的方法,用于检测基因组DNA中的特定序列。
农业科研与育种
用于检测和鉴定农作物及其病原微生 物的基因组序列,研究农作物的遗传 改良和抗病育种等。
02
核酸探针的制备
目的基因的获取
01 基因组DNA提取
从生物样本中提取基因组DNA,作为制备核描述课件
目录
• 核酸探针概述 • 核酸探针的制备 • 核酸探针的检测方法 • 核酸探针的实际应用 • 核酸探针的未来发展
核酸探针技术及应用

核酸探针技术及应用基因检测技术的发展,使对某些疾病的诊断达到了特异性强、敏感性高及简便快速的目的。
近年来各种血清学方法发展很快,但血清学方法主要是测抗体,是间接的证据随着分子生物学的发展,应用DNA—DNA杂交建立了核酸探针(Probe)技术,该技术是目前基因检测最常用的方法,目前已成为诊断各种感染性疾病,恶性肿瘤,遗传病,检测抗生紊的耐药性,法医学鉴定及从分子水平上研究发病机制与流行病学规律等方面的一种重要手段。
本文主要介绍了核酸探针技术的原理,核酸分子杂交方法及核酸探针的应用等方面。
一、核酸探针技术的原理DNA 或RNA 片段能识别特定序列基因的DNA 片段,能与互补的核苷酸序列特异结合,这种用同位素非同位素标记的单链DNA片段即为核酸探针。
核酸探针技术是将双链DNA 经加热或硷处理,使硷基对间的氢链被破坏而变性,解开成两条互补的单链。
它们在一定温度和中性盐溶液条件下,又可按A—T,G—C碱基配对的原则重新组合成双链为复性。
这种重组合只是在两股DNA是互补(同源)或部分互补(部分同源)的条件下才能实现。
正是由于双链DNA的这种可解离与重组合的性质,才可用一条已知的单链DNA,用放射性同位素或其他方法标记后制备成核酸探针,与另一条固定在硝酸纤维素滤膜上的变性单链DNA进行杂交,(另一条DNA链与核酸探针是配对碱基,称为靶)再用放射自显影或其他显色技术检测,以确定有无与探针DNA (或RNA)同源或部分同源的DNA(或RNA)存在。
因为探针只与靶病原体的DNA或RNA杂交,而不与标本中存在的其他DNA 或RNA 杂交。
二、核酸探针技术的基本方法被检标本用去污剂和酶分解以去除非DNA成分或直接提取DNA,用各种方法处理DNA使其变性,把DNA双螺旋的两条链分开,单链DNA结合于固态基质上,(如滤膜)使其固定。
再加上已制备好的探针进行杂交,探针便可找出已固定的DNA中的互补序列,与之配对结合,然后洗掉未结合部分,由于探针已将放射性同位素掺入,再用x射线敏感的胶片自显影,见黑色影印者即为阳性。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
常用核酸探针标记方法
1、切口平移法(nick translation)
原理:先用适量的DNase I 在Mg2+存在下,在双链DNA 上打开若干个单链缺口。
利用E. coli DNA 聚合酶I 的5'- 3' 核酸外切酶活性在切处将旧链从5’-末端逐步切除。
同时DNA 聚合酶I的5'- 3'聚合酶活性的作用下,顺序将dNTP连接到切口的3’-末端-OH上,以互补的DNA 单链为模板合成新的DNA单链。
图1 切口平移法原理示意图
特点:1)各种螺旋状态(超螺旋、闭环及开环)及线性的双链DNA均可作为缺口平移法的标记底物。
但单链DNA和RNA不能采用此方法进标记。
双链DNA小片段(>100-200bp)也不是此法标记的理想底物。
2)DNA多聚酶必须是E·Coli DNA多聚酶的全酶。
3)DNA模板要用纯化过DNA
2、随机引物法(random priming)
原理:将待标记的DNA探针片段变性后与随机引物一起杂交,然后以此杂交的寡核苷酸为引物,大肠杆菌DNA聚合酶I大片段(E·Coli DNA polymerase I Klenow Fragment)的催化下,合成与探针DNA互补的DNA链。
当反应液中含有标记的dNTP时,即形成标记的探针。
图2 随机引物标记法原理示意图
特点:1)除了能进行双链DNA标记外,也可用于单链DNA和RNA探针的标记。
2)所得到的标记产物是新合成的DNA单链,而所加入的DNA片段本身并不能被标记。
3)新形成的标记DNA单链的长度与加入寡核苷酸引物的量成反比,因为加入的寡核苷酸数量越多,合成起点也越多,得到的片段的长度也越短。
按标准方法得到
的标记产物长度一般为200-400bp。
3、末端标记
与切口平移法和随机引物法不同,DNA末端标记法并不将DNA片段的全长进行标记,而是只将其一端(5’或3’端)进行部分标记。
其特点是可得到全长DNA片段,DNA片段并非均匀标记,标记活性不高。
多种酶促反应可用于DNA的末端标记,如T4 DNA聚全酶、T4 多核苷酸激酶、末端脱氧核苷酰转移酶和Klenow DNA聚合酶等。
以下是Klenow DNA 聚合酶进行3’-末端标记的示意图。
图3 用Klenow DNA 聚合酶进行3’-末端填充标记
——以上内容主要摘自《现代分子生物实验技术(第二版)》
佰而林生物科技有限公司技术服务部。
