天根RNA提取试剂盒介绍
天根 一步法逆转录PCR试剂盒说明书

通用型一步法RT-PCR试剂盒说明书前言本试剂盒适用于对各种动、植物、病毒RNA进行PCR 检测。
用户可根据被分析基因,配合一对引物,或同时采用Taqman 荧光探针,先将提取的RNA反转录(reverse transcription).成cDNA,再经过聚合酶链式反应(Polymerase Chain Reaction)技术对特异性片段进行扩增,进行电泳或实时荧光分析。
一步法RT-PCR可使RT及PCR过程在单管中完成,比分开进行RT及PCR更方便。
由于不需要在RT完成后打开反应管,尽可能地避免了样品间的相互污染。
规格20人份适用仪器适用于各种普通PCR仪器和实时荧光PCR仪器。
试剂盒组成试剂准备根据下表配制反应液:振荡混匀后,按每管 40 μl(可根据需要调整)分装,备用。
PCR扩增将均一化后的RNA提取样本10 μl加入各反应管,混匀、离心后,置普通PCR仪器或实时荧光PCR仪器上进行扩增及实时检测。
结果判断扩增产物可直接进行电泳检测;实时荧光检测可根据相应仪器的配套软件进行结果分析。
保存及有效期-20℃保存,有效期为6个月。
注意事项1. 开始检测前请仔细阅读本说明书全文。
2. 整个检测过程中,反应体系的配制、样本处理及加样、PCR扩增(荧光检测)应分区进行以避免污染。
3. 操作人员应戴口罩,经常更换一次性手套,以避免RNA酶的污染;实验中所用器具均应经过除RNA酶处理。
4. 试剂盒组成中的试剂使用前应充分融化并混匀。
5. 进行实时荧光分析时,应使用透光性能较好的一次性薄壁离心管;.荧光探针应避光保存,加入缓冲液中后,应尽快进行扩增。
6. 注意适当处理检测中遗留的样品、扩增产物及可能被污染的试剂。
生产企业:上海蓝创生物科技发展有限公司。
天根切胶回收说明书

天根切胶回收说明书
天根切胶回收试剂盒说明书
一、产品简介
天根切胶回收试剂盒是一种高效的DNA片段回收试剂盒,能够从凝胶中快速、准确地回收目的DNA片段。
该试剂盒采用独特的吸附技术,能够有效地去除凝胶中的杂质,纯化DNA片段。
回收的DNA片段可用于后续的克隆、测序、PCR等实验。
二、产品特点
1. 高效回收:能够从凝胶中快速、准确地回收目的DNA片段,回收率高达90%以上。
2. 纯度高:能够有效去除凝胶中的杂质,纯化DNA片段,提高后续实验的准确性和可靠性。
3. 操作简便:采用独特的吸附技术,无需使用有机溶剂,简化了操作步骤。
4. 稳定性好:试剂盒中的成分稳定,便于长期保存和使用。
三、使用方法
1. 准备凝胶和电泳缓冲液:将凝胶和电泳缓冲液按照要求准备妥当。
2. 切胶:按照实验要求将凝胶切成适当大小的胶块。
3. 洗涤:将切好的胶块放入离心管中,加入适量的洗涤液,充分混匀,洗涤去除凝胶中的杂质。
4. 吸附:将洗涤后的胶块放入吸附柱中,按照说明书要求进行吸附操作。
5. 洗脱:将吸附后的DNA片段进行洗脱,收集洗脱液。
6. 检测:对回收的DNA片段进行检测,如琼脂糖凝胶电泳或紫外分光光度计检测。
四、注意事项
1. 使用前请仔细阅读本说明书,确保按照说明书要求正确操作。
2. 本试剂盒仅供实验室使用,请勿用于食品、药品等领域。
3. 使用过程中请注意安全,避免直接接触眼睛、皮肤和衣物等。
如不慎接触到皮肤或眼睛,请立即用清水冲洗,并及时就医。
4. 本试剂盒中的试剂和耗材均为一次性使用,用后请及时处理废弃物。
天根说明书RNA

RP110413RNAsimple Total RNA KitCat. no. DP419Kit ContentsBuffer RZ should be stored protected from light at 2-8°C; others stored at room temperature (15-25°C).IntroductionRNAsimple Total RNA Kit uses a new RNA isolation technology based on Guanidine Thiocyanate / Phenyl method. It contains a unique buffer RZ that minimizes the contamination of genomic DNA and protein. RNAsimple Total RNA Kit can efficiently isolates high pure RNA from blood, cells, tissues and plant samples in one hour. The purified RNA is ready-to-use in downstream applications such as: RT-PCR and real-time RT-PCR, gene-chips assay, northern blot, dot blot, polyA screening, in vitro transcript, and molecular cloning.Important NoteFor isolation of bacterial RNA, RNAprep pure Kit (For Cell/Bacteria) should be used (Cat.no. DP430).Notes of preventing RNase contamination1.Change gloves regularly. Bacteria on the skin can result inRNase contamination.e RNase-free plastic and tips to avoid cross contamination.3.RNA can be protected in TRNzol. But RNA must be stored orprocessed in RNase-free plastic or glassware. To wipe off RNase, the glassware can be roasted at 150℃for 4 hours, while plastic can be dipped in 0.5 M NaOH for 10min, washed by RNase-free ddH2O thoroughly, and sterilized.e RNase-free ddH2O to confect solution.(Add DEPC intowater in clean glass container to a final concentration of 0.1% (v/v). Incubate overnight and autoclave for 15 min to remove any trace of DEPC.)ProtocolBuffer RD and Buffer RW are supplied as a concentrate. Before using for the first time, add ethanol (96–100%) as indicated on the bottle to obtain a working solution.1.Homogenizing samples.a.Plant (take leaves as an example): Place fresh leaves inliquid nitrogen and grind thoroughly with a mortar andpestle, or grind in Buffer RZ after cut leaves into pieces.This process is suggested to be finished within one minute.Use 1 ml Buffer RZ per 100 mg leaves.b.Tissues (take rat liver as an example): Add 1 ml Buffer RZfor per 30–50 mg of liver sample. Homogenize sampleusing a power homogenizer. Usually, the volume of tissue sample should not exceed 10% of the volume of Buffer RZ.c.Adherent Cells (do not use more than 1 × 107 cells): Cellsgrown in a monolayer in cell-culture vessels can be either lysed directly in the vessel (up to 10 cm diameter) or trypsinized and collected as a cell pellet prior to lysis. (Cells grown in a monolayer in cell-culture flasks should always be trypsinized. )1)Method A: To lyse cells directly. Add 1 ml Buffer RZdirectly to the cells in the culture dish per 10 cm2ofculture dish surface area. Pipette the lysate up anddown several times. Note: the volume of Buffer RZshould be determined according to the surface areainstead of the number of cells. An insufficient volumecan result in DNA contamination of isolated RNA.2)Method B: To trypsinize and collect cells. Determinethe number of cells. Aspirate the medium, and washthe cells with PBS. Aspirate the PBS, and add 0.10–0.25% trypsin in PBS. After the cells detach from thedish or flask, add medium (containing serum toinactivate the trypsin), transfer the cells to an RNase-free glass or polypropylene centrifuge tube (notsupplied), and centrifuge at 300 x g for 5 min.Completely aspirate the supernatant.Note: Make sure that the supernatant has beencompletely removed. Residual medium could lead toincomplete lysis of cells and reduced yield of RNA. d.Suspension Cells: Harvest cells by centrifugation andremove culture medium. Add 1 ml of Buffer RZ per 5 × 106- 107cells from animal, plant or yeast, or 1 × 107 cells of bacterial. Do not wash cells before addition of Buffer RZ toavoid increased chance of mRNA degradation. Samplesfrom some yeast and bacteria maybe need to behomogenized by using a power homogenizer.e.Blood: Take fresh blood, and add three volumes of BufferRZ. Mix thoroughly. (Recommended amount: 0.75 mlBuffer RZ for 0.25 whole blood)2.Incubate homogenized samples at 15-30℃for 5 min, topermit complete dissociation of the nucleoprotein complex.3.Optional step: Centrifuge the sample at 12,000 rpm (~13,400 ×g)for 10 minutes at 4°C. Transfer the supernatant to a freshmicro-centrifuge tube.Note: When preparing samples with high content of fat, proteins, polysaccharides, or extracellular material (e.g., muscle, fat tissue, or tuberous plant material), an additional centrifugation may be required to remove insoluble material from the samples.RNA remains in the upper aqueous phase after centrifugation. However, when dealing with fat tissue, the upper phase is a lipid layer that should be discarded.Retain the clean homogenizing part for next step.4.Add 200 µl of chloroform per 1 ml Buffer RZ used forhomogenization. Cap the tube securely and vortex for 15 s.Incubate for 3 minutes at room temperature.Note: If vortexing is not applicable, shake tube vigorously by hand for 2 min.5.Centrifuge the sample for 10 min at 12,000 rpm (~13,400 × g)at 4°C. The mixture separates into a lower yellow phenol-chloroform phase, an interphase, and a colorless upper aqueous phase. RNA remains exclusively in the aqueous phase.Pipette the aqueous phase out into a new tube.6.Add the 0.5 volume ethanol (96%-100%) to the aqueous phase.Mix thoroughly (participate may appear in this step). Transferthe sample, including any precipitate that may have formed, to an RNase-free Spin Column CR3 placed in a 2 ml RNase-free Collection Tube. Close the lid gently, and centrifuge at 12,000 rpm (~13,400 × g) for 30 s at 4°C. Discard the flow-through.Note: If the sample is more than 700µl, transfer the sample to CR3 in two times and centrifuge separately.7.Add 500 μl Buffer R D to the RNase-free Spin Column CR3(Ensure ethanol has been added). Close the lid gently, and centrifuge at 12,000 rpm (~13,400 × g) for 30 s at 4°C. Discard the flow-through.8.Add 700 μl Buffer RW to the RNase-free Spin Column CR3(Ensure ethanol has been added). Close the lid gently, and centrifuge at 12,000 rpm (~13,400 × g) for 30 s at 4°C. Discard the flow-through.9.Add 500 μl Buffer RW to the RNase-free Spin Column CR3.Close the lid gently, and centrifuge at 12,000 rpm (~13,400 × g) for 30 s at 4°C. Discard the flow-through.10.Set the RNase-free Spin Column CR3 back to the CollectionTube. Centrifuge at 12,000 rpm (~13,400 × g) for 2 min at 4°C to dry the spin column membrane.Note: The long centrifugation dries the spin column membrane, ensuring that no ethanol is carried over during RNA elution. Residual ethanol may interfere with downstream reactions.11.Place the RNase-free Spin Column CR3 in a new 1.5 ml RNase-free Collection Tube (supplied). Add 30-100 μl RNase-free ddH2O directly to the spin column membrane. Close the lid gently, incubate at room temperature (15–25°C) for 2 min.Centrifuge at 12,000 rpm (~13,400 × g) for 2 min at 4°C to elute the RNA.Note: The volume of elution buffer should not be less than30 μl, or it may affect recovery efficiency. To obtain higherproductivity, add the solution gained from step 11 to the center of membrane again, let the columns stand for 1 min, and then centrifuge.Purified RNA should be stored at –70°C.Ordering InformationRNA IsolationRNAstore Reagent 20 ml100 ml DP408-01 DP408-02Reverse TranscriptionQuantscript RT Kit 20 μl × 25 rxns20 μl × 100 rxns KR103-03 KR103-04Real–Time PCRSuperReal PreMix (SYBR Green) 50 μl × 50 rxns50 μl × 200 rxns FP204-01 FP204-02Quant One Step qRT-PCR (SYBR Green) Kit 50 μl × 50 rxns FP303-01。
病毒基因组RNA提取试剂盒使用说明书
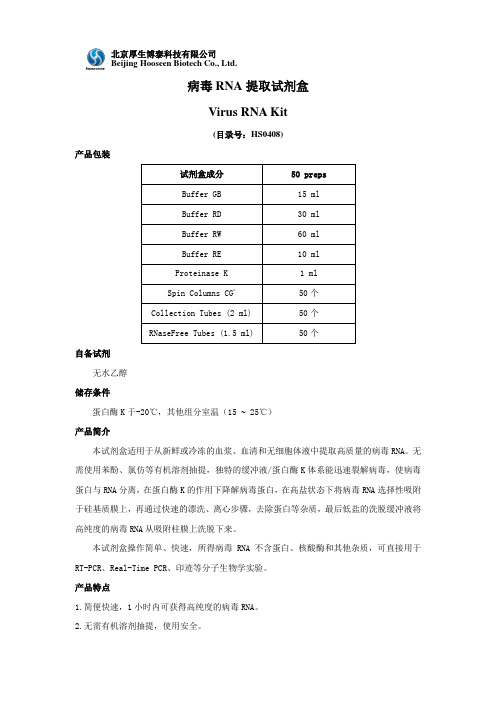
病毒RNA 提取试剂盒Virus RNA Kit(目录号:HS0408)产品包装自备试剂无水乙醇储存条件蛋白酶K 于-20℃,其他组分室温(15 ~ 25℃)产品简介本试剂盒适用于从新鲜或冷冻的血浆、血清和无细胞体液中提取高质量的病毒RNA 。
无需使用苯酚、氯仿等有机溶剂抽提,独特的缓冲液/蛋白酶K 体系能迅速裂解病毒,使病毒蛋白与RNA 分离,在蛋白酶K 的作用下降解病毒蛋白,在高盐状态下将病毒RNA 选择性吸附于硅基质膜上,再通过快速的漂洗、离心步骤,去除蛋白等杂质,最后低盐的洗脱缓冲液将高纯度的病毒RNA 从吸附柱膜上洗脱下来。
本试剂盒操作简单、快速,所得病毒RNA 不含蛋白、核酸酶和其他杂质,可直接用于RT-PCR 、Real-Time PCR 、印迹等分子生物学实验。
产品特点1.简便快速,1小时内可获得高纯度的病毒RNA 。
2.无需有机溶剂抽提,使用安全。
北京厚生博泰科技有限公司 Beijing Hooseen Biotech Co., Ltd.3.重复性好,产量高。
4.所得病毒RNA纯度高,无污染物和抑制剂,方便下游应用。
注意事项1.血清或血浆避免反复冻融,否则会使蛋白变性或产生沉淀,导致提取的RNA片段小,提取量下降。
2.如缓冲液Buffer GB、Buffer RD结晶或产生沉淀,可在56℃水浴溶解。
3.所有离心步骤均为室温下操作。
4.本试剂盒也可提取高质量的病毒DNA,操作步骤相同。
操作步骤1. 取1.5 ml离心管(自备),加入20 ul的Proteinase K溶液。
2. 向离心管中加入200 ul血清或血浆,然后再加入200 ul Buffer GB,涡旋震荡15 sec。
(注意:1、样本体积不足200 ul可以加入0.9% NaCl(自备)补足。
2、为确保样本有效裂解,加入Buffer GB后,需将样本与Buffer GB充分混匀。
)3.56℃孵育15 min,短暂离心,将管壁上的溶液收集到管底。
rna提取试剂盒原理

rna提取试剂盒原理RNA提取试剂盒是用于从细胞或组织样品中提取RNA的一种化学试剂盒。
RNA提取是分子生物学和基因表达研究的重要步骤之一,能够有效地分离RNA并去除DNA、蛋白质和其他杂质。
本文将详细介绍RNA提取试剂盒的原理。
一、RNA提取试剂盒的组成RNA提取试剂盒主要由以下几个部分组成:1. 细胞裂解缓冲液:用于破坏细胞膜和核膜,释放出细胞内的RNA。
2. 溶解液:用于溶解破坏后的细胞和组织样品中的核酸。
3. 酚/氯仿:用于分离RNA。
4. 筛选柱:用于去除DNA、蛋白质和其他杂质。
5. 洗涤缓冲液:用于洗涤筛选柱,去除残留杂质。
6. 去离子水:用于洗涤筛选柱后洗脱纯化后的RNA。
二、RNA提取试剂盒的原理1. 细胞裂解首先,需要将样品中的细胞或组织裂解,释放出RNA。
细胞裂解缓冲液中包含有破坏剂,如SDS和EDTA等,能够破坏细胞膜和核膜,并释放出细胞内的RNA。
2. 核酸溶解经过细胞裂解后,需要将样品中的核酸溶解。
溶解液中含有高浓度的盐和pH调节剂,能够使DNA和RNA分子变性并溶于水相中。
3. RNA分离接下来,需要将RNA与DNA、蛋白质和其他杂质分离。
酚/氯仿是一种常用的分离试剂,可以将RNA从DNA、蛋白质和其他杂质中分离出来。
酚/氯仿具有不同的密度,在试管中形成两个不同层次的相,上层为水相(含有RNA),下层为有机相(含有DNA、蛋白质和其他杂质)。
这样就可以通过移液器或吸管将上层水相取出。
4. RNA纯化虽然通过酚/氯仿可以将RNA分离出来,但仍然存在一些杂质。
因此需要进行纯化步骤。
筛选柱中含有硅胶或磁性珠等材料,能够吸附RNA并去除DNA、蛋白质和其他杂质。
经过洗涤缓冲液的洗涤后,去离子水可以将纯化后的RNA从筛选柱中洗脱。
三、RNA提取试剂盒的应用RNA提取试剂盒广泛应用于基因表达研究、分子生物学和遗传学等领域。
例如,可以用于分离不同组织或细胞类型中的RNA,并进行基因表达谱分析;也可以用于研究基因转录调控机制等。
天根RNA提取说明书

2) 胰蛋白酶处理法:确定细胞数量,吸除培养基,用PBS 洗涤细胞,吸除PBS ,向细胞中加入含有0.1-0.25%胰蛋白酶的PBS 处理细胞,当细胞脱离容器壁时,加入含有血清的培养基失活胰蛋白酶,将细胞溶液转移至RNase-Free 的离心管中,300×g 离心5 min ,收集细胞沉淀,仔细吸除所有上清。
注意:收集细胞时一定要将细胞培养液去除干净,否则会导致裂解不完全,造成RNA 的产量降低。
d. 细胞悬液:离心取细胞。
每5×106-107动物细胞和植物细胞加入1 ml TRNzol-A +。
加入TRNzol-A +前不要洗涤细胞,以免降解mRNA 。
e. 血液处理:直接取新鲜的血液,加入3倍体积TRNzol-A +(推荐0.25ml 全血加入0.75 mlTRNzol-A +),充分振荡混匀。
2. 将匀浆样品在15-30℃放置5 min ,使得核酸蛋白复合物完全分离。
3. 可选步骤:4℃ 12,000 rpm(~13,400×g) 离心10 min ,取上清。
注意:如果样品中含有较多蛋白、脂肪、多糖或肌肉、植物结节部分等,可离心去除。
离心得到的沉淀中包括细胞外膜、多糖、高分子量DNA ,上清中含有RNA 。
处理脂肪组织样品时,上层是大量油脂,应除去。
取澄清的匀浆溶液进行下一步操作。
4. 每使用1 ml TRNzol-A +加0.2 ml 氯仿,盖好管盖,剧烈振荡15 sec ,室温放置3 min 。
注意:如不能旋涡混匀,可手动快速颠倒混匀2 min 。
5. 4℃ 12,000 rpm(~13,400×g)离心10-15 min 。
样品会分成三层:黄色的有机相,中间层和上层无色的水相,RNA 主要在水相中,把水相(约500 μl )转移到新管中。
(如果要分离DNA 和蛋白质,可向天根公司索取提取方法)。
6. 在得到的水相溶液中加入等体积异丙醇,混匀,室温放置20-30 min 。
天根—RNA提取方法

注意事项1.匀浆后,加氯仿前,样品可在-70℃放置一个月。
2.RNA沉淀可以保存在75%乙醇中,2-8℃一个星期以上或-20℃一年。
3.若提取细菌RNA,推荐应用RNAprep pure培养细胞/细菌总RNA提取试剂盒(目录号:DP430)。
预防RNase污染,应注意以下几方面:1.经常更换新手套。
因为皮肤经常带有细菌,可能导致RNase污染。
2.使用RNase-Free的塑料制品和枪头避免交叉污染。
3.RNA在TRNzol–A+试剂中时不会被RNase降解。
但提取后继续处理过程中应使用RNase-Free的塑料和玻璃器皿。
玻璃器皿可在150℃烘烤4h,塑料器皿可在0.5 M NaOH中浸泡10 min,然后用水彻底清洗,再灭菌,即可去除RNase。
4.配制溶液应使用RNase-Free ddH2O。
(将水加入到干净的玻璃瓶中,加入DEPC至终浓度0.1%(V/V),放置过夜,高压灭菌)。
RNA提取操作步骤准备试剂:氯仿、异丙醇、RNase-Free ddH2O 、75%乙醇(用RNase-Free ddH2O配制)。
1. 样品处理a. 植物组织:以叶片RNA提取为例。
取新鲜叶片在液氮中充分研磨或将叶片剪碎后直接在TRNzol–A+中研磨,研磨要迅速,最好不要超过1 min。
大约100 mg 叶片使用1 mlTRNzol–A+。
b. 动物组织:以鼠肝脏RNA提取为例。
取新鲜或-70℃冻存组织,每30-50 mg组织加入1 mlTRNzol–A+,用匀浆仪进行匀浆处理。
样品体积一般不要超过TRNzol–A+体积的10%。
c. 单层培养细胞:单层贴壁细胞的收集(收集细胞数量请不要超过1×107):可直接在培养容器中裂解(容器体积不超过10cm2),或者使用胰蛋白酶处理后离心收集细胞沉淀。
(在摇瓶中培养的单层贴壁细胞通常采用胰蛋白酶处理的方法)。
1) 直接裂解法:直接在培养板中加入TRNzol–A+裂解细胞,每10 cm2面积加入1 mlTRNzol–A+。
天根DP117-无内毒素质粒大提试剂盒说明书

For personal use only in study and research; not for commercial use产品简介本试剂盒采用独特的硅胶模吸附技术,高效专一地结合质粒DNA。
同时采用特殊的溶液P4和过滤器CS1,可有效的出去内毒素、蛋白质杂志;整个提取过程仅需1h,方便快捷。
使用本试剂盒提取的质粒DNA可适用于各种常规操作,包括酶切、PCR、测序、连接、转化和细胞转染多种细胞等试验。
推荐每次菌液使用量:高拷贝质粒推荐使用量为100ml,得率一般在500-1500μg左右;低拷贝质粒推荐使用量为200ml,得率一般在200-600μg左右。
注意事项:请务必在使用本试剂盒之前阅读此注意事项。
1.溶液P1在使用前先加入RNase A (将试剂盒中提供的RNase A全部加入),混匀,置于2-8℃保存。
2.第一次使用前应按照试剂瓶标签的说明先在漂洗液PW中加入无水乙醇。
3.使用前先检查平衡液BL,溶液P2和P4是否出现结晶或者沉淀,如有结晶或者沉淀现象,可在37℃水浴中加热几分钟,即可恢复澄清。
4.注意不要直接接触溶液P2和P4,使用后应立即盖紧盖子。
5.使用过滤器时请将推柄小心缓慢地从过滤管中抽出,避免滤膜因压力而松动。
6.提取的质粒量与细菌培养浓度,质粒拷贝数等因素有关。
如果所提质粒为低拷贝质粒或大于10kb的大质粒,应加大菌体使用量,同时按比例增加P1、P2、P4的用量;洗脱缓冲液推荐在65-70℃水浴中预热,可以适当延长吸附和洗脱时间,以提高提取效率。
7.实验前使用平衡液BL处理吸附柱,可以最大限度激活硅基质膜,提高得率。
8.用平衡液处理过的柱子最好立即使用,放置时间过长会影响使用效果。
质粒DNA 浓度及纯度检测得到的质粒DNA可用琼脂糖凝胶电泳和紫外分光光度计检测浓度与纯度。
OD260值为1相当于大约50μg/ml双链DNA。
纯化的质粒DNA OD260/OD280通常在1.8-2.0左右,可直接应用于细胞转染甚至动物体内实验等对DNA纯度要求很高的实验中。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
天根RNA提取试剂盒介绍
天根RNA提取试剂盒是一种高效、快速和方便的实验试剂盒,用于从多种生物样品中提取RNA。
该试剂盒可广泛应用于分子生物学研究、基因表达分析、转录组学研究等领域。
下面将对天根RNA提取试剂盒进行详细介绍。
一、产品特点
1.高质量RNA提取:利用高效的离心柱,能够高效地纯化RNA,提供高质量的RNA样品。
2.快速操作:只需几个简单的步骤,即可从样品中提取到RNA,并且整个过程耗时较短。
3.高纯度RNA:通过RNA与DNA、蛋白质等细胞组分的选择性结合,能够有效减少杂质的污染,得到高纯度的RNA。
4.广泛的样品适用性:适用于多种样品,包括动植物组织、微生物、血液、细胞培养物、土壤等。
5.高适应性:适应于不同量级的样品,可提取微量到大量的RNA。
二、工作原理
1.细胞裂解:首先,将待提取的样品中的细胞裂解,使RNA从细胞内释放出来。
2. 去除DNA和蛋白质:通过使用DNAse和蛋白酶处理,可以去除样品中的DNA和蛋白质,保证纯化后的RNA质量。
3.RNA结合:将样品中的RNA与离心柱中的RNA结合缓冲液混合,使RNA选择性地与柱内载体结合。
4.洗涤:将样品通过离心柱进行洗涤,去除杂质,保留结合的RNA。
5. Elution:最后,通过加入高纯度的RNase-free水,将结合的RNA从离心柱中洗脱出来。
三、操作步骤
1.样品裂解:将待提取的样品裂解,释放出RNA。
2.DNA和蛋白质去除:通过添加DNA酶和蛋白酶,去除样品中的DNA 和蛋白质。
3.RNA结合:将样品与RNA结合缓冲液混合,使RNA选择性地与柱内的载体结合。
4.洗涤:通过洗涤步骤,去除杂质,保留结合的RNA。
5. 洗脱:最后,使用无RNase的溶液将RNA从离心柱中洗脱出来。
四、注意事项
1.使用前需充分均匀的振荡试剂瓶。
2. 避免样品的污染,使用无RNase和DNase的工具和试剂。
3.不要使用高于建议温度的洗涤缓冲液和洗涤液。
4.严格按照说明书的步骤进行操作,避免操作的误差。
5.存储试剂盒时,需按照建议的温度和条件进行保存,以确保试剂盒的质量。
总结:
天根RNA提取试剂盒是一种高效、快速和方便的试剂盒,适用于多种
生物样品中RNA的提取。
其具有高质量RNA提取、快速操作、高纯度RNA、广泛的样品适用性和高适应性等特点。
通过操作步骤中的细胞裂解、去除DNA和蛋白质、RNA结合、洗涤和洗脱等步骤,可以得到高质量的纯化
RNA样品。
但在操作中需注意一些注意事项,以确保提取的RNA质量和试
剂盒的稳定性。
天根RNA提取试剂盒在分子生物学研究中有着广泛的应用
前景。
