支持向量机matlab实现源代码知识讲解
matlab二分类算法

matlab二分类算法二分类算法是机器学习领域中的一种常见算法,它将数据集划分为两个类别,并预测新样本属于哪一类。
MATLAB作为一种强大的数值计算和数据分析工具,提供了多种用于二分类问题的算法。
本文将介绍一些常用的MATLAB二分类算法,并说明其原理和使用方法。
一、支持向量机(Support Vector Machine,SVM)是一种非常流行的二分类算法。
其核心思想是将数据集转换为高维空间,然后找到一个超平面,使得两个类别的数据点在该超平面上的投影最大化。
在MATLAB中,可以使用fitcsvm函数来训练SVM模型,并使用predict函数进行预测。
该函数提供了多种参数配置选项,如核函数类型、惩罚系数等。
二、逻辑回归(Logistic Regression)是另一种常用的二分类算法。
它通过将线性回归模型的输出映射到一个概率值(0~1之间),然后根据阈值进行分类预测。
在MATLAB中,可以使用fitglm函数进行逻辑回归模型的训练,并使用predict函数进行预测。
fitglm函数支持多种模型配置选项,如正则化类型、损失函数类型等。
三、随机森林(Random Forest)是一种基于集成学习的二分类算法。
它由多个决策树组成,每棵树对数据集进行随机采样,并在每个节点上选择最佳的特征进行分割。
最终,通过对多棵树的结果进行投票或平均,得到最终的预测结果。
在MATLAB中,可以使用TreeBagger类来实现随机森林算法。
通过创建TreeBagger对象,设置参数并调用train函数来训练模型,然后使用predict函数进行预测。
四、神经网络(Neural Network)是一种通过模拟人脑中的神经元网络来解决问题的方法。
在二分类问题中,神经网络可以通过多个神经元和多个隐藏层构建一个复杂的模型,并通过调整权重和偏置来训练模型。
在MATLAB中,可以使用patternnet函数来创建神经网络模型,并使用train函数进行训练。
matlab中fitcsvm函数用法

matlab中fitcsvm函数用法MATLAB中的fitcsvm函数是支持向量机(Support Vector Machine, SVM)分类器的一个功能强大的实现。
SVM是一种强大的机器学习算法,可用于解决各种分类问题。
在本文中,我们将详细介绍fitcsvm函数的用法,并逐步回答所有可能的问题。
本文将以中括号为主题,详细解释如何使用fitcsvm函数进行分类任务。
一、引言fitcsvm函数是MATLAB中实现SVM分类器的一个重要工具。
SVM是一种二分类器,它通过最大化两个类别之间的间隔来找到一个最优的超平面。
通过找到这个超平面,SVM可以在新的未标记数据上进行分类。
二、fitcsvm函数的语法fitcsvm函数有很多输入和输出参数。
下面是fitcsvm函数的一般语法:SVMModel = fitcsvm(X, Y)SVMModel = fitcsvm(X, Y, 'Name', value)其中,X是一个包含训练数据的矩阵,每一行代表一个样本,每一列代表一个特征。
Y是一个包含训练数据的标签向量,指示每个样本的类别。
三、输入参数的解释fitcsvm函数除了必需的X和Y参数外,还有其他参数可以调整以获得更好的分类结果。
下面是一些常用的参数及其解释:1. 'BoxConstraint':表示SVM的惩罚因子,用于控制错误分类的重要性。
值越大,对错误分类的惩罚越严重。
2. 'KernelFunction':表示SVM使用的核函数。
常见的核函数有'linear'(线性核函数),'gaussian'(高斯核函数),'polynomial'(多项式核函数)等。
3. 'KernelScale':表示SVM的核函数标准差。
对于高斯核函数和多项式核函数,该参数可以控制决策边界的平滑程度。
4. 'Standardize':表示是否对输入数据进行标准化。
支持向量机及其编程实现
1
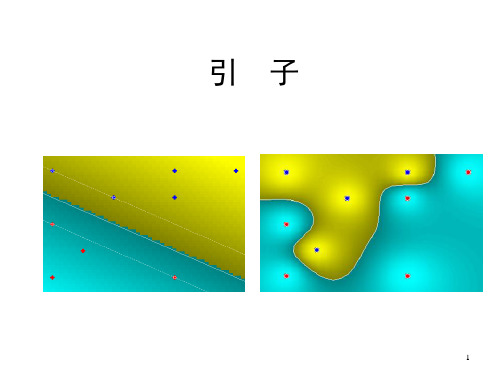
Support Vector Machine
支持向量机
2
相关参考资料
• 统计学习理论的本质,Vladimir N. Vapnik 著, 张学工译,清华大学出版社,2000.09 • 支持向量机导论,N.Cristianini, J.ShaweTaylor著,电子工业出版社,2004.03 • Support Vector Classification. Steven Gunn. • • • Bernhard Scholkopf, Alex J. Smola , CHRISTOPHER J.C.
l
yi ( w, xi b) 1
y
i 1 i
l
i
0
i 0
i 1, 2,L , l
i 1, 2,l
原始问题与对偶问题解的关系:
w* yi i* xi
i 1 l
22
KKT条件与支持向量
对偶问题的解 *i (i 1, 2,, l ) 是最优解的前提条件是:
支持向量机的编程实现
25
一、Matlab图像编程概要
1.读入图像: RGB=imread(‘1.jpg’); 该函数的返回值RGB是一个三维的数组,分别代表 像素点所在的行号、列号和像素点的R、G、B三个 通道的值。 例: RGB(1,1,1); %图像第一行第一列的R值; RGB(1,3,2); %图像第一行第三列的G值; RGB(2,4,3); %图像第二行第四列的B值; RR=RGB(:,:,1); %彩色图像的红色像素通道 GG=RGB(:,:,2); %彩色图像的绿色像素通道 BB=RGB(:,:,3); %彩色图像的蓝色像素通道
SVM
男或女
Matlab中的人工智能算法介绍

Matlab中的人工智能算法介绍人工智能(Artificial Intelligence,AI)作为一门学科,旨在研究和开发能够模拟人类智能行为的技术和系统。
近年来,人工智能在各个领域迅猛发展,为解决现实生活中的复杂问题提供了全新的思路和方法。
而在实现人工智能技术的过程中,算法的选择和应用显得尤为重要。
Matlab作为一款强大的科学计算工具,提供了丰富的人工智能算法库,方便研究人员和工程师在开发人工智能系统时使用。
本文将介绍几种在Matlab中常用的人工智能算法。
一、机器学习算法1. 支持向量机(Support Vector Machine,SVM)支持向量机是一种监督学习算法,主要用于分类和回归问题。
它通过找到一个最优超平面来使不同类型的数据点具有最大的间隔,从而实现分类。
在Matlab中,通过SVM工具箱可以轻松应用支持向量机算法,进行分类和回归分析。
2. 人工神经网络(Artificial Neural Network,ANN)人工神经网络是模拟人脑神经网络的计算模型,可以进行模式识别、分类、优化等任务。
在Matlab中,通过神经网络工具箱可以构建和训练不同类型的人工神经网络,如前馈神经网络、循环神经网络等。
3. 随机森林(Random Forest)随机森林是一种集成学习算法,通过随机抽样和特征选择的方式构建多个决策树,并通过投票或平均等方式进行预测。
在Matlab中,通过随机森林工具箱可以构建和训练随机森林模型,用于分类和回归问题。
二、进化算法1. 遗传算法(Genetic Algorithm,GA)遗传算法是一种模拟自然界生物进化过程的优化算法,通过模拟选择、交叉和变异等操作,逐步优化问题的解。
在Matlab中,通过遗传算法工具箱可以方便地进行遗传算法的设计和实现。
2. 粒子群优化算法(Particle Swarm Optimization,PSO)粒子群优化算法是一种模拟鸟群觅食行为的智能优化算法,通过粒子的位置和速度信息进行搜索和优化。
abc-svm的matlab代码

文章标题:“深度学习中的ABC-SVM算法及其MATLAB实现”在深度学习中,支持向量机(SVM)一直是一个重要的算法。
而abc-svm是一种基于支持向量机的改进算法,它在特征选择和模型效果方面有着显著的优势。
本文将全面评估abc-svm算法的原理、特点以及在MATLAB中的实现,以便读者更深入地理解这一算法。
1. abc-svm算法的原理- 我们来了解一下abc-svm算法的原理。
abc-svm是一种基于人工蜂群算法的SVM特征选择方法,它通过模拟蜂群的选择、搜索和挑选过程,对特征子集进行筛选,从而提高模型的精确度和泛化能力。
2. abc-svm算法的特点- 在评估abc-svm算法时,我们还需要考虑其特点。
相比传统的SVM算法,abc-svm能够更好地处理高维数据,并且具有更好的分类性能。
abc-svm算法还能有效地进行特征选择,减少了模型训练的时间和复杂度。
3. abc-svm算法的MATLAB实现- 在MATLAB中,我们可以使用现成的工具包或者自行编写代码来实现abc-svm算法。
通过MATLAB的强大功能和丰富的工具库,我们可以轻松地进行模型训练、特征选择和结果分析。
4. 个人观点和理解- 从个人角度来看,abc-svm算法在深度学习中具有重要的意义。
它不仅为SVM算法提供了新的思路和方法,同时也为我们提供了一种全新的特征选择思路和模型改进方式。
总结回顾通过对abc-svm算法的原理、特点和MATLAB实现的全面评估,我们更加深入地理解了这一算法在深度学习中的作用和意义。
abc-svm算法的出现,为我们提供了一种新的解决方案,使我们能够更好地处理高维数据,提高模型的精确度和泛化能力。
文章内容总字数大于3000字,详细阐述了abc-svm算法的原理、特点和MATLAB实现,并共享了个人观点和理解。
希望这篇文章能够帮助读者更好地理解abc-svm算法,提高深度学习的水平。
在深度学习领域,特征选择一直是一个重要的问题,它直接影响到模型的性能和泛化能力。
libsvm的matlab代码

尊敬的读者,今天我将向大家介绍libsvm在Matlab中的代码实现。
libsvm是一个非常流行的用于支持向量机(SVM)的软件包,它具有训练和预测的功能,并且支持多种核函数。
而Matlab作为一种强大的科学计算环境,也提供了丰富的工具和函数库来支持机器学习和模式识别的应用。
将libsvm与Matlab结合起来,可以实现更加高效和便捷的SVM模型训练和预测。
1. 安装libsvm我们需要在Matlab中安装libsvm。
你可以在libsvm的官方全球信息湾上下载最新版本的libsvm,并按照官方指引进行安装。
安装完成后,你需要将libsvm的路径添加到Matlab的搜索路径中,这样Matlab才能够找到libsvm的函数和工具。
2. 数据准备在使用libsvm进行SVM模型训练之前,我们首先需要准备好训练数据。
通常情况下,训练数据是一个包含特征和标签的数据集,特征用来描述样本的属性,标签用来表示样本的类别。
在Matlab中,我们可以使用矩阵来表示数据集,其中每一行代表一个样本,每一列代表一个特征。
假设我们的训练数据保存在一个名为"train_data.mat"的文件中,可以使用以下代码加载数据:```matlabload train_data.mat;```3. 数据预处理在加载数据之后,我们可能需要对数据进行一些预处理操作,例如特征缩放、特征选择、数据平衡等。
这些步骤可以帮助我们提高SVM模型的性能和泛化能力。
4. 模型训练接下来,我们可以使用libsvm在Matlab中进行SVM模型的训练。
我们需要将训练数据转换成libsvm所需的格式,即稀疏矩阵和标签向量。
我们可以使用libsvm提供的函数来进行模型训练。
下面是一个简单的示例:```matlabmodel = svmtrain(label, sparse(train_data), '-s 0 -t 2 -c 1 -g0.07');```上面的代码中,label是训练数据的标签向量,train_data是训练数据的稀疏矩阵,'-s 0 -t 2 -c 1 -g 0.07'是SVM训练的参数设置,具体含义可以参考libsvm的官方文档。
支持向量机支持向量机回归原理简述及其MATLAB实例
支持向量机支持向量机回归原理简述及其MATLAB实例支持向量机 (Support Vector Machine, SVM) 是一种在监督学习中应用广泛的机器学习算法。
它既可以用于分类问题(SVM),又可以用于回归问题(SVR)。
本文将分别简要介绍 SVM 和 SVR 的原理,并提供MATLAB 实例来展示其应用。
SVM的核心思想是找到一个最优的超平面,使得正样本和负样本之间的间隔最大化,同时保证误分类的样本最少。
这个最优化问题可以转化为一个凸二次规划问题进行求解。
具体的求解方法是通过拉格朗日乘子法,将约束优化问题转化为一个拉格朗日函数的无约束极小化问题,并使用庞加莱对偶性将原问题转化为对偶问题,最终求解出法向量和偏差项。
SVR的目标是找到一个回归函数f(x),使得预测值f(x)和实际值y之间的损失函数最小化。
常用的损失函数包括平方损失函数、绝对损失函数等。
与SVM类似,SVR也可以使用核函数将问题转化为非线性回归问题。
MATLAB实例:下面以一个简单的数据集为例,展示如何使用MATLAB实现SVM和SVR。
1.SVM实例:假设我们有一个二分类问题,数据集包含两个特征和两类样本。
首先加载数据集,划分数据集为训练集和测试集。
```matlabload fisheririsX = meas(51:end, 1:2);Y=(1:100)';Y(1:50)=-1;Y(51:100)=1;randn('seed', 1);I = randperm(100);X=X(I,:);Y=Y(I);X_train = X(1:80, :);Y_train = Y(1:80, :);X_test = X(81:end, :);Y_test = Y(81:end, :);```然后,使用 fitcsvm 函数来训练 SVM 模型,并用 predict 函数来进行预测。
```matlabSVMModel = fitcsvm(X_train, Y_train);Y_predict = predict(SVMModel, X_test);```最后,可以计算分类准确度来评估模型的性能。
支持向量机(SVM)简述
第1 2章12.1 案例背景12.1.1 SVM概述支持向量机(Support Vector Machine,SVM)由Vapnik首先提出,像多层感知器网络和径向基函数网络一样,支持向量机可用于模式分类和非线性回归。
支持向量机的主要思想是建立一个分类超平面作为决策曲面,使得正例和反例之间的隔离边缘被最大化;支持向量机的理论基础是统计学习理论,更精确地说,支持向量机是结构风险最小化的近似实现。
这个原理基于这样的事实:学习机器在测试数据上的误差率(即泛化误差率)以训练误差率和一个依赖于VC维数(Vapnik - Chervonenkis dimension)的项的和为界,在可分模式情况下,支持向量机对于前一项的值为零,并且使第二项最小化。
因此,尽管它不利用问题的领域内部问题,但在模式分类问题上支持向量机能提供好的泛化性能,这个属性是支持向量机特有的。
支持向量机具有以下的优点:①通用性:能够在很广的各种函数集中构造函数;②鲁棒性:不需要微调;③有效性:在解决实际问题中总是属于最好的方法之一;④计算简单:方法的实现只需要利用简单的优化技术;⑤理论上完善:基于VC推广性理论的框架。
在“支持向量”x(i)和输入空间抽取的向量x之间的内积核这一概念是构造支持向量机学习算法的关键。
支持向量机是由算法从训练数据中抽取的小的子集构成。
支持向量机的体系结构如图12 -1所示。
图12-1 支持向量机的体系结构其中K为核函数,其种类主要有:线性核函数:K(x,x i)=x T x i;多项式核函数:K(x,x i)=(γx T x i+r)p,γ>0;径向基核函数:K(x,x i )=exp(-γ∥x −x i ∥2), γ>0;两层感知器核函数:K(x,x i )=tanh(γx T x i+r )。
1.二分类支持向量机C - SVC 模型是比较常见的二分类支持向量机模型,其具体形式如下:1)设已知训练集:T ={(x 1,y 1),…,(x i ,y i )}∈(X ×Y )ι其中,x i ∈X =R n ,y i ∈Y ={1,-1}( i =1,2,…,ι);x i 为特征向量。
matlab基本代码解读
matlab基本代码解读Matlab(Matrix Laboratory)是一种高级的数学计算软件,广泛用于科学、工程、数据分析和其他领域。
下面是一些基本的Matlab代码片段,并提供了简要的解读:### 示例1:矩阵运算```matlabA = [1 2; 3 4];B = [5 6; 7 8];C = A * B;disp(C);```**解读:**- 创建了两个2x2的矩阵A和B。
- 通过矩阵乘法计算得到矩阵C。
- 使用`disp`函数显示结果。
### 示例2:绘制图形```matlabx = linspace(0, 2*pi, 100);y = sin(x);plot(x, y);```**解读:**- 通过`linspace`生成从0到2π的100个等间隔点作为x。
- 计算这些点的sin值作为y。
- 使用`plot`函数绘制x和y之间的关系图。
### 示例3:条件语句```matlabnum = 15;if num > 10disp('Number is greater than 10');elsedisp('Number is not greater than 10');end```**解读:**- 设置变量num的值为15。
- 使用`if`语句检查num是否大于10。
- 如果条件成立,显示'Number is greater than 10',否则显示'Number is not greater than 10'。
### 示例4:循环语句```matlabfor i = 1:5disp(['Iteration ', num2str(i)]);end```**解读:**- 使用`for`循环从1迭代到5。
- 在每次迭代中,使用`disp`显示当前迭代的信息。
这些是Matlab中一些基本代码片段的简单解读。
傻瓜攻略(十九)——MATLAB实现SVM多分类
傻瓜攻略(十九)——MATLAB实现SVM多分类SVM (Support Vector Machine) 是一种常用的机器学习算法,广泛应用于分类问题。
原始的 SVM 算法只适用于二分类问题,但是有时我们需要解决多分类问题。
本文将介绍如何使用 MATLAB 实现 SVM 多分类。
首先,我们需要明确一些基本概念。
在 SVM 中,我们需要对每个类别建立一个分类器,然后将未知样本进行分类。
这涉及到两个主要步骤:一对一(One-vs-One)分类和一对其他(One-vs-Rest)分类。
在一对一分类中,我们需要对每两个类别都建立一个分类器。
例如,如果有三个类别 A、B 和 C,那么我们需要建立三个分类器:A vs B, A vs C 和 B vs C。
然后,我们将未知样本进行分类,看它属于哪个类别。
在一对其他分类中,我们将一个类别看作是“正例”,而其他所有类别看作是“负例”。
例如,如果有三个类别 A、B 和 C,那么我们需要建立三个分类器:A vs rest, B vs rest 和 C vs rest。
然后,我们将未知样本进行分类,看它属于哪个类别。
接下来,我们将使用一个示例数据集来演示如何使用MATLAB实现SVM多分类。
我们将使用鸢尾花数据集,该数据集包含了三个类别的鸢尾花样本。
首先,我们需要加载数据集。
在 MATLAB 中,我们可以使用`load`函数加载内置的鸢尾花数据集。
代码如下所示:```load fisheriris```数据集加载完成后,我们可以查看数据集的结构。
在 MATLAB 中,我们可以使用`whos`函数查看当前工作空间中的变量。
代码如下所示:```whos``````X = meas;Y = species;```然后,我们可以使用`fitcecoc`函数构建一个多分类 SVM 模型。
`fitcecoc`函数可以自动选择最佳的核函数,并训练多个二分类器来实现多分类。
代码如下所示:```SVMModel = fitcecoc(X, Y);```训练完成后,我们可以使用`predict`函数对未知样本进行分类。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
支持向量机m a t l a b 实现源代码edit svmtrain>>edit svmclassify>>edit svmpredictfunction [svm_struct, svIndex] = svmtrain(training, groupnames, varargin)%SVMTRAIN trains a support vector machine classifier%% SVMStruct = SVMTRAIN(TRAINING,GROUP) trains a support vector machine % classifier using data TRAINING taken from two groups given by GROUP.% SVMStruct contains information about the trained classifier that is% used by SVMCLASSIFY for classification. GROUP is a column vector of% values of the same length as TRAINING that defines two groups. Each% element of GROUP specifies the group the corresponding row of TRAINING % belongs to. GROUP can be a numeric vector, a string array, or a cell% array of strings. SVMTRAIN treats NaNs or empty strings in GROUP as% missing values and ignores the corresponding rows of TRAINING.%% SVMTRAIN(...,'KERNEL_FUNCTION',KFUN) allows you to specify the kernel % function KFUN used to map the training data into kernel space. The% default kernel function is the dot product. KFUN can be one of the% following strings or a function handle:%% 'linear' Linear kernel or dot product% 'quadratic' Quadratic kernel% 'polynomial' Polynomial kernel (default order 3)% 'rbf' Gaussian Radial Basis Function kernel% 'mlp' Multilayer Perceptron kernel (default scale 1)% function A kernel function specified using @,% for example @KFUN, or an anonymous function%% A kernel function must be of the form%% function K = KFUN(U, V)%% The returned value, K, is a matrix of size M-by-N, where U and V have M% and N rows respectively. If KFUN is parameterized, you can use% anonymous functions to capture the problem-dependent parameters. For% example, suppose that your kernel function is%% function k = kfun(u,v,p1,p2)% k = tanh(p1*(u*v')+p2);%% You can set values for p1 and p2 and then use an anonymous function:% @(u,v) kfun(u,v,p1,p2).%% SVMTRAIN(...,'POLYORDER',ORDER) allows you to specify the order of a% polynomial kernel. The default order is 3.%% SVMTRAIN(...,'MLP_PARAMS',[P1 P2]) allows you to specify the% parameters of the Multilayer Perceptron (mlp) kernel. The mlp kernel% requires two parameters, P1 and P2, where K = tanh(P1*U*V' + P2) and P1% > 0 and P2 < 0. Default values are P1 = 1 and P2 = -1.%% SVMTRAIN(...,'METHOD',METHOD) allows you to specify the method used% to find the separating hyperplane. Options are%% 'QP' Use quadratic programming (requires the Optimization Toolbox)% 'LS' Use least-squares method%% If you have the Optimization Toolbox, then the QP method is the default% method. If not, the only available method is LS.%% SVMTRAIN(...,'QUADPROG_OPTS',OPTIONS) allows you to pass an OPTIONS % structure created using OPTIMSET to the QUADPROG function when using% the 'QP' method. See help optimset for more details.%% SVMTRAIN(...,'SHOWPLOT',true), when used with two-dimensional data,% creates a plot of the grouped data and plots the separating line for% the classifier.%% Example:% % Load the data and select features for classification% load fisheriris% data = [meas(:,1), meas(:,2)];% % Extract the Setosa class% groups = ismember(species,'setosa');% % Randomly select training and test sets% [train, test] = crossvalind('holdOut',groups);% cp = classperf(groups);% % Use a linear support vector machine classifier% svmStruct = svmtrain(data(train,:),groups(train),'showplot',true);% classes = svmclassify(svmStruct,data(test,:),'showplot',true);% % See how well the classifier performed% classperf(cp,classes,test);% cp.CorrectRate%% See also CLASSIFY, KNNCLASSIFY, QUADPROG, SVMCLASSIFY.% Copyright 2004 The MathWorks, Inc.% $Revision: 1.1.12.1 $ $Date: 2004/12/24 20:43:35 $% References:% [1] Kecman, V, Learning and Soft Computing,% MIT Press, Cambridge, MA. 2001.% [2] Suykens, J.A.K., Van Gestel, T., De Brabanter, J., De Moor, B.,% Vandewalle, J., Least Squares Support Vector Machines,% World Scientific, Singapore, 2002.% [3] Scholkopf, B., Smola, A.J., Learning with Kernels,% MIT Press, Cambridge, MA. 2002.%% SVMTRAIN(...,'KFUNARGS',ARGS) allows you to pass additional% arguments to kernel functions.% set defaultsplotflag = false;qp_opts = [];kfunargs = {};setPoly = false; usePoly = false;setMLP = false; useMLP = false;if ~isempty(which('quadprog'))useQuadprog = true;elseuseQuadprog = false;end% set default kernel functionkfun = @linear_kernel;% check inputsif nargin < 2error(nargchk(2,Inf,nargin))endnumoptargs = nargin -2;optargs = varargin;% grp2idx sorts a numeric grouping var ascending, and a string grouping % var by order of first occurrence[g,groupString] = grp2idx(groupnames);% check group is a vector -- though char input is special...if ~isvector(groupnames) && ~ischar(groupnames)error('Bioinfo:svmtrain:GroupNotVector',...'Group must be a vector.');end% make sure that the data is correctly oriented.if size(groupnames,1) == 1groupnames = groupnames';end% make sure data is the right sizen = length(groupnames);if size(training,1) ~= nif size(training,2) == ntraining = training';elseerror('Bioinfo:svmtrain:DataGroupSizeMismatch',...'GROUP and TRAINING must have the same number of rows.')endend% NaNs are treated as unknown classes and are removed from the training% datanans = find(isnan(g));if length(nans) > 0training(nans,:) = [];g(nans) = [];endngroups = length(groupString);if ngroups > 2error('Bioinfo:svmtrain:TooManyGroups',...'SVMTRAIN only supports classification into two groups.\nGROUP contains %d different groups.',ngroups) end% convert to 1, -1.g = 1 - (2* (g-1));% handle optional argumentsif numoptargs >= 1if rem(numoptargs,2)== 1error('Bioinfo:svmtrain:IncorrectNumberOfArguments',...'Incorrect number of arguments to %s.',mfilename);okargs = {'kernel_function','method','showplot','kfunargs','quadprog_opts','polyorder','mlp_params'}; for j=1:2:numoptargspname = optargs{j};pval = optargs{j+1};k = strmatch(lower(pname), okargs);%#okif isempty(k)error('Bioinfo:svmtrain:UnknownParameterName',...'Unknown parameter name: %s.',pname);elseif length(k)>1error('Bioinfo:svmtrain:AmbiguousParameterName',...'Ambiguous parameter name: %s.',pname);elseswitch(k)case 1 % kernel_functionif ischar(pval)okfuns = {'linear','quadratic',...'radial','rbf','polynomial','mlp'};funNum = strmatch(lower(pval), okfuns);%#okif isempty(funNum)funNum = 0;endswitch funNum %maybe make this less strict in the futurecase 1kfun = @linear_kernel;case 2kfun = @quadratic_kernel;case {3,4}kfun = @rbf_kernel;case 5kfun = @poly_kernel;usePoly = true;case 6kfun = @mlp_kernel;useMLP = true;otherwiseerror('Bioinfo:svmtrain:UnknownKernelFunction',...'Unknown Kernel Function %s.',kfun);endelseif isa (pval, 'function_handle')kfun = pval;elseerror('Bioinfo:svmtrain:BadKernelFunction',...'The kernel function input does not appear to be a function handle\nor valid function name.')case 2 % methodif strncmpi(pval,'qp',2)useQuadprog = true;if isempty(which('quadprog'))warning('Bioinfo:svmtrain:NoOptim',...'The Optimization Toolbox is required to use the quadratic programming method.') useQuadprog = false;endelseif strncmpi(pval,'ls',2)useQuadprog = false;elseerror('Bioinfo:svmtrain:UnknownMethod',...'Unknown method option %s. Valid methods are ''QP'' and ''LS''',pval);endcase 3 % displayif pval ~= 0if size(training,2) == 2plotflag = true;elsewarning('Bioinfo:svmtrain:OnlyPlot2D',...'The display option can only plot 2D training data.')endendcase 4 % kfunargsif iscell(pval)kfunargs = pval;elsekfunargs = {pval};endcase 5 % quadprog_optsif isstruct(pval)qp_opts = pval;elseif iscell(pval)qp_opts = optimset(pval{:});elseerror('Bioinfo:svmtrain:BadQuadprogOpts',...'QUADPROG_OPTS must be an opts structure.');endcase 6 % polyorderif ~isscalar(pval) || ~isnumeric(pval)error('Bioinfo:svmtrain:BadPolyOrder',...'POLYORDER must be a scalar value.');endif pval ~=floor(pval) || pval < 1error('Bioinfo:svmtrain:PolyOrderNotInt',...'The order of the polynomial kernel must be a positive integer.')endkfunargs = {pval};setPoly = true;case 7 % mlpparamsif numel(pval)~=2error('Bioinfo:svmtrain:BadMLPParams',...'MLP_PARAMS must be a two element array.');endif ~isscalar(pval(1)) || ~isscalar(pval(2))error('Bioinfo:svmtrain:MLPParamsNotScalar',...'The parameters of the multi-layer perceptron kernel must be scalar.'); endkfunargs = {pval(1),pval(2)};setMLP = true;endendendendif setPoly && ~usePolywarning('Bioinfo:svmtrain:PolyOrderNotPolyKernel',...'You specified a polynomial order but not a polynomial kernel');endif setMLP && ~useMLPwarning('Bioinfo:svmtrain:MLPParamNotMLPKernel',...'You specified MLP parameters but not an MLP kernel');end% plot the data if requestedif plotflag[hAxis,hLines] = svmplotdata(training,g);legend(hLines,cellstr(groupString));end% calculate kernel functiontrykx = feval(kfun,training,training,kfunargs{:});% ensure function is symmetrickx = (kx+kx')/2;catcherror('Bioinfo:svmtrain:UnknownKernelFunction',...'Error calculating the kernel function:\n%s\n', lasterr);end% create Hessian% add small constant eye to force stabilityH =((g*g').*kx) + sqrt(eps(class(training)))*eye(n);if useQuadprog% The large scale solver cannot handle this type of problem, so turn it% off.qp_opts = optimset(qp_opts,'LargeScale','Off');% X=QUADPROG(H,f,A,b,Aeq,beq,LB,UB,X0,opts)alpha = quadprog(H,-ones(n,1),[],[],...g',0,zeros(n,1),inf *ones(n,1),zeros(n,1),qp_opts);% The support vectors are the non-zeros of alphasvIndex = find(alpha > sqrt(eps));sv = training(svIndex,:);% calculate the parameters of the separating line from the support% vectors.alphaHat = g(svIndex).*alpha(svIndex);% Calculate the bias by applying the indicator function to the support% vector with largest alpha.[maxAlpha,maxPos] = max(alpha); %#okbias = g(maxPos) - sum(alphaHat.*kx(svIndex,maxPos));% an alternative method is to average the values over all support vectors % bias = mean(g(sv)' - sum(alphaHat(:,ones(1,numSVs)).*kx(sv,sv)));% An alternative way to calculate support vectors is to look for zeros of % the Lagrangians (fifth output from QUADPROG).%% [alpha,fval,output,exitflag,t] = quadprog(H,-ones(n,1),[],[],...% g',0,zeros(n,1),inf *ones(n,1),zeros(n,1),opts);%% sv = t.lower < sqrt(eps) & t.upper < sqrt(eps);else % Least-Squares% now build up compound matrix for solverA = [0 g';g,H];b = [0;ones(size(g))];x = A\b;% calculate the parameters of the separating line from the support % vectors.sv = training;bias = x(1);alphaHat = g.*x(2:end);endsvm_struct.SupportVectors = sv;svm_struct.Alpha = alphaHat;svm_struct.Bias = bias;svm_struct.KernelFunction = kfun;svm_struct.KernelFunctionArgs = kfunargs;svm_struct.GroupNames = groupnames;svm_struct.FigureHandles = [];if plotflaghSV = svmplotsvs(hAxis,svm_struct);svm_struct.FigureHandles = {hAxis,hLines,hSV};end。
