RNAseq(转录组)数据提交 NCBI 快速入门指南
NCBI数据上传指南说明书

NCBI数据上传指南近年来,伴随着高通量测序的广泛应用,海量的测序数据也随之产生。
高通量测序数据分析完成发表文章时,科研工作者需要将数据提交到一个公共平台(例如NCBI数据库)。
下文中我们汇总了如何向NCBI平台提交数据,以及不同测序项目需要提交哪些数据,希望为您发表高分论文提供一些帮助。
一、NCBI数据库及数据类型向NCBI数据库提交数据可参考Submission Portal网页中所列数据库与工具,按照网站提示及说明进行操作,可以在如下输入框中输入关键词查看相关信息。
1. NCBI常用数据库介绍1)GenBank网址:/genbank/GenBank是美国国立卫生研究院(NIH)基因序列数据库,包含所有公开的DNA序列和注释信息。
GenBank数据库也是世界上最大的、最重要的、最有影响力的生物全领域数据库,其数据正被全球数以百万计的研究人员获取与引用。
2)SRA网址:/sra/存储测序平台产生的测序数据。
包括Roche 454 GS System®, Illumina Genome Analyzer®, Applied BiosystemsSOLiD® System, HelicosHeliscope®, Complete Genomics®, and Pacific Biosciences SMRT®。
3)TSA ( Transcriptome Shotgun Assembly )存储由第二代测序数据组装拼接得到的转录本序列。
2. 数据类型上传到NCBI 的数据,依据数据类型,大体可以分为测序原始数据和分析数据。
原始数据(Raw data)指未经任何处理的测序下机文件,包含二代及三代测序数据等。
其中二代测序中最常见的是illumina测序仪产生的fastq文件,例如锐博在项目结题时为您提供的*fastq.gz文件。
这一类型的文件需要提交到NCBI的SRA数据库,具体上传方法我们将在下文中做详细介绍。
GEO数据库上传--RNA-seq发文必备技能

GEO数据库上传--RNA-seq发文必备技能GEO数据库是NCBI开发的基因表达数据库,主要接收通过高通量测序、基因芯片等方法获得的基因表达数据。
除SRA数据库之外,GEO数据库也是目前文章投递过程中要求进行数据上传的数据库之一。
网上关于GEO数据库的上传教程良莠不齐,推荐使用下面的方法快速上传自己的数据到GEO。
第一步向GEO提交联系信息正常登录NCBI,如果是首次进入GEO,页面会出现两个选项,如下图:对于之前从未上传数据到GEO的同学,请选择第二个选项。
按照要求填写相应的信息,包括注册人员信息和上传者信息,其中最重要的是邮箱信息。
填写完毕,管理员会发送通知和链接到你的邮箱。
信息填写完毕后会自动登陆GEO。
第二步创建新提交任务再次登录进入MY submissions选择new submission,自动跳转进入Subimitting data 界面(下面以高通量测序数据为例进行讲解)选择 High-throughput sequence submissions(如下两图)。
第三步文件准备完成上述操作后,页面自动跳转到高通量测序数据上传界面。
按照提示准备好三种文件。
第一种文件 Metadata spreadsheet,主要记录RNA-seq实验的目的、方法、处理过程,以及统计预备上传的所有文件的信息和文件的MD5值等等。
网站提供了相应的案例模版(同一页面具有下载链接),此步骤一定要基于模版,根据自己实验的实际情况填写所有内容,不可以直接拷贝。
模版如下:第二种文件 Processed data files,主要是基因表达的数据文件,一般转换成文本格式,可以是多个文件(例如一个样品对应一个文件)也可以是一个文件(多样品数据整合),或者其他的补充文件,包括gff文件等等。
这一部分的文件是基于测序的原始数据经处理之后的结果。
如果涉及新注释的一些基因的表达数据,则需要提供注释基因的位置信息等(如下图)。
ncbi使用指导

ncbi使用指导导言:NCBI(National Center for Biotechnology Information)是美国国家生物技术信息中心,是全球最大的生物信息学数据库。
NCBI提供了丰富的生物学资源,包括基因序列、蛋白质序列、科学文献等。
本文将为您介绍如何使用NCBI来获取和利用生物学信息。
一、注册NCBI账号在使用NCBI之前,首先需要注册一个账号。
在NCBI官方网站()的主页上,点击右上角的“Sign In”按钮,并选择“Register for an NCBI account”。
根据提示提供相关信息,完成注册流程。
二、搜索和获取基因序列1. 打开NCBI网站后,点击主页上的“Search”按钮,进入搜索页面。
2. 在搜索框中输入您感兴趣的基因名称或者序列标识符,如“BRCA1”。
3. 点击“Search”按钮进行搜索。
4. 在搜索结果中,选择您需要的基因序列,点击链接进入该基因的详细信息页面。
5. 在详细信息页面中,您可以获取该基因的序列信息,可以下载或拷贝该序列以供后续分析使用。
三、检索科学文献1. 在NCBI主页上方的搜索框中,选择“PubMed”为搜索目标。
2. 输入您感兴趣的科学文献关键词,如“cancer treatment”。
3. 点击搜索按钮进行检索。
4. 在搜索结果中,选择您需要的文献,点击链接进入该文献的详细页面。
5. 在详细页面中,您可以阅读文章摘要,并根据需要下载全文或进行其他操作。
四、利用NCBI工具进行序列分析1. 在NCBI主页上方的菜单栏中,选择“Tools”进行工具选择。
2. 根据您的需求选择相应的工具,如“BLAST”进行序列比对分析。
3. 进入选择的工具页面后,按照提示上传或输入您的序列数据。
4. 设定参数并运行分析。
5. 分析完成后,您可以查看比对结果、序列保守性等信息,并进行进一步的分析和解释。
五、订阅NCBI数据库更新信息1. 在NCBI主页上方菜单栏中选择“NCBI Account”。
RNAseq(转录组)数据提交 NCBI 快速入门指南

RNAseq数据提交NCBI快速入门指南官网:/一上传前准备工作1申请NCBI账号:点击申请账号登陆已申请的账号点击申请账号用户名密码邮箱验证码点击注册注意:新申请的账号需要登录邮箱验证后才能提交2准备需要上传的原始数据:注意事项:(1)所有样本的原始数据需放在同一目录下(2)原始数据为fastq格式的压缩文件。
测序公司提供(3)若为双端测序,需要提供双端的原始数据,即R1端和R2端二提交SRA的主要步骤1进入提交界面:点击选择SRA数据库点击进入开始提交2填写提交者信息:*号为必填项名姓邮箱primary邮箱是申请账号的邮箱,系统自动匹配,可更改,secondary邮箱为可选项学院学校邮编和国家学校所在街道和城市3填写释放日期信息:是否创建新BioProject,选择Yes是否创建新BioSsamples,选择Yes数据立即释放数据释放日期可自定义,最长可延迟4年释放4填写Project信息:老师研究项目的标题老师研究项目的简短描述是否提交过数据到NCBI,有还是没有都选择No 该界面中此模块往下都无需填写5选择BioSample类型:红色方框内的类型是转录组样品常用的样品类型样本为细菌,真菌类型样本为动物类型样本为无脊椎动物类型样本为人6填写BioSample属性表:样本为植物类型样本为病毒类型下载TSV格式,填好后点击“选择文件”上传表格,文件只支持tsv和txt格式上传。
此网址详细介绍每一列的信息如何填写动物类型属性表(anism.animal.1.0.tsv )植物类型属性表(Plant.1.0.tsv)绿色为必填项蓝色至少选择一项黄色可不填微生物类型属性表(Microbe.1.0.tsv)7填写SRA Metadata信息:下载文件,填好后点击“选择文件”上传表格,此文件为excel格式。
注:所有黄色区域是需要下拉选择的,不需打字,其余区域需要手动填写8开始上传原始数据:网页版网页版传输入口双端数据都需要上传Linux版高速传输ascp入口,命令行支持doc和linux使用说明9提交前预览:检查释放日期,提交后不能更改检查67步上传的文件是否还需要更改检查样本个数以及双端是否正确提交,等待审核,大约1-2个工作日即可完成10查看投稿所用SRA号:等待提交开始审核审核完成点击投稿所需SRA号。
NCBI使用教程

NCBI使用教程NCBI(National Center for Biotechnology Information)是一个提供生物信息学相关资源和服务的综合性数据库,为研究者和学生们提供了大量的生物学数据、文献和工具,对于研究生物学和相关领域的人来说是非常有价值的资源。
本文将向您介绍如何使用NCBI进行生物信息学的研究和学习。
在DNA/RNA seq页面,可以和浏览生物序列数据。
可以输入序列数据,通过BLAST程序进行序列比对和比对分析。
可以利用高级功能,如限定序列长度、物种、数据库等。
此外,在这个页面上,还可以进行FASTA格式序列的格式化处理,并获得一些特定的DNA/RNA序列数据。
在Gene页面,可以和浏览基因信息。
可以通过基因名、ID等关键字进行。
每个基因都有自己的页面,显示了其基本信息、结构、功能以及相关文献。
在页面底部还可以找到该基因的序列信息、同源基因和调控因子等信息。
在Protein页面,可以和浏览蛋白质信息。
可以输入蛋白质名、ID等关键字进行。
每个蛋白质也有自己的页面,显示了其基本信息、结构、功能等。
在页面底部还可以找到该蛋白质的序列信息、同源蛋白和结构域等信息。
在Nucleotide页面,可以和浏览核苷酸信息。
可以输入核苷酸序列、基因名等关键字进行。
每个核苷酸也有自己的页面,显示了其基本信息、序列、功能等。
在页面底部还可以找到该核苷酸的同源序列和CDS (Coding Sequence)等信息。
在NCBI的Tools页面,提供了许多有用的工具和资源。
如BLAST、序列比对工具、基因注释工具等。
可以根据自己的需要选择相应的工具来进行生物信息学分析和研究。
此外,NCBI还提供了一些教育和培训资源,如教程、视频和在线培训课程,可以帮助用户更好地使用NCBI的数据库和工具。
综上所述,NCBI是一个非常重要和有价值的生物信息学资源和工具,可以帮助生物学和相关领域的研究者和学生进行科研和学习。
NCBI数据库使用方法快速入门!

NCBI数据库使用方法快速入门!
Taxonomy界面,会显示该物种的Nucleotide和Protein,选择“Protein
Protein界面,点击“RefSeq”(该数据库包括具有生物意义上的非冗余基因、转录本和蛋白序列,使用人类基因命名委员会定义的术语,并且包括了官方的基因符号和可选的符号):
二、蛋白信息查询
、将该文件导入搜库软件,可对蛋白进行定性分析,继而得到差异蛋白。
在NCBI Protein”,并输入差异蛋白的Accession Number,点击“Search”:
Protein界面,就会显示该蛋白的信息(如氨基酸个数、蛋白名称、蛋白序列和序列等):
一般情况下,如果蛋白质组所研究的物种已经被测序,推荐使用Uniprot数据库作为搜库的数据库,如果所研究的物种在Uniprot数据库中蛋白数据较少,推荐使用NCBI数据库进行搜库。
好了,今天的介绍就到这里,感兴趣的小伙伴们收藏起来吧~希望对大家有所帮助!。
RNA-seq数据上传GEO

RNA-seq原始数据上传GEO数据库需要软件:7Z压缩软件和FileZilla数据上传软件,下载地址如下链接:/s/1kUPlv9P密码:xaqv1.首先要注册一个NCBI账号,网址https:///account/?back_url=https%3A%2F%2Fwww.ncbi.nl %2F2.然后进入GEO主页,网址:https:///gds3.点击Submit to GEO注册GEO账号,然后点击High-throughput sequence submissions,接下来点击Download metadata spreadsheet (template and examples)下载Metadata spreadsheet表(这个表就是填信息用的,里面有模板,照着填就行了)4.填好信息后就是上传数据了,主要包括3种文件,上述提到的Metadataspreadsheet,RNA-seq 原始数据,以及基因表达量文件(FPKM/RPKM值文件)将所有信息整理好放入一个文件夹中,文件夹以自己的账号名命名。
5.用7Z软件将上述材料打包压缩,先将该文件夹打包成.tar格式,再将.tar格式压缩成.gz格式。
(可以参考/article/4853e1e5725d651909f726d8.html)6.数据上传安装好FileZilla后先设置一下参数,便于文件传输,编辑---设置---连接设置好后就是数据上传了文件---站点管理器,主机设置为:ftp://geo:33%259uyj_fCh%3FM16H@/fasp/用户:geo密码:33%9uyj_fCh?M16H这些在GEO网站上都有点击连接,在状态窗口中可以看出是否连接成功, 如下图A区在B区找到你的.gz压缩文件,在C区找到fasp文件夹,双击进去,最后将你的压缩文件拖到C区的fasp文件夹中。
传输很慢,我9G文件传了14h.文件传好后给GEO发封邮件,************邮件内容主要说你上传了文件,让他查看,如下:Dear Sir,We would like to submit our RNA-seq data to GEO web site. We have upload our data (***.tar.gz) to ftp:///fasp/. The compress file including:RNA-seq raw data files in fastq format:11h-1-1.fq11h-2-1.fq31h-1-1.fq31h-2-1.fq31h-1-1.fq31h-2-1.fq11h-1-2.fq11h-2-2.fq31h-1-2.fq31h-2-2.fq31h-1-2.fq31h-2-2.fqGene expression files in excel format:11h-1.xls11h-2.xls31h-1.xls31h-2.xls31h-1.xls31h-2.xlsWe want to release our data as soon as possible.Thank youBest wishes!your's name。
最全的向NCBI递交序列说明
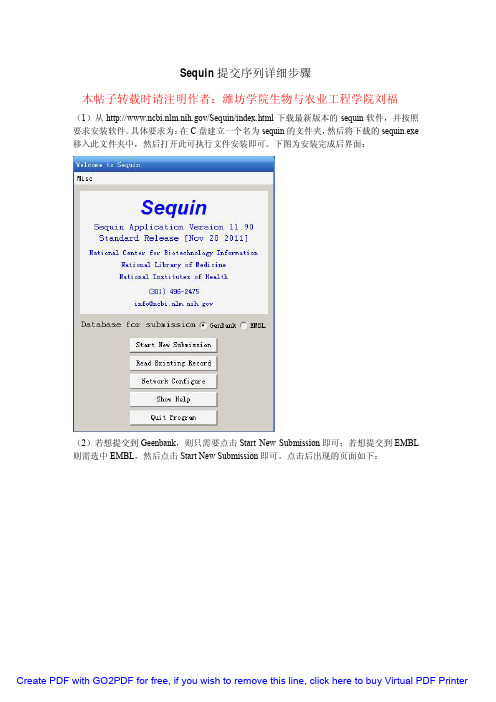
Sequin提交序列详细步骤本帖子转载时请注明作者:潍坊学院生物与农业工程学院刘福(1)从/Sequin/index.html下载最新版本的sequin软件,并按照要求安装软件。
具体要求为:在C盘建立一个名为sequin的文件夹,然后将下载的sequin.exe 移入此文件夹中,然后打开此可执行文件安装即可。
下图为安装完成后界面:(2)若想提交到Geenbank,则只需要点击Start New Submission即可;若想提交到EMBL 则需选中EMBL,然后点击Start New Submission即可。
点击后出现的页面如下:(3)第一个标签Submission:什么时候发表你的序列?可以选择①Immediately After Processing即Geenbank工作人员处理完毕你的序列后就将其发表到Geenbank内。
②Release Date即选择让Geenbank工作人员发表到Geenbank内的时间。
Tentative title for manuscript 这一项一般填写你将发表论文的暂定题目或已发表论文的题目。
(4)完成后点击Next Page,出现以下界面:此处填发见图内,需要解释的是Sfx是别命的意思,中国人一般不需要填写;M.I为中间名,中国人也不需要填;邮箱地址是负责最终提交序列者的,不一定是序列的作者的,如你导师是作者,你是负责给他提交者,则这里就填写你的邮箱,其他信息填写你导师的。
注:0086为中国国际代号,必须加上。
(5)完成后,点击Next Page,出现以下界面:为填写序列作者的界面,前一步填写的联系人被自动列为第一作者,然后往下继续填写其他作者,此处只显示三行,当你填写完第三个后,通过拖动滚动条后面将会还出现填写的表格,直到将所有作者填写完毕。
另外Consortium 为参与序列获取的机构名,当以此机构名义发表序列时可以填写,上面的作者也一并填写。
(6)完成后,点击Next Page。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
RNAseq数据提交NCBI快速入门指南
官网:/
一上传前准备工作
1申请NCBI账号:
点击申请账号
登陆已申请的账号点击申请账号
用户名密码
邮箱
验证码
点击注册注意:新申请的账号需要登录邮箱验证后才能提交
2准备需要上传的原始数据:
注意事项:
(1)所有样本的原始数据需放在同一目录下
(2)原始数据为fastq格式的压缩文件。
测序公司提供
(3)若为双端测序,需要提供双端的原始数据,即R1端和R2端
二提交SRA的主要步骤
1进入提交界面:
点击
选择SRA数据库
点击进入
开始提交
2填写提交者信息:
*号为必填项名姓
邮箱primary邮箱是申请账号的邮箱,系统自动匹配,可更改,secondary邮箱为可选项
学院
学校
邮编和国家
学校所在街道和城市
3填写释放日期信息:
是否创建新BioProject,选择Yes
是否创建新BioSsamples,选择Yes
数据立即释放
数据释放日期可自定义,最长可延迟4年释放
4填写Project信息:
老师研究项目的标题
老师研究项目的简短描述
是否提交过数据到NCBI,有还是没有都选择No 该界面中此模块往下都无需填写
5选择BioSample类型:
红色方框内的类型是转录组样品常用的样品类型
样本为细菌,真菌类型
样本为动物类型
样本为无脊椎动物类型
样本为人
6填写BioSample属性表:
样本为植物类型
样本为病毒类型
下载TSV格式,填好后点击“选择文件”上传表格,文件只支持tsv和txt格式上传。
此网址详细介绍每一列的信息如何填写
动物类型属性表(anism.animal.1.0.tsv )
植物类型属性表(Plant.1.0.tsv
)绿色为必填项蓝色至少选择一项
黄色可不填
微生物类型属性表(Microbe.1.0.tsv)
7填写SRA Metadata信息:
下载文件,填好后点击“选择文件”上传表格,此文件为excel格式。
注:所有黄色区域是需要下拉选择的,不需打字,其余区域需要手动填写
8开始上传原始数据:
网页版
网页版传输入口
双端数据都需要上传
Linux版
高速传输ascp入口,命令行支持doc和linux
使用说明
9提交前预览:
检查释放日期,提交后不能更改
检查67步上传的文件是否还需要更改
检查样本个数以及双端是否正确
提交,等待审核,大约1-2个工作日即可完成
10查看投稿所用SRA号:
等待提交
开始审核
审核完成
点击
投稿所需SRA号。
